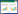文章信息
- 史延华, 任磊, 贾阳, Ruth Nahurira, 闫艳春
- SHI Yan-hua, REN Lei, JIA Yang, RUTH Nahurira, YAN Yan-chun
- 施氏假单胞菌YC-YH1对甲基对硫磷的降解及其代谢产物检测
- Biodegradation of Methyl Parathion by Pseudomonas stutzeri YC-YH1 and Detection of Its Metabolites
- 农业环境科学学报, 2015, 34(11): 2097-2104
- Journal of Agro-Environment Science, 2015, 34(11): 2097-2104
- http://dx.doi.org/10.11654/jaes.2015.11.009
-
文章历史
- 收稿日期: 2015-05-05
甲基对硫磷,化学名称为O,O-二甲基-O-(4-硝基苯基)硫代磷酸酯,属高毒有机磷农药,因其半衰期长、毒性大而广受关注,在环境和粮食中均有检出[1, 2, 3]。甲基对硫磷对哺乳动物的危害主要为神经性毒剂,通过抑制乙酰胆碱酯酶活性导致乙酰胆碱积累,从而引起神经功能紊乱,严重时可致死,对生态环境与人体健康具有较大威胁[4]。
环境中有机污染物的传统处理方法主要包括化学法或焚烧、掩埋等,这些方法的副作用较大,容易造成第二次污染,因此有机磷农药污染在环境中最有效的清除主要是以生物降解为主[5]。生物降解具有降解彻底、无二次污染、速效性和广谱性等优点。细菌等原核生物代谢农药通常是同化吸收,将其作为基本的营养物质和能源,因此微生物在环境污染物的降解过程中占据着主导地位[6]。
对于农药的微生物降解,已经分离获得大量的降解菌株,可降解污染物包括杀虫剂、杀菌剂和除草剂等,微生物类型主要为细菌与真菌[7, 8, 9]。本研究以实验室保藏的有机磷农药降解菌株YC-YH1为研究对象,对菌株进行鉴定,测定其对甲基对硫磷的降解效率并克隆降解相关基因。同时,建立基于HPLC对甲基对硫磷代谢产物对硝基苯酚的检测方法并检测降解产物对YC-YH1生长的影响。
1 材料与方法 1.1 药品、仪器和培养基本研究所用的甲基对硫磷(纯度97.2%)与对硝基苯酚(纯度99.1%)均购自国药集团。甲醇和乙腈为色谱纯级(美国,Fisher公司),其余药品为分析纯级,均购自国药集团。
所用仪器有Agilent 1200高效液相色谱仪(美国,Agilent公司),Biolog微生物鉴定系统(美国,Biolog公司),Bio-Rad C1000 PCR仪(美国,Bio-Rad公司),BX63显微镜(日本,Olympus公司),S-3000N扫描电镜(日本,Hitachi公司),ScanSpeed 1580R冷冻高速离心机(丹麦,Labogene公司),Agilent 1260 HPLC- Agilent 6420 三重四极杆液质联用仪(美国,Agilent 公司),Supcre G6R全自动菌落分析仪(中国,迅数)。
普通培养基(LB)[10]:胰蛋白胨10 g,酵母粉5 g,NaCl 10 g,去离子水1 L,pH=7.2±0.2。
无机盐离子培养基(MSM)[11]:NH4NO3 1.0 g,NaCl 0.5 g,(NH4)2SO4 0.5 g,KH2PO4 0.5 g,K2HPO4 1.5 g,去离子水1 L,pH=7.2±0.2。
上述培养基中加入1.5%的琼脂粉即得到对应的固体培养基,所有培养基均在121 ℃灭菌30 min备用。
1.2 菌株来源菌株为中国农业科学院研究生院生物学教研室分离保藏的有机磷农药降解菌YC-YH1,菌株分离自常年生产有机磷农药的邢台农药厂排污口活性污泥。菌种保藏于中国普通微生物菌种保藏管理中心,保藏号为CGMCC No. 9624。
1.3 菌株的鉴定将YC-YH1菌株在LB固体平板上划线,30 ℃条件下培养过夜,观察平板上生长的菌落形态。挑取单菌落放到载玻片上,固定后进行革兰氏染色,显微镜观察菌株的菌体形态。对菌落进行制片,进行电镜扫描观察。
YC-YH1菌株的生理生化特性鉴定使用博检革兰氏阴性细菌鉴定系统进行(青岛海博生物技术有限公司)[12],同时使用Biolog微生物鉴定系统和GenⅢ微孔板测试菌株YC-YH1利用不同化合物的能力[13]。该微孔板上含有94个不同的表型测试,可将该微生物鉴定到种的水平上。
采用16S rRNA基因序列分析对菌株YC-YH1进行鉴定,主要步骤:①细菌基因组DNA提取(Takara,细菌基因组小提试剂盒)。②以获得的基因组为模板进行16S rRNA序列PCR扩增(引物序列见表 1)。PCR反应条件为:a.95 ℃ 5 min;b.95 ℃ 0.5 min;c.50 ℃ 0.5 min;d.72 ℃ 1.5 min(重复b~d循环34次);e.72 ℃ 10 min。③PCR产物纯化并进行测序(上海生工生物工程股份有限公司)。测序结果进行BLAST比对分析(http://blast.ncbi.nlm.nih.gov/Blast.cgi)。
将菌株YC-YH1在LB液体培养基中培养至对数期,取1 mL菌液,6000 r·min-1离心2 min收集菌体,用新鲜的MSM培养基冲洗并悬浮至OD600nm=1.0。按1%接种量接入10 mL含有100 mg·L-1甲基对硫磷的MSM无机盐培养基中作为处理组,以相同条件下不接菌的含甲基对硫磷的MSM培养基作为对照组,在180 r·min-1、30 ℃的摇床中培养。每12 h取样一次,检测培养液中甲基对硫磷浓度。
以正己烷作为萃取剂提取培养液中的甲基对硫磷,预实验表明该方法的回收率为98.7%~101.2%,证实该方法可行。向培养液中加入等体积的正己烷,颠倒混匀5 min,超声波振荡10 min,4 ℃条件下静置1 h。取上层有机相,经无水硫酸钠脱水后真空冷冻抽干,重溶于等体积甲醇中,经孔径为0.22 μm的滤膜过滤,用Agilent 1200高效液相色谱仪进行检测。色谱柱为Zorbax Eclipse Plus C18(4.6 mm×150 mm×5 μm),流动相为甲醇∶乙腈=80∶20,流速为1 mL·min-1,柱温30 ℃,进样量2 μL,检测波长288 nm,同时以甲基对硫磷标准品建立标准曲线(R2=0.993 1)。利用SPSS 15.0 软件分析实验数据,并绘制相关标准曲线和降解曲线。
1.5 甲基对硫磷降解相关基因检测根据已有报道[14, 15, 16],分别对有机磷水解酶基因mpd、ophC2和opdA设计引物以进行PCR扩增,引物序列见表 1。PCR反应条件为:a.95 ℃ 5 min;b.95 ℃ 0.5 min;c.(mpd为53 ℃,ophC2为55 ℃,opdA为60 ℃)0.5 min;d.72 ℃ 1 min(重复b~d循环34次);e.72 ℃ 10 min。将获得的PCR产物进行琼脂糖凝胶电泳检测,产物纯化后连接载体转化入大肠杆菌DH5α,挑选阳性克隆子进行测序,进行BLAST比对分析并将序列递交至GenBank(http://www.ncbi.nlm.nih.gov/)。
1.6 基于HPLC-MS的甲基对硫磷及其代谢产物检测取在含100 mg·L-1甲基对硫磷的MSM培养基中培养48 h的培养液20 mL,10 000 r·min-1离心5 min,收集上清液并用0.22 μm滤膜过滤。利用HPLC检测样品中的对硝基苯酚,流动相为甲醇∶乙腈∶水=48∶42∶10,流速为1 mL·min-1,柱温30 ℃,进样量2 μL,检测波长320 nm。同时以对硝基苯酚标准品建立标准曲线(R2=0.987 2)。
利用HPLC-MS检测菌株YC-YH1降解甲基对硫磷的代谢产物,电喷雾电离(ESI源),负离子模式扫描。流动相100%甲醇,流速0.2 mL·min-1,1 μL样品直接注射至质谱中。质谱检测条件:毛细管电压和碎裂电压分别为3500 V和150 V,源温度和去溶剂化温度分别为150 ℃和350 ℃。扫描离子范围50~200 Da。用Agilent MassHunter软件收集并分析数据。
1.7 甲基对硫磷代谢产物对菌株YC-YH1生长的影响通过底物抑菌实验分析甲基对硫磷代谢产物对YC-YH1生长的影响。分别制备普通LB固体平板、含甲基对硫磷(100 mg·L-1)的LB固体平板和含对硝基苯酚(100 mg·L-1)的LB固体平板。用无菌的直径为0.8 cm的滤纸片蘸取在LB培养基中培养至对数期的YC-YH1菌液,将滤纸片置于制备的固体LB平板中央。在30 ℃、避光条件下培养,定时观察并用迅数Supcre G6R全自动菌落分析仪拍照记录。
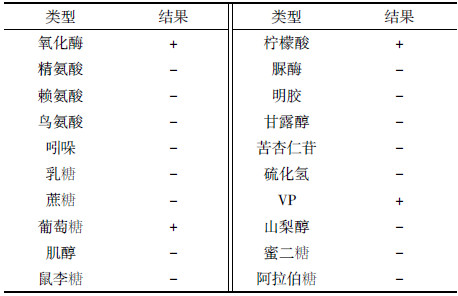
2 结果与讨论 2.1 菌株YC-YH1的鉴定经革兰氏染色、显微镜观察和电镜扫描观察,菌株YC-YH1的特征如图 1所示:菌株为革兰氏阴性菌,菌落为淡黄色,干燥,扁平,边缘不整,不透明,表面粗糙,有褶皱;电镜扫描观察发现菌体为杆状,菌体细长,顶部生有单根鞭毛,长度为3~4 μm。
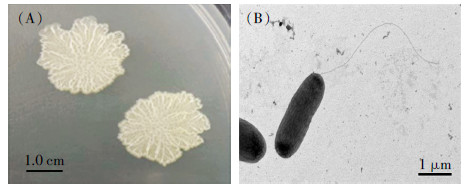
|
| 图 1 菌株YC-YH1的菌落(A)及电镜扫描(B)观察 Figure 1 Colonic morphology (A) and SEM image (B) of strain YC-YH1 |
根据博检革兰氏阴性细菌鉴定系统说明书中提供的方法对培养结果进行判读,得到菌株对各种生化反应的结果,如表 2所示。
Biolog分析菌株YC-YH1的代谢指纹并与数据库进行比对。鉴定结果表明,菌株YC-YH1最具可能性为Pseudomonas stutzeri,其PROB值为0.827、SIM值为0.540、DIST值为5.029,说明鉴定结果较为可信[13]。
将菌株的16S rRNA基因序列提交到GenBank数据库,登录号为KJ786450。经BLAST比对,构建系统发育树(图 2),表明该菌株的16S rRNA基因序列与菌株Pseudomonas stutzeri ATCC 17588的相似度高达98%。结合上述结果,将菌株YC-YH1鉴定为施氏假单胞菌(Pseudomonas stutzeri)[12]。
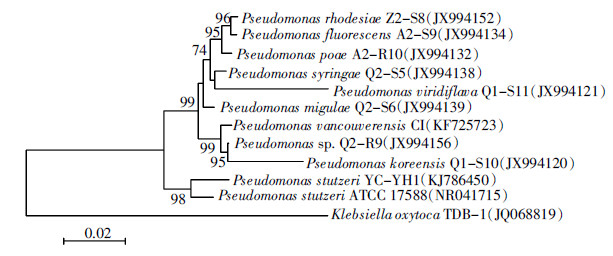
|
| 图 2 菌株YC-YH1 与相关种属菌株的系统发育树 Figure 2 Phylogenetic tree of strain YC-YH1 and other bacteria within a genera |
菌株YC-YH1在72 h内对甲基对硫磷的降解情况如图 3所示。结果表明:100 mg·L-1甲基对硫磷在60 h内被彻底降解,降解率为100%,而对照组中甲基对硫磷的浓度为99.3 mg·L-1。同时,菌株YC-YH1接种到含甲基对硫磷的无机盐培养基中后,因与原LB生长环境差别较大,菌体内需要产生许多适应新环境的变化,如降解基因的表达和降解酶合成等,故在0~12 h之间存在一个短暂的适应期,在12 h以后进入快速降解阶段。可见,菌株YC-YH1能够高效的降解甲基对硫磷,其对甲基对硫磷的降解效率高于Bacillus cereus HY-1[17]和Candida blankii JMUPMD-1[8]。
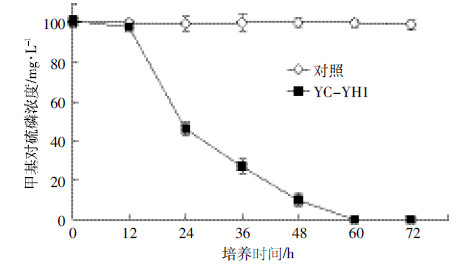
|
| 图 3 菌株YC-YH1 对甲基对硫磷的降解曲线 Figure 3 Degradation curve of methyl parathion by strain YC-YH1 |
以菌株YC-YH1的基因组为模板,以设计的引物分别进行PCR扩增并经电泳检测产物(图 4),其中mpd与ophC2基因检测结果为阳性。对获得的PCR产物进行回收、克隆并测序,测序结果进行BLAST比对分析。mpd基因与Pseudomonas sp. WBC-3的mpd基因相似性为100%,ophC2基因与Stenotrophomonas sp. SMSP-1的ophC2基因相似性为99%。将获得的mpd与ophC2基因序列递交至GenBank,登录号分别为KP207597和KP207598。这两种基因分别报道自Pseudomonas sp. WBC-3[18]和Pseudomonas pseudoalcaligenes[19],在这两种菌中均只含有一种降解酶。本研究首次报道了在一个菌株内同时存在这两种有机磷水解酶的情况。
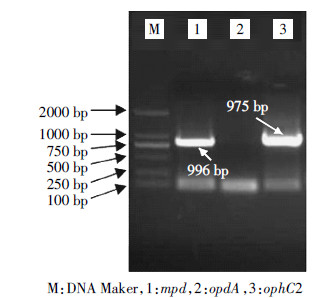
|
| 图 4 甲基对硫磷降解相关基因检测 Figure 4 Detection of methyl parathion degrading genes |
根据已有报道[20, 21],mpd和ophC2分别编码甲基对硫磷水解酶MPH和有机磷水解酶OPHC2,它们都能作用于有机磷农药的P-O酯键,进行水解作用。MPH和OPHC2能够将甲基对硫磷水解为二甲基硫代硫酸酯和对硝基苯酚。因此,可以初步判断,菌株YC-YH1通过对硝基苯酚途径对甲基对硫磷进行水解。
2.4 基于HPLC-MS的甲基对硫磷代谢产物检测根据甲基对硫磷降解相关基因检测的结果,初步判断菌株YC-YH1降解甲基对硫磷生成对硝基苯酚。因此,利用HPLC-MS对甲基对硫磷及其代谢产物进行检测,在负离子模式下质谱分析检出质荷比分别为m/z=138.100 0[M-H]-、m/z=141.300 0[M-H]-的两种不同物质,其中前者与对硝基苯酚的分子量相吻合(图 5A),后者推测为甲基对硫磷降解另一产物二甲基硫代硫酸酯;以对硝基苯酚标准品进行质谱负离子模式扫描分析,在m/z=138.1000[M-H]-处出现目标峰(图 5B),据此可判断YC-YH1中的甲基对硫磷水解酶MPH和有机磷水解酶OPH将溶液中的甲基对硫磷水解为对硝基苯酚和二甲基硫代硫酸酯。通过HPLC-MS对代谢中间产物进行了分析,得出菌株YC-YH1是通过对硝基苯酚途径降解甲基对硫磷,与已报道的Pseudomonas aeruginosa HS-D38[22]和Klebsiella sp. MP-6[23]的代谢方式均一致。
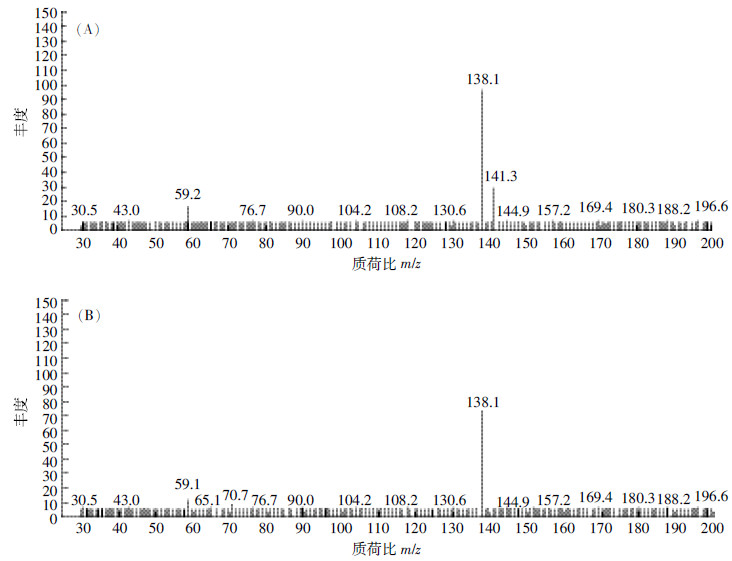
|
| 图 5 菌株YC-YH1降解甲基对硫磷后代谢产物质谱(A)和对硝基苯酚标准品质谱(B) Figure 5 Mass spectral analysis of metabolic intermediates of methyl parathion by strain YC-YH1(A) and p-nitrophenol standard (B) |
本研究使用HPLC法对甲基对硫磷及对硝基苯酚浓度进行检测,结果如图 6所示。图 6A和图 6B分别为甲基对硫磷与对硝基苯酚标准品的HPLC检测结果,甲基对硫磷的保留时间为4.21 min,对硝基苯酚的保留时间1.59 min;图 6C和图 6D为接入菌株YC-YH1后未进行培养时甲基对硫磷和对硝基苯酚的色谱峰,可以看出此时培养液中无对硝基苯酚的存在;接菌培养72 h后,甲基对硫磷色谱峰消失(图 6E),而此时检测到对硝基苯酚的浓度为43.7 mg·L-1(图 6F)。因此,菌株YC-YH1可高效降解甲基对硫磷,在72 h内将100 mg·L-1甲基对硫磷彻底降解,并生成对硝基苯酚。
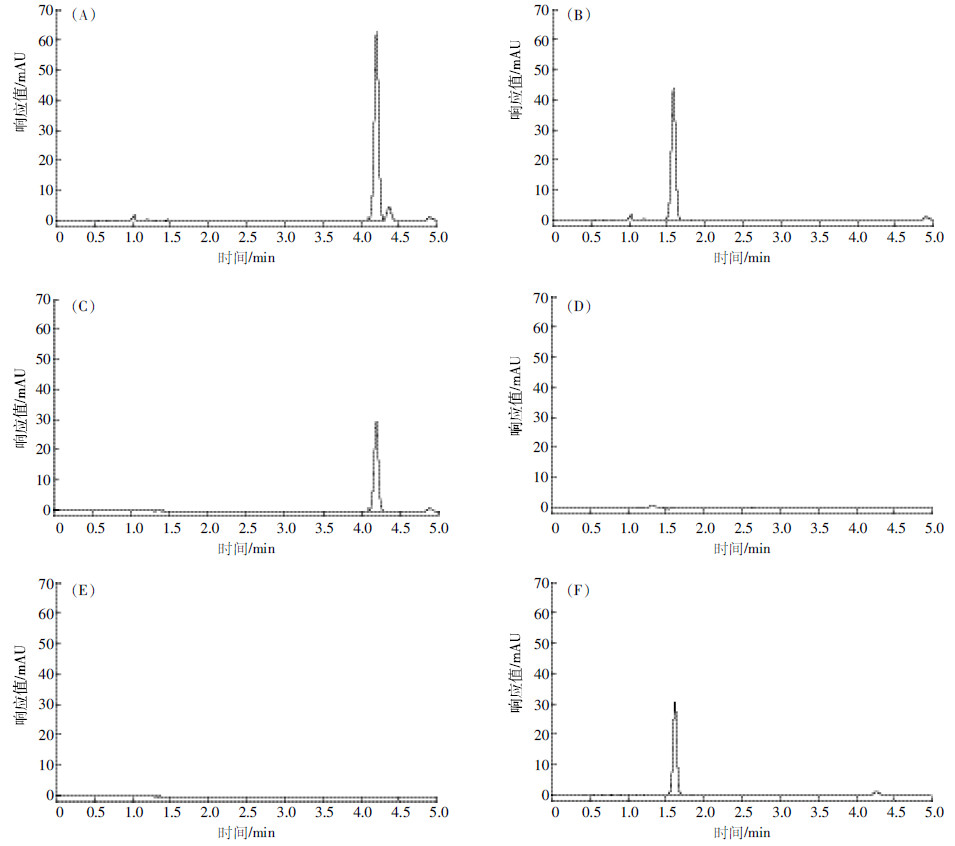
|
|
(A)甲基对硫磷标准品;(B)对硝基苯酚标准品;(C)0 h培养液中甲基对硫磷检测;(D)0 h培养液中对硝基苯酚检测;(E)72 h培养液中甲基对硫磷检测;(F):72 h培养液中对硝基苯酚检测 (A) methyl parathion standard;(B) p-nitrophenol standard;(C) methyl parathion in culture at 0 h;(D) p-nitropehnol in culture at 0 h;(E) methyl parathion in culture at 72 h;(F) p-nitropehnol in culture at 72 h 图 6 甲基对硫磷与对硝基苯酚HPLC检测 Figure 6 HPLC analyses of methyl parathion and p-nitrophenol |
在污染物的生物降解过程中,代谢中间产物的积累会反馈抑制上游反应酶的活性,甚至抑制菌株的生长[24]。某些代谢中间产物还可破坏环境微生物的多样性,影响环境微生态,如毒死蜱降解后的产物三氯吡啶酚已被证实能够显著减少环境微生物多样性,同时导致毒死蜱的降解受抑制[25, 26]。本研究检测了甲基对硫磷降解中间代谢产物对菌株YC-YH1生长的影响,结果如图 7所示。图 7A表明菌株YC-YH1在含甲基对硫磷的固体LB平板上生长72 h后,在滤纸片周围出现了黄色水解圈,但菌株基本没有生长,可以判断YC-YH1的代谢中间产物对菌株的生长产生了显著的抑制作用,提取黄色水解圈中的物质并进行HPLC-MS分析,结果表明其主要成分为对硝基苯酚;图 7B表明菌株YC-YH1在含对硝基苯酚的固体LB平板上生长72 h后,平板中对硝基苯酚的颜色(黄色)并无任何变化,且菌株的生长同样也受到显著抑制;图 7C可以看出菌株YC-YH1在不含甲基对硫磷和对硝基苯酚的固体LB平板上培养72 h时,生长良好。综上所述,菌株YC-YH1降解甲基对硫磷产生对硝基苯酚,而对硝基苯酚的积累会对菌株YC-YH1的生长产生显著的抑制作用。
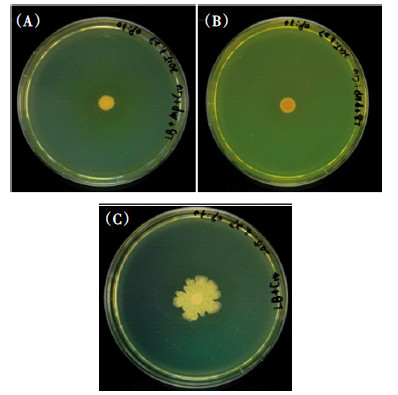
|
|
(A) YC-YH1在含甲基对硫磷平板生长72 h;(B) YC-YH1在含对硝基苯酚平板生长72 h;(C) YC-YH1在普通LB平板上生长72 h (A) Growth of strain YC-YH1 on plate containing methyl parathion at 72 h;(B) Growth of strain YC-YH1 on plate containing p-nitrophenol at 72 h;(C) Growth of strain YC-YH1 on normal LB plate at 72 h 图 7 甲基对硫磷代谢产物对菌株YC-YH1的影响 Figure 7 Effect of methyl parathion metabolites on growth of strain YC-YH1 |
污染物的降解,在很大程度上减小了污染物对环境及人体健康的危害。但不彻底降解所产生的代谢中间产物,不仅在毒性上与母体分子产生较大差异,同时理化性质及结构等均不同于母体分子。总的来说,大多数代谢中间产物的毒性通常较母体分子毒性低,如三氯吡啶酚作为毒死蜱的代谢中间产物毒性被降低[27],这也导致了对于代谢中间产物危害的认识不足;同时,许多水溶性差的母体分子经过降解后得到的中间产物往往具有良好的水溶性,如三氯吡啶酚和对硝基苯酚,这使得污染物的代谢中间产物在环境中具有更好的流动性[28],也更容易进入地下水环境中,因而导致了更大的潜在威胁;而且,部分代谢中间产物的积累可导致母体分子降解受到抑制,一方面是代谢中间产物抑制了菌株的生长从而导致降解过程的减缓,另一方面是代谢中间产物的积累抑制降解途径上游酶的活性。
3 结论研究获得了甲基对硫磷水解酶基因mpd与有机磷水解酶基因ophC2,并通过共降解实现了对甲基对硫磷的彻底降解。
首次证实了甲基对硫磷的降解存在代谢中间产物抑制效应,为甲基对硫磷的生物降解研究及实际应用提供了参考依据。
| [1] | 马晓艳, 蒋建荣, 潘喻佳. 2012年苏州市水生蔬菜中有机磷农药残留状况监测及分析[J]. 中国卫生检验杂志, 2013, 23(15):3112-3113.MA Xiao-yan, JIANG Jian-rong, PAN Yu-jia. Analysis of organophosphorus pesticide residues in aquatic vegetables in Suzhou in 2012[J]. Chinses Journal of Health Laboratory Technology, 2013, 23(15):3112-3113. |
| [2] | Liu F Y, Hong M Z, Liu D M, et al. Biodegradation of methyl parathion by Acinetobacter radioresistens USTB-04[J]. Journal of Environmental Sciences(China), 2007, 19(10):1257-1260. |
| [3] | 杨卫萍, 陆天友. 贵州省饮用水源地有机磷农药污染研究现状[J]. 西南给排水, 2014, 36(4):51-55.YANG Wei-ping, LU Tian-you. Status of organophosphorus pesticide contamination of drinking water sources in Guizhou Province[J]. Shouthwest Water and Wastewater, 2014, 36(4):51-55. |
| [4] | Lu P, Li Q, Liu H, et al. Biodegradation of chlorpyrifos and 3, 5, 6-trichloro-2-pyridinol by Cupriavidus sp. DT-1[J]. Bioresource Technology, 2013, 127:337-342. |
| [5] | Deng S, Chen Y, Wang D, et al. Rapid biodegradation of organophosphorus pesticides by Stenotrophomonas sp. G1[J]. Journal of Hazardous Materials, 2015, 297:17-24. |
| [6] | Song J, Gu J, Zhai Y, et al. Biodegradation of nicosulfuron by a Talaromyces flavus LZM1[J]. Bioresource Technology, 2013, 140:243-248. |
| [7] | 李 阳, 孙庆元, 宗 娟, 等. 一株降解氟磺胺草醚的黑曲霉菌特性[J]. 农药, 2009, 48(12):878-880.LI Yang, SUN Qing-yuan, ZONG Juan, et al. Characteristics of fungus degeneration fomesafen[J]. Agrochemicals, 2009, 48(12):878-880. |
| [8] | 张 霞, 张书泰, 谢顺昌, 等. 一株甲基对硫磷降解菌-布朗克假丝酵母JMUPMD-1的分离与鉴定[J]. 激光生物学报, 2014, 23(2):183-188.ZHANG Xia, ZHANG Shu-tai, XIE Shun-chang, et al. Isolation and identification of a parathion-methyl degrading fungus-Candida blankii JMUPMD-1[J]. Acta Laser Biology Sinica, 2014, 23(2):183-188. |
| [9] | 杨德玉, 李小明, 戴速航, 等. 一株耐盐性莠去津高效降解菌Halomonas sp. SY-AD-9的分离、鉴定及其特性[J]. 农药, 2015, 54(1):34-38.YANG De-yu, LI Xiao-ming, DAI Su-hang, et al. Isolation, identification and characterization of an salt tolerance atrazine-degrading bacteria SY-AD-9(Halomonas sp.)[J]. Agrochemicals, 2015, 54(1):34-38. |
| [10] | Dejonghe W, Berteloot E, Goris J, et al. Synergistic degradation of linuron by a bacterial consortium and isolation of a single linuron-degrading Variovorax strain[J]. Applied and Environmental Microbiology, 2003, 69(3):1532-1541. |
| [11] | Wang S, Zhang C, Yan Y. Biodegradation of methyl parathion and p-nitrophenol by a newly isolated Agrobacterium sp. strain Yw12[J]. Biodegradation, 2012, 23(1):107-116. |
| [12] | 史延华. 农药降解菌施氏假单胞菌YC-YH1的分离鉴定及降解机理研究[D]. 北京:中国农业科学院, 2015.SHI Yan-hua. Isolation and identification of pesticides-degrading bacterium Pseudomonas stutzeri YC-YH1 and study of degradation mechanism[D]. Beijing: Chinese Academy of Agricultural Sciences, 2015. |
| [13] | 罗小溪, 高建忠, 陈再忠, 等. 新型脱氮菌Rhizobium radiobacter的分离鉴定及其硝化特征分析[J]. 生物技术通报, 2015, 31(5):167-172.LUO Xiao-xi, GAO Jian-zhong, CHEN Zai-zhong, et al. Isolation and identification of novel denitrifier strain Rhizobium radiobacter and its nitrifying characterisation[J]. Biotechnology Bulletin, 2015, 31(5):167-172. |
| [14] | Yang C, Liu N, Guo X, et al. Cloning of mpd gene from a chlorpyrifos-degrading bacterium and use of this strain in bioremediation of contaminated soil[J]. FEBMS Microbiology Letters, 2006, 265(1):118-125. |
| [15] | Fanyunliu W D S L. Isolation, purification and characterization of a new organphosphorus hydrolase OPHC2[J]. Chinese Science Bulletin, 2004, 49(3):268-272. |
| [16] | Horne I, Sutherland T D, Harcourt R L, et al. Identification of an opd(organophosphate degradation) gene in an Agrobacterium isolate[J]. Applied and Environmental Microbiology, 2002, 68(7):3371-3376. |
| [17] | 段海明, 王开运, 王 冕, 等. 蜡状芽孢杆菌Hy-1降解甲基对硫磷和毒死蜱的影响因素研究[J]. 农业环境科学学报, 2010, 29(3):437-443.DUAN Hai-ming, WANG Kai-yun, WANG Mian, et al. Degradative characteristics of Bacillus cereus HY-1 to methyl-parathion and chlorpyrifos[J]. Journal of Agro-Environment Science, 2010, 29(3):437-443. |
| [18] | Dong Y, Bartlam M, Sun L, et al. Crystal structure of methyl parathion hydrolase from Pseudomonas sp. WBC-3[J]. Journal of Molecular Biology, 2005, 353(3):655-663. |
| [19] | Wu N, Deng M, Shi X, et al. Isolation, purification and characterization of a new organphosphorus hydrolase OPHC2[J]. Chinese Science Bulletin, 2004, 49(3):268-272. |
| [20] | Shen Y J, Lu P, Mei H, et al. Isolation of a methyl parathion-degrading strain Stenotrophomonas sp. SMSP-1 and cloning of the ophC2 gene[J]. Biodegradation, 2010, 21(5):785-792. |
| [21] | Li X H, He J A, Li S P. Isolation of a chlorpyrifos-degrading bacterium, Sphingomonas sp. strain Dsp-2, and cloning of the mpd gene[J]. Research in Microbiology, 2007, 158(2):143-149. |
| [22] | 郑永良, 刘德立, 陈舒丽, 等. 一株甲基对硫磷高效降解菌的鉴定及特性研究[J]. 环境科学研究, 2006, 19(4):100-104.ZHENG Yong-liang, LIU De-li, CHEN Shu-li, et al. Study on isolation and characteristic identification of a novel high-efficiency degrading strain methyl parathion[J]. Research of Environmental Sciences, 2006, 19(4):100-104. |
| [23] | 耿芳芳, 王 慧, 秦 伟, 等. 基于构建微生物传感器的甲基对硫磷降解菌的分离鉴定及其降解特性研究[J]. 农业资源与环境学报, 2014, 31(6):521-526.GENG Fang-fang, WANG Hui, QIN Wei, et al. Isolation and characterization of methyl parathion-degrading bacteria based on microbial sensor construction[J]. Journal of Agricultural Resources and Environment, 2014, 31(6):521-526. |
| [24] | 黄 栩, 骆苑蓉, 胡 忠, 等. 持久性有机污染物(POPs)生物修复研究进展[J]. 环境科学学报, 2006, 26(3):353-361.HUANG Xu, LUO Yuan-rong, HU Zhong, et al. Recent advance in the study of persistent organic pollutants bioremediation[J]. Acta Scientiae Circumstantiae, 2006, 26(3):353-361. |
| [25] | Wang D, Xue Q, Zhou X, et al. Isolation and characterization of a highly efficient chlorpyrifos degrading strain of Cupriavidus taiwanensis from sludge[J]. Journal of Basic Microbiology, 2015, 55(2):229-235. |
| [26] | Li J, Liu J, Shen W, et al. Isolation and characterization of 3, 5, 6-trichloro-2-pyridinol-degrading Ralstonia sp. strain T6[J]. Bioresource Technology, 2010, 101(19):7479-7483. |
| [27] | 张 扬, 王 飞, 黄 彦, 等. 罗尔斯顿菌(Ralstonia sp. )T6对三氯吡啶醇污染土壤的修复及能完全矿化毒死蜱工程菌株的构建[J]. 生态与农村环境学报, 2014, 30(6):761-767.ZHANG Yang, WANG Fei, HUANG Yan, et al. Remediation of 3, 5, 6-Trichloro-2-pyridinol polluted soil with Ralstonia sp. T6 and construction of a chlorpyrifos-mineralizing engineered strain[J]. Journal of Ecology and Rural Environment, 2014, 30(6):761-767. |
| [28] | Cao L, Xu J, Wu G, et al. Identification of two combined genes responsible for dechlorination of 3, 5, 6-trichloro-2-pyridinol(TCP) in Cupriavidus pauculus P2[J]. Journal of Hazardous Materials, 2013, 260:700-706. |
 2015, Vol. 34
2015, Vol. 34