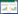文章信息
- 谢登科, 符安, 林耀东, 秦华明, 莫测辉
- XIE Deng-ke, FU An, LIN Yao-dong, QIN Hua-ming, MO Ce-hui
- BDE-209对土壤微生物PLFAs特性的影响
- Effect of BDE-209 on Characteristics of Soil Microbial PLFAs
- 农业环境科学学报, 2015, 34(3): 478-484
- Journal of Agro-Environment Science, 2015, 34(3): 478-484
- http://dx.doi.org/10.11654/jaes.2015.03.009
-
文章历史
- 收稿日期:2014-10-18
十溴联苯醚(Decabromodiphenyl ether,BDE-209)是一种高溴代持久性有机污染物,它作为阻燃剂被大量添加于塑料、电子产品(印刷线路板)、家具及纺织品中[1, 2]。由于十溴联苯醚结构稳定且不易被降解,在自然界中的衰减周期长,并可以通过转化、运输、累积、生物放大等作用,使得大气、水体和土壤环境中十溴联苯醚的含量越来越高[3],最终影响环境,危害人体健康[1, 2, 4]。近年来,十溴联苯醚的污染日益受到关注[1],对其在各种环境介质中的含量检测、对水生环境系统的作用机制、毒理等方面的研究较多[5, 6],但对其在土壤环境微生态系统产生影响的相关研究报道并不多见。
土壤微生物群落被认为是土壤生态系统变化的预警及敏感指标,指示土壤质量变化[7]。磷脂脂肪酸(Phospholipid fatty acids,PLFAs)是构成生物细胞膜的主要成分,只存在于活细胞膜中,对环境因素敏感,特定菌群PLFAs的变化可反映出原位土壤微生物活体生物量与菌群结构[8],利用磷脂脂肪酸法分析土壤微生物群落结构是一种可靠的分析方法[9]。本文采用室内模拟的方法,通过考察在不同浓度的BDE-209污染下土壤中微生物的磷脂脂肪酸的变化,以期为污染胁迫环境生态毒理的早期诊断以及筛选污染胁迫下有效的生物标志物及环境监测提供更多依据。 1 材料与方法 1.1 土壤性质
试验土壤样品采自广州市华南农业大学试验基地表层0~20 cm 的农田水稻土。土壤性质如下:pH 值6.54,阳离子交换量(CEC)26.69 cmol·kg-1,有机质含量18.73 g·kg-1,全N 0.74 g·kg-1,有效P 10.42 mg·kg-1,速效K 92.06 mg·kg-1,BDE-209未检出。 1.2 实验设计
土壤风干磨碎过2 mm筛,分装于12个1 L玻璃器皿中,每个装入400 g,设置3个浓度梯度,同时设置对照,即CK、T1、T2和T3分别代表BDE-209的浓度为0、1、10、100 mg·kg-1,每个处理3个重复。
以甲苯为溶剂,配置浓度为5000 mg·L-1的十溴联苯醚母液。添加母液使各处理组污染物浓度满足上述设置,并确保每组中甲苯的含量一致。定期搅拌使污染物混合均匀,经测定土壤BDE-209含量T1为(0.80±0.07)mg·kg-1,T2为(8.45±0.35)mg·kg-1,T3为(89.51±2.56)mg·kg-1,CK组未检出BDE-209。定时定量添加去离子水维持烧杯中土壤含水率为田间最大含水率的60%,将所有处理组置于恒温30 ℃暗室培养。在添加污染物的第1、7、15、28、56 d采样测定。 1.3 测定方法
土壤磷脂脂肪酸的测定在Schutter[10]的方法上加以改进,具体步骤为:将2 mL 的0.2 mol·L-1的KOH甲醇溶液和500 mg的土样加到10 mL的玻璃离心管中,混合均匀,充入氮气为保护气,在37 ℃下温育1 h(脂肪酸释放,并甲酯化,样品10 min涡旋1次)。温育结束后,加入0.4 mL 1.0 mol·L-1的醋酸溶液中和pH值,充分摇匀。随后加入2 mL正己烷,充分涡旋振荡,再加入2 mL的去离子水,3000 r·min-1离心10 min,使磷脂脂肪酸(PLFA)转移到有机相中,取上层有机相转至干净试管中,用氮气吹干后溶解在990 μL正己烷∶甲基丁基醚(1∶1,V/V)的溶液中,并加入10 μL甲酯化的C19:0为内标。
实验检测采用岛津 GCMS-QP 2010。色谱柱为DB-5MS(30 m×0.25 mm×0.25 μm)石英毛细管柱,GC-MS分析条件:柱温140 ℃保持2 min,3 ℃·min-1升温到260 ℃,保持1 min,进样口温度为250 ℃,载气为He(1 mL·min-1),离子源温度230 ℃,质谱仪采用电子电离方式(EI),电子能量为70 eV,质谱全扫描范围50~500 m/z。脂肪酸定量用峰面积和内标曲线法,通过与标准品的保留时间比对和根据谱库检索定性,PLFA含量用nmol·g-1表示。 1.4 磷脂脂肪酸命名及一些特征性的PLFAs组分之间的比值
脂肪酸采用Frostegard等[11]方法命名,分子式以“(i/a)X:YωZ(c/t)”表示,其中X代表脂肪酸分子的C原子总数,Y代表不饱和烯键的数目,ω代表烯键距离羧基的位置,Z为烯键或环丙烷链的位置,后缀c和t分别代表双键的顺式和反式结构,前缀i(iso)代表顺式支链、a(anteiso)代表反式支链。cy代表环丙基,br 表示甲基链的位置未知,10Me表示第10个碳原子的甲基(从羟基端起)。
磷脂脂肪酸与微生物生物量间存在显著的正相关关系[12],磷脂类化合物的量可准确地表示土壤不同类型微生物的生物量,一些特征性的PLFAs组分之间的比值也常常用来表征土壤微生物在受到污染胁迫时的响应指标。本文用到了下列一些脂肪酸之比:真菌与细菌之比[F/B,(18:2ω6,9/(14:0+ i14:0+15:0+i15:0+a15:0+i16:0+16:0+16:1ω7t+16:1ω7c+i17:0+a17:0+cy17:0+17:0+18:1ω7t+18:1ω9t+18:0+cy19:0)],革兰氏阳性细菌与革兰氏阴性细菌之比[(i14:0+i15:0+a15:0+i16:0+i17:0+a17:0)/(16:1ω7t+16:1ω7c+18:1ω7t+18:1ω9t+cy17:0+cy19:0,16:1ω7t和16:1ω7c的比值。 1.5 数据分析
采用方差分析、主成分分析等数学统计方法,分析软件为Microsoft Excel 2007及SPSS16.0统计软件。 2 结果与分析 2.1 BDE-209胁迫下土壤微生物PLFAs的组成和含量的变化
BDE-209处理下土壤磷脂脂肪酸的类型和含量如表 1所示。BDE-209胁迫下土壤微生物包括内标19:0在内,共检测出20种磷脂脂肪酸,磷脂脂肪酸的碳链长度从C14到C19,包含各种饱和脂肪酸,如14:0、15:0、16:0等,其中脂肪酸16:0含量最高;单不饱和脂肪酸如16:1ω7t、16:1ω7c、18:1ω7t等,多不饱和脂肪酸如18:2ω6,9,分支脂肪酸如i15:0、a15:0、i16:0等,环化脂肪酸如cy17:0和cy19:0。可见,在BDE-209≤100 mg·kg-1胁迫下,土壤微生物群落PLFAs种类还是较丰富的。
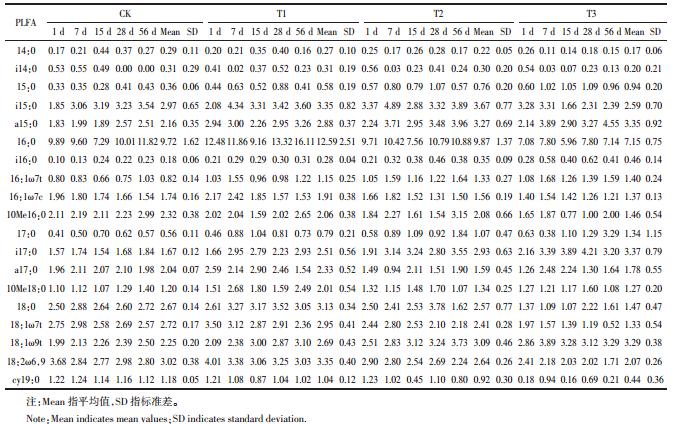 |
土壤中磷脂脂肪酸的组成可以表示土壤微生物群落的生物量和结构。对不同浓度BDE-209胁迫下土壤微生物群落结构的分析见表 2。与对照组相比,T1、T2组在处理期内土壤总微生物量和细菌均显著增加,同一时期T1组的增量要高于T2组,其中以T1组第7 d增加的最多,比对照组增加了25.07%;而T3组的土壤总微生物量和细菌较CK少,说明低浓度的BDE-209对土壤总微生物量和细菌有一定的刺激作用,促进微生物的生长,高浓度的BDE-209产生了毒害作用,抑制微生物的生长。在BDE-209不同浓度处理下,总微生物量和细菌的变化趋势相同,说明该土壤中主要的微生物类群是细菌;对于真菌和放线菌来说,在BDE-209浓度为1 mg·kg-1时(T1),真菌和放线菌均呈增加趋势,其中以第7 d增加的最多,其中真菌增加了19.01%,放线菌增加了42.17%。而在较高BDE-209浓度(T2、T3)时,真菌和放线菌均呈减少趋势,浓度越高,减少程度越大,其中以100 mg·kg-1(T3)处理最低,此时真菌在第56 d比对照组减少最多达38.93%,而放线菌在第15 d比对照组减少最多达38.99%。
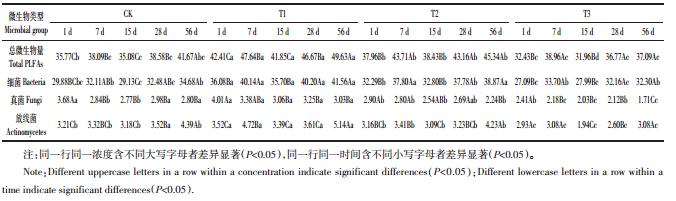 |
对BDE-209胁迫下土壤微生物PLFAs组成和含量的进一步分析结果如图 1所示。与对照(CK)相比,T1处理可将PLFAs的变化分为3类:第1类为下降型,包括4种生物标记,其中10Me16:0指示放线菌,cy19:0、14:0、i14:0指示细菌;第2类为一般增加型(增加50%以下),包括11种生物标记,其中18:2ω6,9指示真菌,其余的指示细菌;第3类为中等增加型(增加50%~100%),包括4种生物标记,其中i17:0、i16:0、15:0指示细菌,10Me18:0指示放线菌。T2处理可将PLFAs的变化分为4类:第1类为下降型,包括9种生物标记,其中18:2ω6,9指示真菌,10Me16:0指示放线菌,其余的指示细菌;第2类为一般增加型(增加50%以下),包括4种生物标记,其中10Me18:0指示放线菌,其余的指示细菌;第3类为中等增加型(增加50%~100%),包括5种生物标记,全部指示细菌;第4类为显著增加型(增加100%以上),生物标记15:0指示细菌。T3处理可将PLFAs的变化分为4类:第1类为下降型,包括11种生物标记,其中10Me16:0指示放线菌,18:2ω6,9指示真菌,其余的指示细菌;第2类为一般增加型(增加50%以下),包括2种生物标记,其中10Me18:0指示放线菌,18:1ω9t指示革兰氏阴性细菌;第3类为中等增加型(增加50%~100%),包括2种生物标记,其中a15:0指示革兰阳性菌,16:1ω7t指示革兰氏阴性菌;第4类为显著增加型(增加100%以上),包括i17:0、17:0、i16:0和15:0,全部指示细菌。
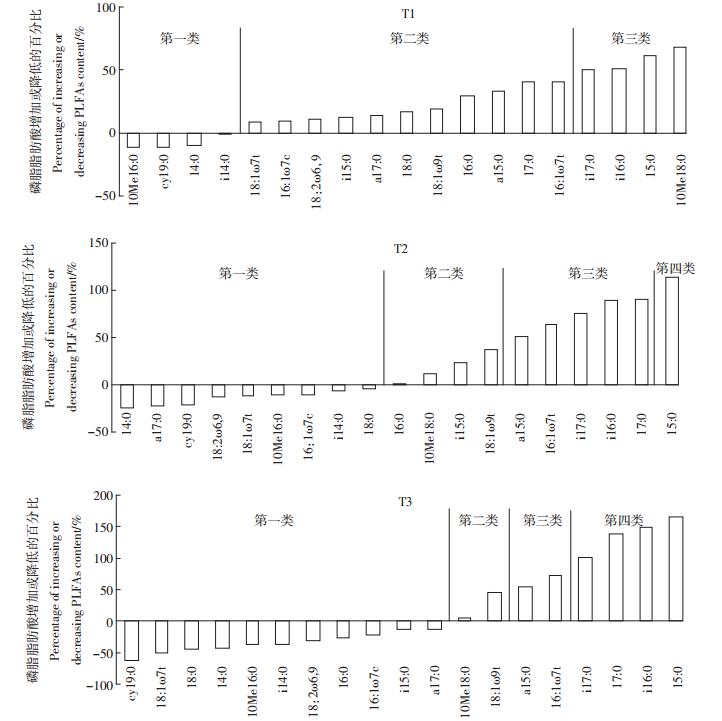
|
| 图 1 BDE-209 处理下土壤微生物 PLFAs 含量的变化分类 Figure 1 Effect of BDE-209 on changes of soil microbial of PLFA contents |
结果分析表明,与对照组相比,T1、T2和T3处理脂肪酸增加前五位中都含有15:0、i16:0和i17:0,且它们均指示细菌,说明BDE-209胁迫下,对细菌可能有刺激作用,促进它们的增长。 2.2 BDE-209胁迫下土壤微生物PLFAs特征性比值分析
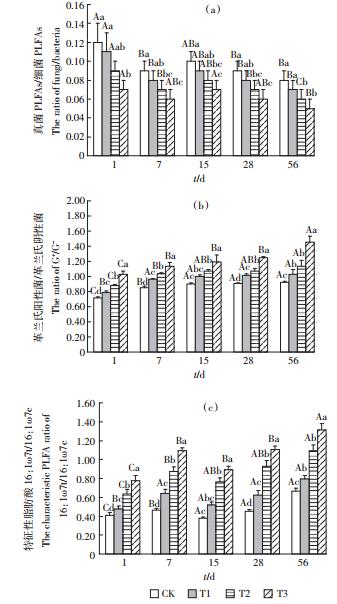
土壤微生物PLFAs的一些特征性比值如真菌/细菌、G+/G-和16:1ω7t/16:1ω7c等可以作为反映土壤生态系统缓冲能力的重要指标,指示污染物对土壤生态系统的干扰[13],或用来表征细胞受到胁迫时的指示指标[14]。
对BDE-209胁迫下土壤微生物PLFAs的一些特征性比值进行分析,结果如图 2所示。图 2(a)的结果表明,无论是否添加BDE-209处理,真菌/细菌值总体上均随处理时间的延长而下降,在同一时期,处理组的真菌/细菌值要比对照组小,且与BDE-209浓度呈负相关。这说明在此实验条件下,真菌对BDE-209更敏感,而细菌对BDE-209更耐受。图 2(b)、图 2(c)的结果表明,无论是否添加BDE-209处理,各样本的G+/G-、16:1ω7t/16:1ω7c值均随处理时间的延长总体呈增加趋势,在同一时期,处理组的G+/G-、16:1ω7t/16:1ω7c值要比对照组大,且与BDE-209浓度呈正相关,浓度越大,增加的越显著。这说明BDE-209浓度越高,对土壤生态系统的干扰越大,同时说明革兰氏阴性菌对BDE-209污染的响应更为敏感,革兰氏阳性细菌更容易存活,而含16:1ω7t的微生物比含16:1ω7c的微生物抗逆性更强,更容易在高浓度的BDE-209环境下生存。
|
| 同一浓度含不同大写字母者差异显著 (P<0.05 ), 同一时间含不同小写字母者差异显著 (P<0.05 ) Different uppercase letters in a concentration indicate significant differences (P<0.05 ); Different lowercase letters in a time indicate significant differences (P<0.05 )图 2 DE-209 处理对真菌/细菌、 G + /G-和 16: 1ω7t/16: 1ω7c 的影响 Figure 2 Effect of BDE-209 on fungi/ bacteria, G + /G- and 16: 1ω7t/16: 1ω7c ratios |
为进一步分析不同浓度BDE-209处理对土壤微生物群落结构的影响,对供试土壤微生物磷脂脂肪酸进行主成分分析。结果表明,第一主成分解释了微生物群落结构变异的55.63%,第二主成分解释了微生物群落结构变异的44.03%,并能够把不同浓度的BDE-209区分开来,大致呈现CK、T1、T2和主成分一呈显著正相关,T3和主成分二呈显著正相关。
根据主成分因子得分矩阵,将两个主成分分别表示为
Y1=1.088CK+0.778T1+0.069T2-1.210T3
Y2= -0.849CK -0.493T1+0.314T2+1.747T3
由磷脂脂肪酸在各个BDE-209处理下的数值,可得出磷脂脂肪酸在2个主成分上面的得分,其中:16:0、18:0、18:1ω7t、18:2ω6,9在主成分一上面的得分较高,可以认为主成分一是它们的代表因子,16:0、18:0是细菌的特征脂肪酸,18:1ω7t是革兰氏阴性菌的特征脂肪酸,18:2ω6,9是真菌的特征脂肪酸,说明革兰氏阴性菌和真菌对高浓度BDE-209较敏感,16:0、18:0表征的细菌在主成分一中是优势种群;i17:0、a15:0在主成分二上面的得分较高,可以认为主成分二是它们的代表因子,i17:0、a15:0都是革兰氏阳性菌的特征脂肪酸,说明革兰氏阳性菌对高浓度的BDE-209抗逆性较强,在主成分二中是优势种群。 3 讨论
磷脂脂肪酸被广泛作为土壤微生物生物量与微生物生物群落的生物标记。本研究发现,与对照组相比,不同浓度BDE-209处理下土壤总微生物量都是先升高、后随着污染物浓度的增大而下降。BDE-209作为一种有机污染物,在进入土壤后,低浓度的BDE-209能够作为碳源被微生物所利用,从而刺激微生物的生长,高浓度的BDE-209可以对细胞DNA造成难以修复的损伤[15],抑制微生物的生长繁殖,甚至直接导致细胞死亡[16]。
污染物对土壤微生物区系和群落结构的影响因不同的研究对象而不同。通过比较不同浓度BDE-209污染下微生物群落结构和一些特征性的PLFAs组分之间的比值变化发现,随着不同浓度BDE-209的添加,土壤总微生物量、细菌、真菌和放线菌的量增加或减少,但增加或减少的对应浓度不一样:在BDE-209浓度为1、10 mg·kg-1时,土壤总微生物量和细菌的量增加,BDE-209浓度为100 mg·kg-1土壤总微生物量和细菌的量减少,而真菌和放线菌量在BDE-209浓度为10、100 mg·kg-1出现减少,是由于不同微生物对BDE-209耐受能力不同,同时F/B随着BDE-209浓度的增加而减少,也说明细菌对BDE-209的耐受能力大于真菌。这与相关研究有类似之处[17],也可能与本研究中所测定的指示真菌的磷脂脂肪酸为18:2ω6,9有关。此外,目前直接从土壤中提取的真菌磷脂脂肪酸种类仍十分有限,由此也可能造成很多种类的真菌在土壤中并没有真正反映出来[18]。
革兰氏阳性菌和革兰氏阴性菌的比值(G+/G-)可作为指示环境胁迫时的指标[19]。在本研究中,在BDE-209胁迫下,G+/G-随BDE-209浓度的增加而增加,尤其是含i17:0、a15:0的微生物明显增多,由于i17:0、a15:0指示的均为革兰氏阳性细菌,而革兰氏阳性属于多样性和种类都非常丰富的细菌类群。张新颖[20]在研究中也发现,G+比G-对持久性有机污染物芘的耐受性更强。但是Su等[21] 的研究表明,革兰氏阳性菌对多环芳烃的毒性也很敏感,Frostegard等[22]也发现多环芳烃耐受菌和多环芳烃降解菌很多都属于革兰氏阴性菌,说明不同环境中的不同类型的微生物对不同的污染物的响应是不同的。进一步的主成分分析表明,BDE-209浓度越低,其在主成分分布图上向X轴的正方向发展,BDE-209浓度越高,其在主成分分布图上向Y轴正方向发展,其中PC1以含16:0、18:0、18:1ω7t、18:2ω6,9 的微生物为优势种群,PC2以含i17:0、a15:0的微生物为优势种群。
利用土壤微生物PLFAs可以分析BDE-209污染胁迫引起的土壤微生物生物量和群落结构的变化,为筛选有效的污染胁迫生物标志物,以及对污染胁迫环境生态毒理进行早期诊断和环境监测提供了更多参考。 4 结论
(1)在1~100 mg·kg-1 BDE-209胁迫下,土壤微生物群落PLFAs种类还较丰富。
(2)BDE-209对以PLFAs表征的土壤细菌、真菌、放线菌和总微生物量都有显著的影响,且真菌对BDE-209更敏感,细菌对BDE-209更耐受。细菌中G-菌对BDE-209污染的响应更为敏感,G+菌更容易存活,含16:1ω7t的微生物比含16:1ω7c的微生物抗逆性更强。
(3)以PLFAs表征的土壤微生物量和群落结构对BDE-209的响应非常敏感,可以作为BDE-209污染下指示土壤质量变化的一个有效的生物指标。
| [1] | Jing Y F, Wang X T, Kun Z. Occurrence, compositional patterns, and possible sources of polybrominated diphenyl ethers in agricultural soil of Shanghai[J]. China Chemosphere, 2012, 89:936-943. |
| [2] | Braune B, Mallory M, Gilchrist H, et al. Levels and trends of organoch-lorines and brominated flame retardants in ivory gull eggs from the Canadian Arctic, 1976 to 2004[J]. Science of the Total Environment, 2007, 378(3):403-417. |
| [3] | Luo Y, Luo X J, Zhen L, et al. Polybrominated diphenyl ethers in road and farmland soils from an e-waste recycling region in Southern China, Concentrations, source profiles, and potential dispersion and deposition[J]. Science of the Total Environment, 2009, 407(3):1105-1113. |
| [4] | Sverdrup L E, Hartnik T, Mariussen E, et al. Toxicity of three halogenated flame retardants to nitrifying bacteria, red clover(T5 Rifolium pratense), and a soil invertebrate(Enchytraeus crypticus)[J]. Chemosphere, 2006, 64(1):96-103. |
| [5] | Liu W X, Li W B, Hu J, et al. Sorption kinetic characteristics of polybrominated diphenyl ethers on natural soils[J]. Environmental Pollution, 2010, 158:2815-2820. |
| [6] | Zhang K, Zhang B Z, Li S M. Regional dynamics of persistent organic pollutants(POPs) in the Pearl River Delta, China:Implications and perspectives[J]. Environmental Pollution, 2011, 159:2301-2309. |
| [7] | 于 树, 汪景宽, 李双异. 应用PLFA方法分析长期不同施肥处理对玉米地土壤微生物群落结构的影响[J]. 生态学报, 2008, 28(9):4221-4227. YU Shu, WANG Jing-kuan, LI Shuang-yi. Effect of long-term fertilization on soil microbial community structure in corn field with the method of PLFA[J]. Acta Ecologica Sinica, 2008, 28(9):4221-4227. |
| [8] | Liu B R, Jia G M, Chen J, et al. A Review of methods for studying microbial diversity in soils[J]. Pedosphere, 2006, 16(1):18-24. |
| [9] | 赵 帅, 张静妮, 赖 欣, 等. 放牧与围栏内蒙古针茅草原土壤微生物生物量碳、氮变化及微生物群落结构PLFA分析[J]. 农业环境科学学报, 2011, 30(6):1126-1134. ZHAO Shuai, ZHANG Jing-ni, LAI Xin, et al. Analysis of microbial biomass C, N and soil microbial community structure of stipa Steppes using PLFA at grazing and fenced in Inner Mongolia, China[J]. Journal of Agro-Environment Science, 2011, 30(6):1126-1134. |
| [10] | Schutter M E, Dick R P. Comparison of fatty acid methyl ester(FAME)methods for characterizing microbial communities[J]. Soil Science Society of America Journal, 2000, 64(5):1659-1668. |
| [11] | Frostegard A, Baath E, Tunlid A. Shift in the structure of soil microbial communities in limed forests as revealed by phospholipid fatty acid analysis[J]. Soil Biology and Biochemistry, 1993, 25(6):723-730. |
| [12] | Yoshitake S, Uchida M, Nakatsubo T, et al. Characterization of soil microflora on a successional glacier foreland in the high Arctic on Ellesmere Island, Nunavut, Canada using phospholipid fatty acid analysis[J]. Polar Bioscience, 2006, 19:73-84. |
| [13] | 廖 敏, 陈雪花, 陈承利. 土壤-青菜系统中铅污染对土壤微生物活性及多样性的影响[J]. 环境科学学报, 2007, 27(2):220-227. LIAO Min, CHEN Xue-hua, CHEN Cheng-li. The influence of lead contamination on soil-microbial activity and community structure diversity in a soil-greengrocery system[J]. Acta Scientiae Circumstantiae, 2007, 27(2):220-227. |
| [14] | 陈振翔, 于 鑫, 夏明芳, 等. 磷脂脂肪酸分析方法在微生物生态学中的应用[J]. 生态学杂志, 2005, 24(7):828-832. CHEN Zhen-xiang, YU Xin, XIA Ming-fang, et al. Application of phospholipid fatty acid(PLFA) analysis in microbial ecology[J]. Chinese Journal of Ecology, 2005, 24(7):828-832. |
| [15] | 许超群. 四溴联苯醚(BDE-47)对菲律宾蛤仔解毒代谢酶基因表达与毒性效应的研究[D]. 青岛:中国海洋大学, 2010. XU Chao-qun. The Primary study on gene expressions and toxic effects of BDE-47 on Venerupis philippinarum[D]. Qingdao:Ocean University of China, 2010. |
| [16] | 宋任芳. 环境中苯基缩水甘油醚和多溴联苯醚的基因和细胞毒性初步研究[D]. 广州:中国科学院研究生院, 2007. SONG Ren-fang. Basic investigation of the genotoxicity and cellular toxicology by environmental phenyl glycidyl ether(PGE) and polybrominated diphenyl ethers(PBDEs) organic pollutants[D]. Guangzhou:the Graduate School of the Chinese Academy of Sciences, 2007. |
| [17] | 蒋 琴. 电子垃圾拆解区域十溴联苯醚污染土壤的植物修复研究[D]. 杭州:浙江大学, 2013. JIANG Qin. The phytoremediation of decabromodiphenyl ether polluted soil in e-waste diamantling area[D]. Hangzhou:Zhejiang University, 2013. |
| [18] | Calderón F J, Jackson L E, Scow K M, et al. Short-term dynamics of nitrogen, microbial activity, and phospholipid fatty acid after tillage[J]. Soil Science Society of America Journa1, 2001, 65:118-126. |
| [19] | Mckinley V L, Peacock A D, White D C. Microbial communities PLFA and PHB responses to ecosystem restoration in tallgrass prairie soils[J]. Soil Biology and Biochemistry, 2005, 37(10):1946-1958. |
| [20] | 张新颖. 薦草及其根际对模拟湿地中芘污染的降解机制[D]. 上海:上海大学, 2012. ZHANG Xin-ying. Degradation mechanism of pyrene by scripus triqueter and its rhizosphere in the simulated wetland[D]. Shanghai:Shanghai University, 2012. |
| [21] | Su Y H, Yang X Y. Interactions between selected PAHs and the microbial community in rhizosphere of a paddy soil[J]. Science of the Total Environmental, 2009, 407(3):1027-1034. |
| [22] | Frostegard A, Tunlid A, Baath E. Phospholipid fatty acid composition, biomass, and activity of microbial communities from two soil types experimentally exposed to different heavy meatals[J]. Applied Environmental Microbiol, 1993, 59(11):3605-3617. |
 2015, Vol. 34
2015, Vol. 34