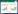文章信息
- 王鹤潼, 何蕾, 宋杰, 孙梨宗, 崔伟娜, 台培东, 贾春云, 刘宛
- WANG He-tong, HE Lei, SONG Jie, SUN Li-zong, CUI Wei-na, TAI Pei-dong, JIA Chun-yun, LIU Wan
- 改进MSAP-PCR技术应用于Cd胁迫下拟南芥DNA甲基化分析
- Assay of DNA Methylation in Arabidopsis Under Cd Stress Using Improved MSAP-PCR Technique
- 农业环境科学学报, 2015, 34(8): 1618-1624
- Journal of Agro-Environment Science, 2015, 34(8): 1618-1624
- http://dx.doi.org/10.11654/jaes.2015.08.027
-
文章历史
- 收稿日期:2015-03-16
2. 辽宁大学, 沈阳 110036;
3. 上海应用技术学院, 上海 201418
2. Liaoning University, Shenyang 110036, China;
3. Shanghai Institute of Technology, Shanghai 201418, China
DNA甲基化是表观遗传修饰的重要组成之一,其在植物生长发育、基因表达调控以及抗逆胁迫中起着至关重要的作用[1]。植物DNA甲基化是通过DNA 甲基转移酶催化S-腺苷甲硫氨酸上的甲基基团转移到DNA分子的腺嘌呤或胞嘧啶碱基上完成的。植物中主要存在着3类胞嘧啶甲基转移酶:维持DNA 甲基化转移酶(METI)家族、染色质域甲基化酶(CMT)和结构域重新甲基化酶家族[2],它们在DNA 甲基化过程中表现出不同的功能和活性。近年来,人们对植物DNA甲基化的研究愈来愈关注,并已有了较深入的了解,而且开发出一系列检测DNA甲基化的方法。根据研究内容和目的的不同,可分为检测特异性位点甲基化和全基因组甲基化两类方法,其中特异性位点甲基化的研究方法主要以重亚硫酸盐限制酶组合分析[3](COBRA)、甲基化特异性PCR[4](MSP)和亚硫酸盐测序法[5, 6, 7]为主,由于COBRA法所检测的位点受内切酶的限制,而MSP法也随着测序技术的普及而被取代,在目前特异性位点甲基化研究中亚硫酸盐测序法的使用最为广泛。
与特异性位点甲基化相比,全基因组甲基化的研究方法较多,主要包括甲基化敏感的扩增片段长度多态性法[8](Methylation Sensitive Amplification Polymorphism,MSAP)、高效液相色谱法[9]、甲基化敏感的限制性内切酶结合Southern杂交分析法[10]和甲基化特异性单核苷酸引物延伸[11](Ms-SNuPE)等。在这些方法中,MSAP法是目前在植物甲基化研究中使用最多的方法,其原因在于该方法操作简单且对实验条件要求较低,仅需PCR和一些酶促反应便可通过聚丙烯酰胺凝胶电泳获得多态性较丰富的甲基化变异结果,而且实验条件容易优化,实验成本较低,因此该方法已成为植物全基因组甲基化研究的主要方法之一。
甲基化敏感的随机引物扩增片段长度多态性[12](Methylation-sensitive Arbitrarily Primed PCR,MSAP-PCR)是另一种检测全基因组甲基化的方法,于1997年被Huang 等[13]首次应用于乳腺癌研究中,并在几年后开始应用于人类细胞在放射胁迫下的表观遗传变异[14, 15],近些年已被应用于动物生态毒理学研究[16]。该方法原理与MSAP法相似,同样以甲基化敏感性酶酶切产生的多态性差异为基础,但区别在于该方法多态性产生的原理与PCR过程中引物引发扩增效率有关,通过设计和筛选专用引物对酶切结果直接PCR扩增来得到甲基化敏感性酶酶切多态性,而不需进行接头连接及多次PCR扩增。本实验通过优化实验条件和重新设计、筛选引物,首次对MSAP-PCR法应用于植物表观遗传研究进行评估,为植物抗逆胁迫研究和环境污染的早期诊断提供一种比传统MSAP法更加简单、快速、经济的表观遗传损伤检测手段。 1 材料与方法 1.1 植物材料的培养与处理
选用拟南芥哥伦比亚型种子,经10%次氯酸钠消毒后,于灭菌超纯水中4 ℃浸种24 h打破春化。浸种后在无菌台上将种子转移至灭菌后含0、0.2、0.8、3.5、4.5 mg·L-1 Cd2+的0.5×MS培养基(0.5%庶糖)中,并于21 ℃ 10 h光照培养15 d,每个处理做三次平行实验。 1.2 DNA的提取
将相同处理三次平行实验培养的幼苗混合,使用北京康为世纪生物科技有限公司的新型植物基因组DNA提取试剂盒(CW0531)提取混合幼苗地上部DNA,并将提取的相同处理DNA混合,使用Eppendorf Biophotometor Plus检测DNA含量,根据测定的结果上样50 ng DNA进行琼脂糖电泳,用Takara DL2000 DNA Marker进行含量校准并分装成每管1 μg。 1.3 MSAP-PCR分析
在分装后的1 μg DNA中加入1 μL EcoRⅠ和5 μL EcoRⅠBuffer,加ddH2O至50 μL,37 ℃酶切1.5 h后65 ℃失活20 min,使用Takara MiniBest DNA Fragment Purification Kit对酶切产物纯化,向纯化后的DNA加入1 μL HpaⅡ或MspⅠ和5 μL Cutsmart Buffer,加ddH2O至50 μL,37 ℃酶切1.5 h后80 ℃失活20 min,产物使用上述试剂盒纯化,每次纯化按75%回收率计算两次酶切后DNA含量用于进行后续实验。
实验所用PCR体系(25 μL):1U Takara rTaq、50~250 ng DNA、2.5 μmol·L-1引物、2.5 μL 10×Buffer和200 mmol·L-1 dNTP。PCR条件:95 ℃ 3 min预变性,94 ℃变性30 s,40 ℃退火1 min,72 ℃延伸90 s,循环5次;94 ℃变性15 s,55 ℃退火15 s,72 ℃延伸1 min,循环30次[13]。
本实验共使用5条引物(表 1),其中MLG2为通用引物[12],其余4条为本实验室设计。PCR产物使用3%琼脂糖、1.8% MetaPhor琼脂糖(Lonza)、5%变性聚丙烯酰胺凝胶、5%非变性聚丙烯酰胺凝胶和4%聚丙烯酰胺凝胶(50%尿素)电泳检测,琼脂糖胶使用EB染色,聚丙烯酰胺凝胶使用银染。
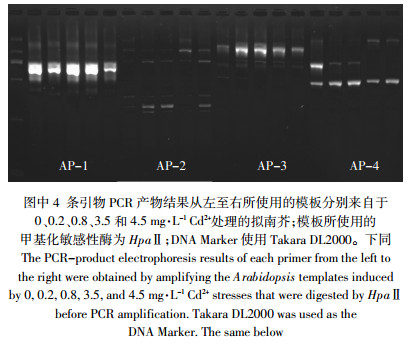
MSAP-PCR法是以随机引物PCR扩增为基础,并且两种甲基化敏感性同工酶HpaⅡ和MspⅠ的识别位点均为CCGG,所以在设计引物时通过在引物的3′端加上CCGG序列,就可将未被酶切的CCGG位点(位点发生甲基化)PCR扩增出来。然而,发生酶切的位点(位点未甲基化或去甲基化)由于引物3′端无法互补匹配且CG碱基稳定性和特异性较强未能引发PCR扩增。因此,本实验室根据随机引物PCR(AP-PCR)的通用引物序列,并结合MSAP-PCR的特征设计了4条引物(表 1),另外,还选择了在人类癌症研究中所用的引物MLG2[12]。根据实验的筛选结果,我们发现在本实验室设计的4条引物中AP-2、AP-3和AP-4三条引物的PCR产物条带分布较分散,适宜用于MSAP-PCR分析及后续实验;而AP-1引物由于其条带较少、分布过于集中而且有相对过强扩增效率的片段,在电泳中不宜分离且显色程度不好控制,不适合选用(图 1)。另外,我们还检验了MLG2引物在植物中PCR产物的分布,结果发现该引物在植物中依然有较好的扩增效率及产物分布(图 2),并可以用于植物MSAP-PCR分析。
|
| 图 1 自设计引物的筛选结果 Figure 1 Screening results of designed primers |
|
| 图 2 MLG2 引物 PCR 产物 1.8% MetaPhor 琼脂糖电泳检验 Figure 2 PCR products of MLG2 primer assayed by 1.8% MetaPhor Agarose gel electrophoresis |
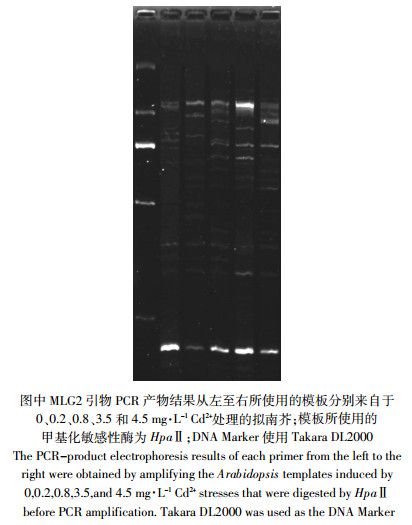
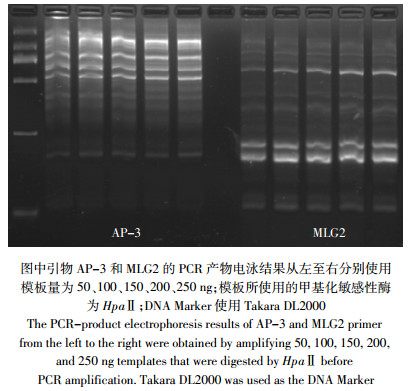
由于MSAP-PCR法需要进行两次酶切并纯化,DNA模板有一定损失,需求模板量略大,本实验对PCR模板使用量进行优化,寻求高效PCR扩增下的最低模板用量。我们选用AP-3和MLG2两个引物对PCR体系中的模板使用量进行优化(图 3),结果发现5种模板量之间的效果差异并不是很大,其中150 ng至250 ng的模板量在电泳中难以分辨出差异,综合考虑实验效果和模板使用量,我们选择使用150 ng作为实验模板量。
|
| 图 3 不同模板浓度下的 PCR 扩增效率 Figure 3 PCR amplification efficiencies under different template concentrations |
在对酶切片段长度多态性检测中,凝胶和电泳方式的选择对实验结果至关重要,电泳显色后条带的多少、强弱以及分布会直接影响实验后对电泳结果的分析和统计。在本实验中,我们使用四种不同的凝胶对PCR产物进行电泳检测,结果发现1.8% MetaPhor琼脂糖胶(超高分辨率琼脂糖)经EB染色后,由于其敏感度不够,造成条带显色不全,条带丢失严重,致使处理间变化不规律(图 2)。而在聚丙烯酰胺凝胶电泳中发现,非变性凝胶虽条带分散,且产物分布较均匀,但多态性差,很难检测到处理间差异(图 4A);变性凝胶虽多态性好,但条带过于密集,很难分辨,同时会造成银染显色难以控制,小片段显色后大片段会过显色(图 4B)。在使用4%聚丙烯酰胺凝胶(50%尿素)电泳后发现,该方法可以良好地综合前两种PAGE方法的优点,将其应用于MSAP-PCR检测即可得到良好的处理间多态性,也可使条带分散易于显色和便于分辨(图 4C)。
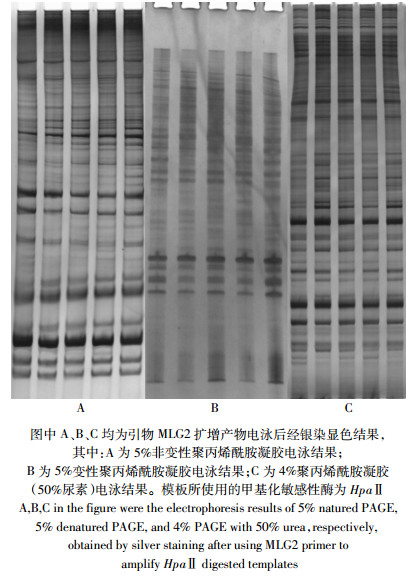
|
| 图 4 不同电泳方法检测 PCR 产物的效果比较 Figure 4 Comparison of test performances of different electrophoresis methods to assay PCR products |
MSAP-PCR引物均为随机扩增引物,不同引物在基因组结合的热点位置和扩增的片段均不相同,因此所选择引物的不同也意味着对处理样品的多态性和敏感性不同。通过对不同浓度Cd胁迫处理下各引物的多态性条带统计来分析引物特性,结果发现在本实验所选引物中有4条引物多态性丰富,并且在0.2 mg·L-1 Cd2+水平下便可检测到多态性差异;通过对多个引物综合统计,发现与对照相比多态性变化较为丰富,各处理间的多态性差异较为明显(表 2)。该结果说明此方法与其他全基因组甲基化检测方法相比同样有较高的敏感性和多态性,是一种快速、简便且经济的甲基化检测方法,可用来替代传统的MSAP法。
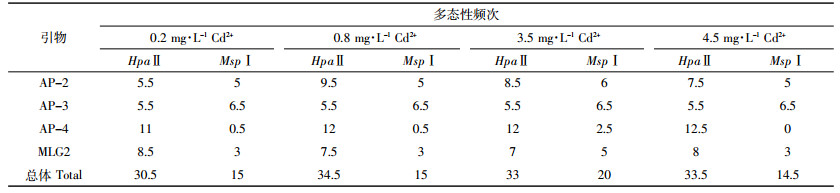 |
为了与MSAP-PCR方法对比,本文使用了近期本实验室的MSAP数据(处理浓度为0.25、1.0、3.0、5.0 mg·L-1 Cd2+),方法参照实验室之前发表文章[17],而且为了获得更准确的多态性结果,目前染色方法采用银染。传统MSAP方法由于酶切后需要连接接头,并进行预扩和选扩,造成其步骤繁琐,实验周期较长,虽然通过增加选择性引物可提高所得结果的多态性,却不宜作为污染早期诊断的快速检测手段。MSAP-PCR由于其在酶切后可直接PCR扩增,并且多态性条带的多少取决于引物的选择,该方法更适合用于污染早期示警和诊断以及作为环境污染研究的生物胁迫标记物。另外,由于该方法在0.2 mg·L-1 Cd2+水平便可检测到较好的多态性,其对Cd胁迫的敏感性与MSAP法相近,而且所得的甲基化变化趋势与MSAP相似,都呈倒“U”字型(图 5)。在传统MSAP法中,主要使用6%左右100%变性聚丙烯酰胺凝胶电泳,该方法在电泳时如若上样少会导致小片段条带染色不清(图 6B),上样多会造成大片段条带染色过深而无法分辨(图 6A),而优化过的MSAP-PCR法由于条带分散,即使上样略多也不会造成染色过重而无法读带的情况(图 4C),与传统方法相比上样量范围较宽、容易掌控,因此该方法可作为替代MSAP法的一种快速、有效的新方法应用于植物甲基化研究。

|
| 图 5 MSAP-PCR 法与 MSAP 法检测甲基化变化的结果比较 Figure 5 Comparison of methylation variation detected by MSAP-PCR and MSAP methods |
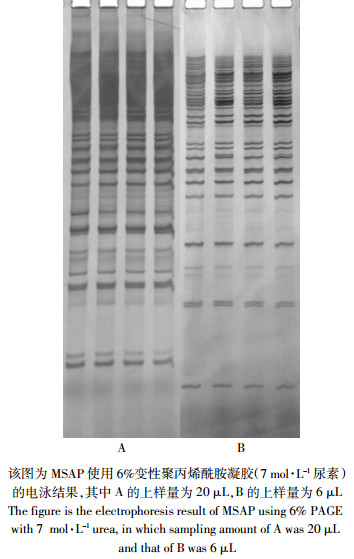
|
| 图 6 不同上样量的变性聚丙烯酰胺凝胶电泳结果比较 Figure 6 Comparison of PAGE results with different sampling amounts |
随着环境污染的日趋严重,生物生命活动已受到直接的影响,其中长期暴露于外界环境下的植物更会受到各种各样的胁迫,并且在最初时植物并不适应这些胁迫,所以在植物适应之前,这些胁迫会对植物自身的营养生长及生殖生长产生直接的负面影响[10]。随着人们对该现象的逐渐重视,引来更多学者对生物胁迫的关注和研究,然而经过研究发现,环境胁迫不仅对生理生长产生影响,还可产生明显的遗传损伤,且经研究证明该遗传损伤是可遗传至后代的[18, 19, 20]。遗传损伤对植物的影响更为直接,也较为敏感,已被认为是良好的生物胁迫标记物[21, 22],同时DNA甲基化作为当前热门的表观遗传修饰之一,可参与胁迫调控并且具有高敏感性,更是作为植物胁迫标记物的首选[23, 24, 25]。随着分子生物学的发展,基因组甲基化的研究手段也愈加丰富,各种研究方法和技术都有各自的优势,但是若从胁迫标记物和污染早期诊断方面来考量,具有简便、敏感和经济等特点的技术则应是最佳的选择,MSAP-PCR技术也正是基于该原因被本研究所推荐。
在应用MSAP-PCR方法研究植物全基因组甲基化过程中,引物的选择对实验效果以及胁迫后植物基因组甲基化多态性影响极大。不同的引物在相同PCR条件下会产生差异很大的电泳条带图谱,其中条带分布较集中、片段长度过大或存在强扩增效率条带的图谱由于无法控制后期染色显色的时间,会造成无法均匀显色而导致无法读带,因而扩增出该类图谱的引物需舍弃。另外,不同引物对胁迫的敏感性也不尽相同,例如,在本研究中,AP-4和MLG2引物在0.2 mg·L-1 Cd2+浓度下便有较高的甲基化多态性变化,说明此两条引物对该类胁迫较敏感;而在对CpG和CHG位点的甲基化敏感性上,不同引物间也存在差异,如AP-2和AP-3在CHG位点的敏感性就远高于AP-4和MLG2。综上所述,在研究环境胁迫引物植物基因组甲基化变化时,可通过筛选引物和增加所用引物数量来获得实验所需的甲基化多态性,以及寻找对某种胁迫具有特异性和敏感性的生物胁迫标记物。
MSAP-PCR法与MSAP法原理相似,都应用内切酶酶切基因组DNA而产生的多态性图谱,该类方法对电泳方法的要求较严格,因为不同的电泳方法和显色手段会对实验结果的多态性产生较显著的影响。MSAP-PCR法最早来源于人类癌症研究,从他们的研究中发现其所使用的电泳方法并不统一[13, 14, 15],由于植物基因组甲基化状态与人类的差别较大,我们对多种电泳方法进行了验证和评价,发现在人类癌症的报道中拥有较好多态性的1.8% MetaPhor琼脂糖胶并不适用于植物研究。根据本文的结果发现该电泳结果由于为非变性胶,条带少及多态性较低,而且EB染色灵敏度较差并且受PCR效果影响较大,不适宜使用。使用传统100%变性聚丙烯酰胺凝胶电泳则因实验结果中条带过于密集不利于读带,而且对显色时间的控制要求严格,所以实际检测效果并不理想。本实验通过多次调节和比较凝胶及尿素浓度,最终发现使用4%聚丙烯酰胺凝胶(50%尿素)实验效果最为理想,条带分散且清晰,显色时间易掌控而且多态性较高,适合作为MSAP-PCR的电泳检测方法,并建议MSAP法也改用此电泳方法检测。
与MSAP法、高效液相色谱以及以Southern杂交和同位素标记为基础的方法相比,MSAP-PCR法的操作更加简单,实验周期更短且实验成本更加经济,并且通过增加和筛选引物也同样能达到上述方法的多态性和敏感性,而且经过本研究对引物的筛选和对实验条件的摸索及优化,使该方法在植物基因组甲基化研究中更加简单、便捷和有效,为植物抗逆胁迫研究和环境污染的早期诊断提供了一种新型表观遗传损伤检测手段和生物胁迫标记物。 4 结论
(1)本研究首次采用MSAP-PCR法应用于植物全基因组甲基化检测,与传统方法相比,该方法操作更加简单,实验周期缩短,实验成本也更加低廉,可用于快速检测植物基因组甲基化变化。
(2)该方法通过重新设计和筛选引物,获得不低于传统方法的甲基化多态性和胁迫敏感性,可作为植物抗逆胁迫研究和环境污染的早期诊断的一种新型表观遗传损伤检测手段和生物胁迫标记物。
(3)本研究在电泳方法上的优化和改良,弥补了传统MSAP方法中电泳后染色效果不佳的弊端,使MSAP-PCR方法在植物甲基化的研究中更加完善,实验效果更加理想。
| [1] | Bird A P. CpG-rich islands and the function of DNA methylation[J]. Nature, 1986, 321(6067):209-213. |
| [2] | Finnegan E J, Brettell R I, Dennis E S. The role of DNA methylation in the regulation of plant gene expression[J]. EXS, 1993, 64:218-261. |
| [3] | Xiong Z, Laird P W. COBRA:A sensitive and quantitative DNA methylation assay[J]. Nucleic Acids Res, 1997, 25(12):2532-2534. |
| [4] | Herman J G, Graff J R, Myohanen S, et al. Methylation-specific PCR:A novel PCR assay for methylation status of CpG islands[J]. Proc Natl Acad Sci U S A, 1996, 93(18):9821-9826. |
| [5] | Sadri R, Hornsby P J. Rapid analysis of DNA methylation using new restriction enzyme sites created by bisulfite modification[J]. Nucleic Acids Res, 1996, 24(24):5058-5059. |
| [6] | Cokus S J, Feng S, Zhang X, et al. Shotgun bisulphite sequencing of the Arabidopsis genome reveals DNA methylation patterning[J]. Nature, 2008, 452(7184):215-219. |
| [7] | Lister R, O'Malley R C, Tonti-Filippini J, et al. Highly integrated single-base resolution maps of the epigenome in Arabidopsis[J]. Cell, 2008, 133(3):523-536. |
| [8] | Reyna-Lopez G E, Simpson J, Ruiz-Herrera J. Differences in DNA methylation patterns are detectable during the dimorphic transition of fungi by amplification of restriction polymorphisms[J]. Mol Gen Genet, 1997, 253(6):703-710. |
| [9] | Johnston J W, Harding K, Bremner D H, et al. HPLC analysis of plant DNA methylation:A study of critical methodological factors[J]. Plant Physiol Biochem, 2005, 43(9):844-853. |
| [10] | Ou X F, Zhang Y H, Xu C M, et al. Transgenerational inheritance of modified DNA methylation patterns and enhanced tolerance induced by heavy metal stress in rice(Oryza sativa L.)[J]. PLoS One, 2012, 7(9):e41143. |
| [11] | Gonzalgo M L, Jones P A. Rapid quantitation of methylation differences at specific sites using methylation-sensitive single nucleotide primer extension(Ms-SNuPE)[J]. Nucleic Acids Res, 1997, 25(12):2529-2531. |
| [12] | Gonzalgo M L, Liang G, Spruck C H, et al. Identification and characterization of differentially methylated regions of genomic DNA by methylation-sensitive arbitrarily primed PCR[J]. Cancer Res, 1997, 57(4):594-599. |
| [13] | Huang T H, Laux D E, Hamlin B C, et al. Identification of DNA methylation markers for human breast carcinomas using the methylation-sensitive restriction fingerprinting technique[J]. Cancer Res, 1997, 57(6):1030-1034. |
| [14] | Aypar U, Morgan W F, Baulch J E. Radiation-induced epigenetic alterations after low and high LET irradiations[J]. Mutat Res, 2011, 707(1-2):24-33. |
| [15] | Kaup S, Grandjean V, Mukherjee R, et al. Radiation-induced genomic instability is associated with DNA methylation changes in cultured human keratinocytes[J]. Mutat Res, 2006, 597(1-2):87-97. |
| [16] | Pierron F, Baillon L, Sow M, et al. Effect of low-dose cadmium exposure on DNA methylation in the endangered European eel[J]. Environmental Science & Technology, 2014, 48(1):797-803. |
| [17] | 李照令,王鹤潼,陈瑞娟,等.运用MSAP研究镉胁迫对拟南芥幼苗基因甲基化的影响[J].农业环境科学学报, 2014, 33(1):28-36. LI Zhao-ling, WANG He-tong, CHEN Rui-juan, et al. Studying genomic methylation of Arabidopsis thaliana seedlings under cadmium stress using MSAP[J]. Journal of Agro-Environment Science, 2014, 33(1):28-36. |
| [18] | Molinier J, Ries G, Zipfel C, et al. Transgeneration memory of stress in plants[J]. Nature, 2006, 442(7106):1046-1049. |
| [19] | Boyko A, Kovalchuk I. Genome instability and epigenetic modification:Heritable responses to environmental stress?[J]. Curr Opin Plant Biol, 2011, 14(3):260-266. |
| [20] | Verhoeven K J, Jansen J J, van Dijk P J, et al. Stress-induced DNA methylation changes and their heritability in asexual dandelions[J]. New Phytol, 2010, 185(4):1108-1118. |
| [21] | Labra M, Di Fabio T, Grassi F, et al. AFLP analysis as biomarker of exposure to organic and inorganic genotoxic substances in plants[J]. Chemosphere, 2003, 52(7):1183-1188. |
| [22] | Da Silva F R, Kvitko K, Rohr P, et al. Genotoxic assessment in tobacco farmers at different crop times[J]. Science of the Total Environment, 2014, 490:334-341. |
| [23] | How Kit A, Nielsen H M, Tost J. DNA methylation based biomarkers:Practical considerations and applications[J]. Biochimie, 2012, 94(11):2314-2337. |
| [24] | Naydenov M, Baev V, Apostolova E, et al. High-temperature effect on genes engaged in DNA methylation and affected by DNA methylation in Arabidopsis[J]. Plant Physiol Biochem, 2015, 87:102-108. |
| [25] | Plitta B P, Michalak M, Bujarska-Borkowska B, et al. Effect of desiccation on the dynamics of genome-wide DNA methylation in orthodox seeds of Acer platanoides L.[J]. Plant Physiol Biochem, 2014, 85:71-77. |
 2015, Vol. 34
2015, Vol. 34