文章信息
- 马丹, 孙瑞, 权伟, 王莹莹
- MA Dan, SUN Rui, QUAN Wei, WANG Ying-ying
- 典型超微细菌降解邻苯二甲酸二丁酯的途径及水合酶的表达纯化研究
- Microbial degradation pathway of dibutylphthalate by a typical ultramicrobacterium and the expression and purification of 4-oxalomesaconate hydratase
- 农业环境科学学报, 2016, 35(10): 1992-1997
- Journal of Agro-Environment Science, 2016, 35(10): 1992-1997
- http://dx.doi.org/10.11654/jaes.2016-1010
文章历史
- 收稿日期: 2016-08-06
邻苯二甲酸酯PAEs是一种重要的内分泌干扰素水体环境污染物[1]。广泛用于塑料、化妆品、农药、橡胶制造、喷漆当中的添加剂,该类物质是难于降解的有机物[2-3]。超过60种邻苯二甲酸酯类化合物被生产和使用。美国环境保护署(United States Environmental Protection Agency),欧盟和中国国家环境监测中心(The European Union and China National Environmental Monitoring Center)均将其列为首要污染物[4]。邻苯二甲酸酯类(PAEs)对我国水体污染较严重[5-6],微生物降解是自然环境中PAEs完全矿化的主要过程,也是修复PAEs污染水体的重要方法[7]。革兰氏阳性菌和革兰氏阴性菌对其降解途径不同,一般来讲开环酶4, 5-双加氧酶一般存在于革兰氏阴性菌中,而开环酶3, 4-加氧酶大多数存在于革兰氏阳性菌中[8-9]。在PAEs的微生物好氧降解过程中,酯键的水解是一个共同的起始步骤,PAEs在微生物酯酶作用下水解形成邻苯二甲酸单酯,进而生成邻苯二甲酸。研究显示,革兰氏阴性菌和革兰氏阳性菌通常通过两条不同途径降解邻苯二甲酸,但最终都形成原儿茶酚[10-11]。原儿茶酚是许多芳香族化合物代谢途径中的重要中间代谢物,经间位或邻位开环后形成相应的有机酸。4-oxalomesaconate水合酶(OMH)是原儿茶酸间位裂解途径中的关键酶之一,以4-oxalomesaconate(OMA)为底物,催化其降解为4-carboxy-4-hydroxy-2-oxoadipate(CHA)。2000年Hara等人在Sphingomonas paucimobilis SYK-6菌株中纯化出OMH,由于OMA通常是4-羟基-3-甲氧基苯酸酯和丁香酸的中间代谢产物,在以上两个代谢途径中OMH也起到了重要作用,在其研究中并对OMH进行酶活及代谢途径分析的研究[10]。2007年Li等人从Pseudomonas ochraceae NGJ1菌株中克隆并纯化了OMH蛋白,研究了半胱氨酸残基在该酶催化中的作用,利用了定点突变的方法,将8个单独的半胱氨酸残基突变为丝氨酸残基[12]。经过一系列的降解反应形成CHA后,进而转化成丙酮酸、琥珀酸、草酰乙酸等进入三羧酸循环,最终转化为CO2和H2O,完成污染物的降解过程。
虽然PAEs的微生物降解受到了人们的广泛关注,但在寡营养条件下利用微生物尤其是超微细菌降解PAEs的机理研究还尚无报道。目前,有关超微细菌的研究多数局限于菌株的分离与培养方法。研究者对于这类细菌在污染环境中的作用还很不了解。超微细菌对污染环境修复机理的研究还基本处于空白阶段。为此,本文针对实验室前期分离的一株典型超微细菌菌株Curvibacter sp.strain PAE-UM[13]展开其降解邻苯二甲酸二甲酯途径,进行关键性降解酶的表达纯化的初步研究,以期通过蛋白三维结构的研究,揭示超微细菌降解PAEs的分子机理,为利用超微细菌修复邻苯二甲酸酯污染水体提供实验依据、奠定理论基础。
1 材料与方法 1.1 材料邻苯二甲酸二丁酯(DBP),购自梯希爱(上海)化成工业发展有限公司,纯度≥98.0%。AKTA纯化仪均购买自GE公司;蛋白结晶条件试剂盒购买自Hampton公司;限制性内切酶和T4连接酶购买自Thermo公司;大肠杆菌BL21(DE3)感受态购买自北京全式金生物技术有限公司。
1.2 方法 1.2.1 菌株生长及DBP检测寡营养培养基由依云水过膜(0.22 μm)后巴氏消毒,DBP为唯一碳源,降解初始浓度为4 mg·L-1,接菌量为1×103 cells·mL-1。30 ℃下摇床培养。用流式细胞仪(CyFlow Space)检测细菌生长情况,每次取样1 mL,加入10 μL SYBR Green I染料进行染色,振荡混匀后黑暗染色15 min。检测时绿色荧光在520(±20)nm处收集;红色荧光(FL3)在630(±20)nm处收集。所有数据都采用Flowmax软件进行分析。
采用高效液相色谱(Waters)测定DBP的浓度,色谱柱采用Agilent Eclipse XDB-C8(5 μm×4.6 mm×250 mm),柱温30 ℃,流动相配比为乙腈:水=0.8:0.2,流速为1.0 mL·min-1,检测波长为226 nm。利用气相色谱检测降解中间产物,样品经固相萃取后,采用安捷伦Thermo DSQⅡ气相色谱-质谱联用仪。气相色谱条件:色谱柱为HP-5(30 m×0.32 mm×0.25 μm)。升温程序:100 ℃,保持1 min,以8 ℃··min-1升至300 ℃,保持3 min,进样口温度:250 ℃,进样量:1 μL。
1.2.2 OMH的克隆和表达使用限制性内切酶EcoRI和XhoI将OMH全长基因从质粒模板上切下,将基因片段通过T4连接酶连入表达载体pET-28a中,将质粒转化到质粒扩增型的感受态细胞(trans5)中,进行质粒扩增,然后提取质粒(AXYGEN Plasmid Miniprep Kit质粒提取试剂盒)送公司测序,经测序正确后将质粒转化到表达型的感受态细胞BL21(DE3)中,进行蛋白的外源表达。于LB固体培养基(1.5%琼脂)平板上,挑取单克隆菌落,至5 mL LB液体培养基(卡那霉素浓度为100 mg·L-1),37 ℃、200 r·min-1,培养10 h。将长势良好的大肠杆菌转入1 L含100 mg·L-1卡那霉素的LB液体培养基中,37 ℃、200 r·min-1,培养至OD 600值达到0.6至0.8。低温冷却,加入诱导剂异丙基硫代半乳糖苷(IPTG)至终浓度为0.5 mmol·L-1。在16 ℃诱导培养16~18 h,将菌液5 000 r·min-1离心15 min,收集菌体沉淀。
1.2.3 OMH的纯化用Mcac0缓冲液(20 mmol·L-1 Tris,0.5 mmol·L-1 NaCl,100 mL·L-1甘油,pH 8.5)垂悬收集的菌体后进行超声破碎裂解细胞,裂解后的菌液在低温4 ℃进行高速离心,18 000 r·min-1离心40 min,上清液中包含有目的蛋白。取上清上样于Mcac0缓冲液预平衡好的Ni-NTA亲和层析柱上,挂柱洗脱掉非特异结合的杂蛋白后,将目的蛋白用Elution buffer(20 mmol·L-1 Tris,0.5 mmol·L-1 NaCl,100 mL·L-1甘油,300 mmol·L-1咪唑,pH 8.5)洗脱。使用低盐缓冲液A(50 mmol·L-1 Tris,pH 8.0)及高盐缓冲液B(50 mmol·L-1 Tris,pH 8.0),配制缓冲液C(20 mmol·L-1 Tris,150 mmol·L-1 NaCl,pH 8.0),因该蛋白在低盐缓冲液A条件下产生沉淀,故在亲和层析后只过分子筛。将目的蛋白用缓冲液C完全稀释并浓缩至500 μL以下,在缓冲液C(20 mmol·L-1 Tris, 150 mmol·L-1 NaCl,pH 8.0)的环境中注入分子筛Supdex200 10/300GL,根据分子大小的不同将目的蛋白与杂质蛋白分离开。根据A280紫外吸收监测来收集蛋白样品,使用SDS-PAGE检测目的蛋白纯度。
1.2.4 OMH晶体的筛选和优化将纯化后的目的蛋白浓缩至8 mg·mL-1和15 mg·mL-1,采用坐滴气相扩散法在20 ℃恒温室进行晶体生长条件初筛,尝试了所有本研究组常用的晶体生长试剂盒(Hampton Research公司的Crystal Screen Kit、Crystal Screen KitⅡ、Index、Salt Rx、PEG/ion1-2和Emerald生物系统公司的Wizard Ⅰ-Ⅳ)。定期用显微镜进行观察,约10 d后发现晶体,通过优化蛋白浓度、晶体生长条件和添加剂等方法,获得衍射能力好的蛋白质晶体,用于X射线衍射实验。
1.2.5 OMH蛋白晶体X射线衍射数据的收集和处理将晶体保存在低温液氮罐中,送至上海光源同步辐射(SSRF)BL-19U线站收集数据。收集的数据使用HKL2000软件[14]进行数据的处理。数据处理流程一般为指标化(Index)、积分(Integrate)和归一化(Scale)。
2 结果与讨论 2.1 PAE-UM菌株降解DBP效率及途径微生物降解是DBP降解的主要途径[7],已有多种可降解邻苯二甲酸酯的菌株被报道[8, 13, 15-16]。菌株Curvibacter sp.strain PAE-UM可利用DBP为唯一碳源,对DBP的降解率为94%(图 1)。菌株最大生长速率为0.237 2 h-1。已经完成了该菌株的全基因组测序,GenBank登录号为LKCX00000000[17],该菌为β-变形菌门丛毛单胞菌属(Curvibacter sp.),是革兰氏阴性菌株,在降解邻苯二甲酸酯过程中通过邻苯二甲酸4,5双加氧酶的作用氧化邻苯二甲酸生成顺式-4,5-二羟基-4,5-二氢邻苯二甲酸,然后脱氢生成4,5-二羟基邻苯二甲酸,后者再通过脱羧生成原儿茶酚。
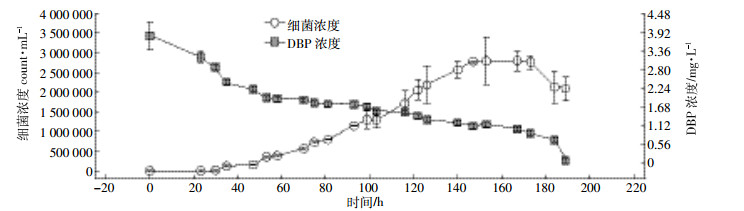
|
| 图 1 DBP浓度为4 mg·L-1时菌株的生长及DBP降解情况 Figure 1 Bacterial growth curve and DBP degradation |
已经分析出基因组中DBP降解的相关降解酶,并结合文献[3-4, 7]分析出该菌降解DBP的具体途径(图 2)。在Curvibacter sp.strain PAE-UM菌降解DBP过程中,原儿茶酸间位裂解,经原儿茶酸4, 5-双加氧酶,4-羧基-2-羟粘康酸-6-半醛脱氢酶(CHMS脱氢酶), 2-吡喃酮-4,6-二羧酸酯水解酶(PDC水解酶)生成OMA,最终进入TCA循环,转化为CO2和H2O。由ligJ基因编码的OMH具有催化OMA降解为4-carboxy-4-hydroxy-2-oxoadipate(CHA)的能力[18-20],该酶是降解邻苯二甲酸酯过程中的关键酶之一,也是芳香化合物降解过程中的重要降解酶[21]。本研究针对OMH进行了进一步的晶体学研究。

|
| 图 2 DBP降解途径及基因组中对应的降解酶 Figure 2 Degradation pathway of DBP and key enzymes involved |
OMH经过Ni-NTA亲和层析和凝胶过滤层析后,发现该蛋白在低盐缓冲液A条件下产生沉淀,说明该蛋白不耐受低盐条件,故在亲和层析后只过分子筛。经分子筛Superdex200 10/300 GL进行进一步纯化,图 3为分子筛纯化的A280吸收监测,显示为单一的洗脱峰,说明目的蛋白OMH在溶液状态下分子大小均一。用13% SDS-PAGE蛋白胶检测目的蛋白,得到纯度大于95%的OMH蛋白如图 4。

|
| 图 3 OMH经分子筛Superdex200 10/300 GL纯化的A280吸收峰 Figure 3 A280 absorption of gel filtration Superdex200 10/300 GL purification of OMA hydratase |
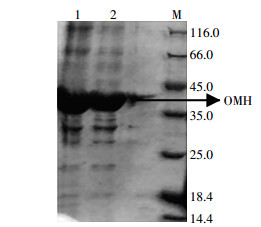
|
| 图 4 用13% SDS-PAGE蛋白胶检测目的蛋白 Figure 4 13%SDS-PAGE using to detect the target protein 泳道M为蛋白marker,单位为kDa,第1道及第2道为分子筛收集峰,目的蛋白OMH39.0 kDa用箭头标记 |
经过结晶条件的筛选和优化,在0.2 mol·L-1磷酸氢二铵,20%(m/V)聚乙二醇3350的条件下获得了衍射能力好的蛋白质晶体,如图 5所示。

|
| 图 5 OMH的晶体 Figure 5 Crystals of native OMH |
OMH晶体的X射线衍射实验在上海光源同步辐射(SSRF)BL-19U的同步辐射光源进行,选择0.5°的回摆角和1 s的曝光时间进行数据收集,共收集360张衍射图,如图 6,收集的衍射数据使用HKL2000软件进行处理,结果如表 1所示。

|
| 图 6 OMH晶体衍射图 Figure 6 Crystal diffraction pattern of native OMH |
在蛋白质结构数据库(Protein data bank, PDB)中,OMH的蛋白结构以Rhodopseudomonas palustris中N端结构基因目的蛋白Rpr66(PDB ID: 2GWG)作为分子置换模型,该模型与OMH氨基酸序列同源率达到80%,后续将使用PHENIX修正解析OMH的晶体结构。目前利用结构生物学手段解析PAEs关键降解酶的研究并不多,在蛋白结构数据库(http://www.rcsb.org/pdb)中发表的OMH的结构解析只有两个,分别是编号为3IJ6和2GWG蛋白结构,其中3IJ6的结构经X-ray衍射,结构解析为2.00 Å,该水合酶片段来自Lactobacillus acidophilus;2GWG的结构经X-ray衍射,结构解析为1.80 Å,该水合酶片段来自Rhodopseudomonas palustris。本文中超微细菌PAE-UM菌株中的OMH的蛋白晶体经X-ray衍射,分辨率为2.00 Å。该分辨率较高,从目前解析的OMH的数量来看,足以见得该酶在结构生物学的解析方面的研究还有待加强。
在寡营养条件下,利用微生物尤其是超微细菌降解PAEs的机理研究还鲜有报道。因此本研究对课题组分离出的PAE-UM菌株进行全基因组测序[17],通过全基因组数据及文献分析对比发现该菌降解PAEs的降解途径,并对其中关键酶利用蛋白结晶技术及分子生物学手段,解析能够降解污染物菌株中关键酶的三维结构,进一步阐明其降解DBP的分子机理及其代谢途径,为利用超微细菌修复邻苯二甲酸酯污染水体提供重要实验依据,奠定了理论基础。随着更多的对野生型和突变体的结构和生化研究,更多的降解酶结构被解析,将会有助于提高降解菌降解污染物的能力。
3 结论(1)菌株Curvibacter sp.strain PAE-UM可利用DBP为唯一碳源,DBP降解率为94%。
(2)经过全基因组分析,在Curvibacter sp.strain PAE-UM降解DBP过程中OMH催化OMA降解为4-carboxy-4-hydroxy-2-oxoadipate,该酶是DBP降解的关键酶之一。
(3)通过结构生物学手段,在大肠杆菌中表达得到OMH蛋白,经多种方法纯化后筛选获得了高质量蛋白质晶体。
(4)OMH的晶体经X-ray衍射、数据收集分析,得到了分辨率为2.00 Å的数据,为进一步解析其结构、阐明其作用机理奠定了基础。
致谢 感谢Mark Bartlam教授在晶体衍射数据分析方面的指导。| [1] | Teil M J, Tlili K, Blanchard M, et al. Polychlorinated biphenyls, polybrominated diphenyl ethers, and phthalates in roach from the Seine River basin(France): Impact of densely urbanized areas[J]. Archives of Environmental Contamination and Toxicology , 2014, 66 (1) : 41–57. DOI:10.1007/s00244-013-9955-8 |
| [2] | Magdouli S, Daghrir R, Brar S K, et al. Di 2-ethylhexylphtalate in the aquatic and terrestrial environment, a critical review[J]. Journal of Environmental Management , 2013, 127 (9) : 36–49. |
| [3] | Wang J L, Chen L J, Shi H C, et al. Microbial degradation of phthalic acid esters under anaerobic digestion of sludge[J]. Chemosphere , 2000, 41 (8) : 1245–1248. DOI:10.1016/S0045-6535(99)00552-4 |
| [4] | Fang C R, Yao J, Zheng Y G, et al. Dibutyl phthalate degradation by Enterobacter. sp. T5 isolated from municipal solid waste in landfill bioreactor[J]. International Biodeterioration & Biodegradation , 2010, 64 (6) : 442–446. |
| [5] | Zhang J, Liu L, Wang X, et al. Low-Level environmental phthalate exposure associates with urine metabolome alteration in a Chinese male cohort[J]. Environmental Science & Technology , 2016, 50 (11) : 5953–5960. |
| [6] | 李彬, 吴山, 梁金明, 等. 珠江三角洲典型区域农产品中邻苯二甲酸酯(PAEs)污染分布特征[J]. 环境科学 , 2016, 37 (1) : 317–324. LI Bin, WU Shan, LIANG Jin-ming, et al. Distribution characteristics and risk assessment of phthalic acid esters in agricultural products around the Pearl River Delta, South China[J]. Chinese Journal of Environmental Science , 2016, 37 (1) : 317–324. |
| [7] | Net S, Sempéré R, Delmont A, et al. Occurrence, fate, behavior and ecotoxicological state of phthalates in different environmental matrices[J]. Environmental Science & Technology , 2015, 49 (7) : 4019–4035. |
| [8] | Chang H K, Mohseni P, Zylstra G J. Characterization and regulation of the genes for a novel anthranilate 1, 2-Dioxygenase from Burkholderiacepacia DBO1[J]. Journal of Bacteriology , 2003, 185 (19) : 5871–5881. DOI:10.1128/JB.185.19.5871-5881.2003 |
| [9] | Eaton R W. Plasmid-encoded phthalate catabolic pathway in Arthrobacterkeyseri12B[J]. Journal of Bacteriology , 2001, 183 (12) : 3689–3703. DOI:10.1128/JB.183.12.3689-3703.2001 |
| [10] | Hara H, Masai E, Katayama Y, et al. The 4-oxalomesaconate hydratase gene, involved in the protocatechuate 4, 5-cleavage pathway, is essential to vanillate and syringate degradation in Sphingomonas paucimobilis SYK-6[J]. Journal of Bacteriology , 2000, 182 (24) : 6950–2657. DOI:10.1128/JB.182.24.6950-6957.2000 |
| [11] | Keyser P, Pujar B G, Eaton R W. Biodegradation of the phthalates and their esters by bacteria[J]. Environmental Health Perspectives , 1976, 18 (12) : 159–166. |
| [12] | Li S, Kimura M, Takashima T, et al. Role of cysteine residues in 4-oxalomesaconate hydratase from Pseudomonas ochraceae NGJ1[J]. Bioscience Biotechnology and Biochemistry , 2007, 71 (2) : 449–457. DOI:10.1271/bbb.60503 |
| [13] | 孙瑞, 韩东东, 马丹, 等. 寡营养条件下超微细菌对邻苯二甲酸酯的生物降解[J]. 安全与环境学报 , 2015, 15 (6) : 260–264. SUN Rui, HAN Dong-dong, MA Dan, et al. Biodegradation of phthalate esters by the ultramicro-bacterial consortium under the oligotrophic condition[J]. Journal of Safety and Environment , 2015, 15 (6) : 260–264. |
| [14] | Otwinowski Z, Minor W. Processing of X-ray diffraction data collected in oscillation mode[J]. Methods in Enzymology , 1997, 276 : 307–326. DOI:10.1016/S0076-6879(97)76066-X |
| [15] | 段星春, 易筱筠, 杨晓为, 等. 两株邻苯二甲酸二丁酯降解菌的分离鉴定及降解特性的研究[J]. 农业环境科学学报 , 2007, 26 (5) : 1937–194. DUAN Xing-chun, YI Xiao-yun, YANG Xiao-wei, et al. Isolation and characterization of two Di-n-butyl phthalate degrading bacteria[J]. Journal of Agro-Environment Science , 2007, 26 (5) : 1937–194. |
| [16] | 刘华, 沈新天, 孙丽娜, 等. 普通小球藻对邻苯二甲酸二丁酯的富集与降解研究[J]. 农业环境科学学报 , 2008, 27 (6) : 2391–2395. LIU Hua, SHEN Xin-tian, SUN Li-na, et al. Accumulation and biodegradation of dibutyl phthalate in chlorella vulgaris[J]. Journal of Agro-Environment Science , 2008, 27 (6) : 2391–2395. |
| [17] | Ma D, Hao Z, Sun R, et al. Genome sequence of a typical ultramicrobacterium, Curvibacter sp. Strain PAE-UM, capable of phthalate ester degradation[J]. Genome announcements , 2016, 4 (1) : 1–2. |
| [18] | Zhang S, Ma G, Liu Y, et al. Theoretical study of the hydrolysis mechanism of 2-pyrone-4, 6-dicarboxylate(PDC) catalyzed by LigI[J]. Journal of Molecular Graphics & Modelling , 2015, 61 (9) : 21–29. |
| [19] | Badger J. Structural analysis of a set of proteins resulting from a bacterial genomics project[J]. Proteins , 2005, 60 (4) : 787–796. DOI:10.1002/prot.20541 |
| [20] | Johnston J M, Arcus V L, Morton C J, et al. Crystal structure of a putative methyltransferase from Mycobacterium tuberculosis, misannotation of a genome clarified by protein structural analysis[J]. Journal of Bacteriology , 2003, 185 (14) : 4057–4065. DOI:10.1128/JB.185.14.4057-4065.2003 |
| [21] | Ni B, Zhang Y, Chen D W, et al. Assimilation of aromatic compounds by Comamonas testosteroni: Characterization and spreadability of protocatechuate 4, 5-cleavage pathway in bacteria[J]. Applied Microbiology and Biotechnology , 2013, 97 (13) : 6031–6041. DOI:10.1007/s00253-012-4402-8 |
 2016, Vol. 35
2016, Vol. 35