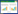文章信息
- 孙雪微, 许修宏, 孟庆欣, 成利军, 张文浩, 门梦琪, 许本姝, 孙瑜
- SUN Xue-wei, XU Xiu-hong, MENG Qing-xin, CHENG Li-jun, ZHANG Wen-hao, MEN Meng-qi, XU Ben-shu, SUN Yu
- 牛粪堆肥中氨氧化细菌群落结构及其与环境因子相关性研究
- Research of ammonia oxidizing bacterial community structure and its correlation with environmental factors in cow manure composting
- 农业环境科学学报, 2017, 36(1): 189-197
- Journal of Agro-Environment Science, 2017, 36(1): 189-197
- http://dx.doi.org/10.11654/jaes.2016-0960
文章历史
- 收稿日期: 2016-07-25
随着我国畜禽养殖业的大规模发展,畜禽粪便排放量也在逐年递增,给环境带了巨大的压力[1]。好氧堆肥是一种对牛粪等有机固体废弃物进行资源化、无害化和稳定化的经济高效的处理方法[2]。畜禽粪便经堆肥处理后转变成有机肥料,实现了资源再利用,但在堆肥过程中,往往伴随着严重的氮素损失,一般损失量为初始总氮量的17%~76%[3],既降低了堆肥产品的质量,也会释放臭气和氮氧化物等气体,对环境造成二次污染[4]。因此,控制堆肥过程氨氧化反应造成的氮素损失,已成为提高堆肥质量的重要途径之一。
硝化作用作为全球氮循环最重要的环节之一,以亚硝酸盐作为载体将氨盐氧化成硝酸盐[5]。由氨氧化细菌催化的氨氧化反应是硝化作用的限速步骤,在全球氮素生物化学循环中起着重要的作用[6]。氨氧化细菌属于专性化能自养的功能性微生物[7],其驱动的氨氧化反应是通过氨单加氧酶(Ammonia monooxygenase,AMO)催化,编码AMO 基因来完成的[8]。随着现代分子生物学技术的发展,对氨氧化细菌群落结构的研究也引起了国内外研究者的关注,如Yamada等[9]在氨含量较高的牛粪堆肥中检测到了氨氧化细菌群落的动态变化,邱珊莲等[10]在鸡粪堆肥中发现氨氧化细菌占据优势地位。但在堆肥领域中,氨氧化细菌的理论知识仍未完善,无法全面地对生产实践起到理论指导的作用,所以,将氨氧化细菌作为研究对象,研究其群落结构的动态演替及其与环境因子之间的相关性,对于揭示堆肥过程中氮素转移与变化规律具有深远的意义。
本研究采用变性梯度凝胶电泳(Denaturing gradient gel electrophoresis,DGGE)和Real-time PCR 技术检测牛粪堆肥过程中氨氧化细菌群落结构及数量的动态变化,同时采用冗余分析(Redundancy analysis,RDA)方法分析氨氧化细菌群落结构与堆肥环境因子之间的相关性,来了解堆肥过程中氮素的转移与转化规律,为减少氮素损失提供理论依据。
1 材料与方法 1.1 堆肥及样品采集堆肥材料由牛粪和水稻秸秆组成(表 1)。牛粪源于哈尔滨幸福乡奶牛养殖场,水稻秸秆源于哈尔滨市香坊区东北农业大学实验基地。将水稻秸秆截成5cm 左右,与牛粪按质量1:3.5 的比例均匀混合,并调整水分含量为65%~70%,置于规格为50 cm×50 cm×100 cm(长×宽×高)的堆肥模拟容器中,每隔30 min通风一次,采用上、中、下三点取样方式,均匀混合,每次取样后翻堆。堆肥过程共36 d,取样时间为:原始样品第1 d,升温阶段第2 d,高温阶段第3、4、7、11、14d,腐熟阶段第19、25、36 d,样品编号分别为D1、D2、D3、D4、D7、D11、D14、D19、D25 和D36。堆肥样品分为两份:一份在-4℃储存,用于理化指标的测定;另一份在-80℃储存,用于DNA 提取。
用LNI-TUT325 数字电子温度计测定堆体上、中、下三个位置的温度。堆肥样品的pH采用鲍士旦[11]的农化分析方法进行测定。水溶性铵态氮和硝态氮以2mol·L-1 氯化钾溶液浸提,采用连续流动分析仪(AA3,德国)测定其含量。堆肥潜在硝化能力(PAO)的测定采用He 等[12]的研究方法进行测定。称取10 g堆肥样品,置于100 mL 25 mg N·L-1 NH4Cl缓冲液中,25℃,150 r·min-1恒温摇床振荡24 h,每隔4 h取培养液一次,用连续流动分析仪测定NO2-N的含量。全碳和全氮分别采用重铬酸钾容量法和凯氏定氮法[11]测定。
1.3 DNA 提取和PCR 扩增取堆肥样品3~5 g,采用Liu等[13]的方法提取堆肥样品的总DNA,用1%的琼脂糖凝胶电泳检测后,用DNA 纯化试剂盒(Omega,美国)纯化回收DNA,再用1% 的琼脂糖凝胶电泳检测结果,于-20℃的冰箱保存备用。
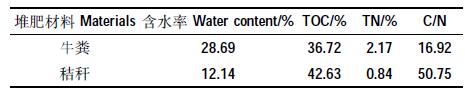
PCR 扩增采用氨氧化细菌的16S rRNA 特异性引物进行巢式PCR(Nested PCR)扩增,引物序列和反应程序见表 2。第一轮,反应体系为25 μL,包含400μmol·L-1 dNTP 2.5 μL,10×PCR Buffer(含MgCl2)2.5μL,引物对各0.2 μL(上游引物为CTO189fAB 与CTO189fC 按照2 :1 的比例混合,下游引物为CTO654r)[14],Taq 聚合酶0.3 μL,模板DNA 0.5 μL,添加无菌水调整终体积到25 μL。PCR 扩增产物大小为465 bp,将其稀释100 倍作为第二轮PCR 反应的DNA 模板。第二轮PCR 扩增反应条件同上,引物选用细菌特异性引物338f-GC 和518r[15],PCR 扩增产物大小为234 bp。最后用1% 的琼脂糖凝胶电泳检测PCR 扩增产物。
使用Bio-Rad DCodeTM system(Bio-Rad,美国)对氨氧化细菌的16S rRNA PCR 扩增片段进行DGGE分析,最适的变性梯度为30%~60%(100%的变性剂量中含有40% 的去离子甲酰胺和7 mol·L-1尿素),聚丙烯酰胺胶的浓度为8%[丙烯酰胺:双丙烯酰胺=37.5:1(m·V-1)]。电泳运行条件:在1×TAE 电泳缓冲液中,恒温60℃,恒压120 V,电泳12 h。电泳完成后,采用硝酸银银染法染色,拍照后将回收的优势DGGE条带置于灭菌的1.5 mL离心管中并编号,加30 μL无菌水,-4℃保存过夜,采用引物338f 和518r 进行PCR扩增后,将扩增产物送测序公司完成测序。
1.5 Real-time PCRReal-time PCR 反应采用SYBY Green 法,在CFX-96TM Real-Time PCR 仪上进行,采用的氨氧化细菌引物对为CTO189fAB、CTO189fC 和CTO654r,反应体系为20 μL,包含10 μL SYBR Premix Ex Taq域、1.6μL 稀释10 倍的DNA 模板以及上下游引物各0.8μL,其余以6.8 μL 无菌水补齐。Real-time 反应程序为:95℃30 s;95℃5 s,60℃30 s共40个循环。RealtimePCR标准曲线通过特异性引物PCR扩增DNA,扩增产物纯化后,连接到PMD-18T载体上,再转化到大肠杆菌DH5α感受态细胞中,通过蓝白斑试验,利用引物M13F(5'-CGCCAGGGTTTTCCCAGTCACGAC-3')和M13R(5'-AGCGGATAACAATTTCACACAGGA-3')筛选阳性克隆,提取质粒后,采用A260/A280 核酸蛋白测定仪测定其浓度,以8倍稀释梯度获取氨氧化细菌的标准曲线。标准曲线的扩增效率和相关系数分别为99.9%和0.94。
1.6 数据分析采用Quantity One 软件(Bio-Rad,美国)对DGGE图谱进行分析,采用UPGAMA 法进行聚类分析。采用多样性指数(H),均匀度指数(E)和丰富度(S)来分析氨氧化细菌16S rRNA 基因的多样性,计算公式如下:

式中:H 为Shannon 多样性指数;E 为均匀度指数;S为DGGE 胶中条带数目,即丰富度;Pi为第i 条带灰度占该样品总灰度的比率[16]。
通过Blast 程序将所得优势条带的序列与已知序列进行比对,采用Mega 6.0 软件构建氨氧化细菌16SrRNA 基因序列的系统发育树;采用Canoco 4.5 软件对堆肥样品中氨氧化细菌群落结构与环境因子进行冗余分析。
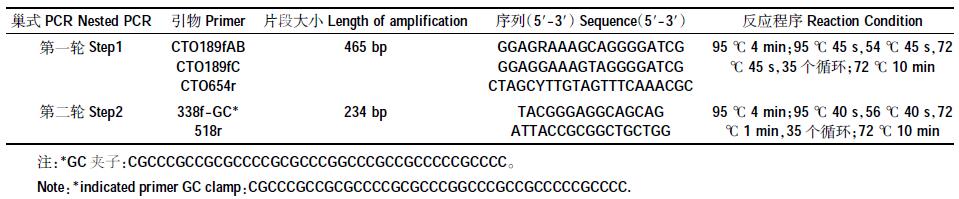
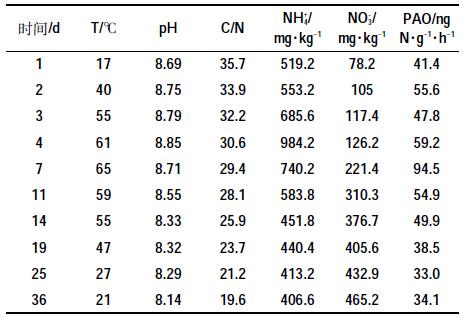
2 结果与讨论 2.1 堆肥过程中理化指标的变化理化指标的测定结果见表 3。随着堆肥的发酵,微生物快速分解有机质,释放大量的热量,使堆体温度急剧上升,在第7 d堆体温度达到最高温度,并且温度超过55℃的堆肥时间持续了11 d,杀死大量的病原菌,达到了基本堆肥的卫生标准[17],堆体温度在第14 d开始持续下降,且堆肥后期温度接近室温。在堆肥过程中,pH 变化范围为8.85~8.14,在第4 d达到最大值,一直处于弱碱性的状态。在堆肥前期,含氮有机物快速降解,铵态氮大量积累,从而使堆体的pH值不断上升,而在堆肥后期,铵态氮减少,堆体pH 值开始下降[18]。C/N是评价堆肥腐熟度的一种方法,在整个堆肥过程中C/N 持续下降,在堆肥结束时下降到19.6,据此可以认为堆肥已达到腐熟标准[19]。
在堆肥过程中,铵态氮含量呈现先升后降的趋势,因为在堆肥初期,微生物氨化作用及矿化作用加速了氮素的分解,所以堆肥前期铵态氮含量不断增多;而堆肥后期,由于部分铵态氮转化为硝态氮和氨气挥发等原因,导致铵态氮含量下降[20]。硝态氮含量呈现波动性上升的趋势,含量增加速度缓慢,可能是由于过多的铵态氮和过高的温度,在一定程度上抑制了微生物的活性,使硝态氮生成速度减缓[21]。PAO 的变化范围是33.0~94.5 ng N·g-1·h-1,在第7 d达到最大值。
2.2 样品总DNA的提取和PCR 的扩增采用Liu 等[13]的研究方法成功地提取了所用堆肥样品的总DNA,其长度约23 kb。将纯化后的DNA作为进行巢式PCR 第一轮的模板,所有样品均获得了特异性扩增产物,其大小为465 bp 左右,将其稀释100 倍作为第二轮PCR 的DNA 模板,均得到了234bp 左右的特异性片段。
2.3 氨氧化细菌丰度的分析采用Real-time PCR 技术对堆肥样品进行氨氧化细菌16S rRNA 基因的定量分析,获得的氨氧化细菌基因丰度如图 1所示。在整个堆肥过程中,氨氧化细菌基因丰度较低,基因拷贝数的变化范围为1.62×106~3.8×107 copies·g-1,在堆肥前期氨氧化细菌的基因拷贝数持续下降,第14 d达到最大值。氨氧化细菌基因丰度基本上都处于同一数量级,波动范围并不是特别大。这与Lu等[22]的研究结果一致,在淡水水产养殖池塘中氨氧化细菌的基因丰度较低。随着堆肥进入发酵高温期,氨氧化细菌基因拷贝数明显降低。这可能与温度影响有关,温度过高,抑制了多数微生物的生长繁殖,仅一些耐热微生物在快速生长,所以高温期的氨氧化细菌相对较少[23],而在堆肥第14 d,堆体温度达到55℃,随后进入降温阶段,有利于微生物快速生长,所以氨氧化细菌的基因拷贝数明显增多。在本研究中高铵离子浓度可能是导致氨氧化细菌丰度低的另一个关键原因,随着铵态氮的逐渐积累,在一定程度上抑制了氨氧化细菌的生长,在堆肥后期受铵态氮浓度的影响,氨氧化细菌的生长减缓,导致基因丰度下降[24]。这与Wuchter 等[25]的研究结果一致,在海水中氨氧化微生物的基因丰度与铵态氮的浓度显著负相关。

|
| 图 1 堆肥过程中氨氧化细菌的基因拷贝数 Figure 1 amoA gene copy numbers per gram compost sample during composting |
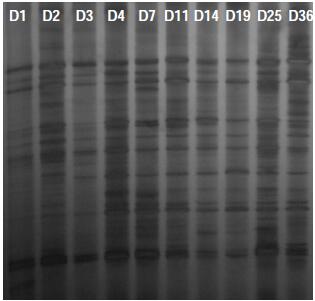
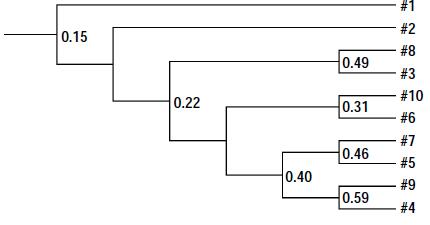
采用氨氧化细菌16S rRNA 基因特异性引物对扩增产物进行DGGE,得到的氨氧化细菌DGGE 图谱如图 2所示。图谱中各个泳道的条带有较大的差异,说明堆肥各时期氨氧化细菌的群落变化较剧烈。利用Quantity One 软件进行数字化处理,聚类分析(图 3)结果也显示了氨氧化细菌在堆肥过程中群落结构存在明显的差异,泳道3 和8、6 和10、5 和7、4 和9 彼此聚合,相似性系数分别达49%、31%、46%和59%。这说明各泳道群落组成相似性较高,而泳道1 和2 并未与其他泳道聚为一类,表明进入高温期后氨氧化细菌的菌群组成发生了剧烈的变化。
|
| 图 2 牛粪堆肥中氨氧化细菌DGGE 图谱 Figure 2 DGGE profile of ammonia oxidizing bacteria in cow manure composting |
|
| 图 3 氨氧化细菌DGGE 图谱聚类分析 Figure 3 Cluster analysis of DGGE profile of ammonia oxidizing bacteria in cow manure composting |
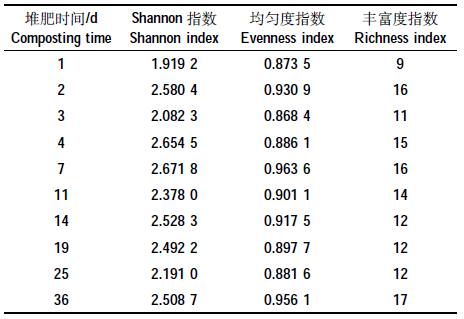
通过Quantity One 软件计算出氨氧化细菌群落的多样性指数、均匀度指数和丰富度指数(表 4),结果显示,均匀度指数变化不大,但多样性指数和丰富度指数出现了明显的改变。随着堆肥的进行,氨氧化细菌的多样性指数呈现先升后降的趋势,在第7 d达到最大值2.671 8。这可能是因为在堆肥前期,铵态氮等硝化作用底物含量充足,营养物质丰富,利于氨氧化细菌的生长,导致多样性指数上升;而随着堆肥的不断发酵、温度的升高和铵态氮浓度的下降,部分氨氧化细菌的生长受到了影响而被淘汰,使多样性指数逐渐减小[26]。
 |
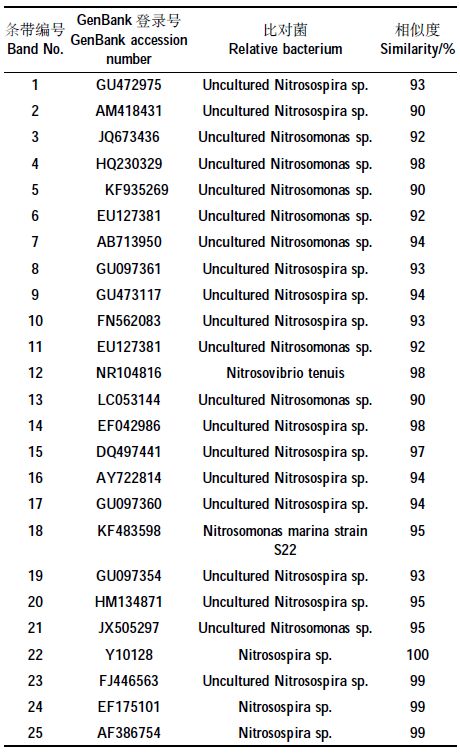
氨氧化细菌DGGE 图谱优势条带切胶测序比对结果见表 5。序列比对结果表明,大多数序列与不可培养的亚硝化螺菌属Uncultured Nitrosospira sp. 和不可培养的亚硝化单胞菌属Uncultured Nitrosomonassp. 这两个属的细菌相似度较高,在90%~100%之间,其中,与Uncultured Nitrosospira sp.相似度高的氨氧化细菌为优势菌,约占60%,而Uncultured Nitrosomonassp.菌群约占40%。由所获得优势菌序列构建的氨氧化细菌系统发育树见图 4。由图可知,氨氧化细菌序列被划分成Uncultured Nitrosospira sp. 和UnculturedNitrosomonas sp.两大分支,其中Uncultured Nitrosospi-ra sp. 分支占优势。
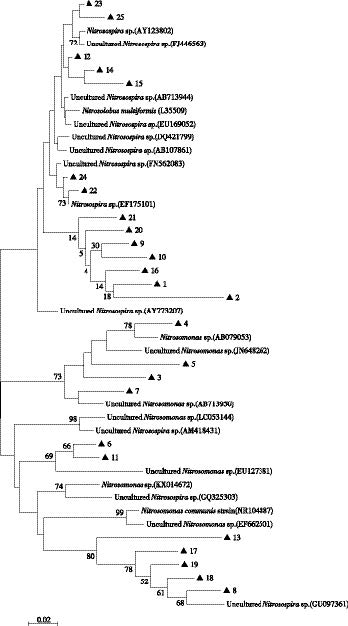
|
| 图 4 牛粪堆肥中氨氧化细菌16S rRNA基因的系统发育树 Figure 4 Phylogenetic tree of ammonia oxidizing bacteria based on 16S rRNA gene in cow manure composting |
通过氨氧化细菌16S rRNA 基因系统发育树分析可知,在牛粪堆肥过程中氨氧化细菌都隶属于茁-变形菌纲,多数序列与Uncultured Nitrosospira sp. 和Uncultured Nitrosomonas sp.的相似度较高。这与虞泳等[27]研究结果一致,在农业好氧堆肥中也发现了Ni原trosomonas sp.和Nitrosospira sp.优势菌群。由此推测,在堆肥过程中氨氧化细菌主要是Nitrosomonas sp.和Nitrosospira sp.菌。在本研究中,以Nitrosospira sp.属的氨氧化细菌为优势菌群,可能是由于高浓度的铵态氮含量所引起的。Enwall 等[28]的研究结果表明,在铵态氮浓度较高的条件下,Nitrosospira sp. 菌生长更为旺盛。
2.6 氨氧化细菌群落结构与堆肥环境因子的冗余分析采用冗余分析方法分析堆肥过程中氨氧化细菌群落结构变化与理化因子的相关性,结果见图 5。二维排序轴的前两个排序轴分别解释了种群与环境的积累变化率为79.2%和24.4%。在二维排序图中,箭头连线的长短表示群落与该环境因子之间相关系数的大小,箭头连线与排序轴的夹角表示该环境因子与排序轴相关性的大小,夹角越大,其相关性越小[29]。硝态氮和PAO 箭头连线较长,铵态氮、C/N、温度和pH的箭头连线较短,所以根据箭头连线的长度可知,环境因子对氨氧化细菌群落结构影响的相关性大小顺序为PAO>硝态氮>铵态氮>C/N>温度>pH。从图中也可以看到,随着堆肥的进行,氨氧化细菌的群落结构变化显著,在升温阶段(day2 和day3),氨氧化细菌群落结构较为相似,而在高温阶段与降温阶段,氨氧化细菌群落结构剧烈变化。
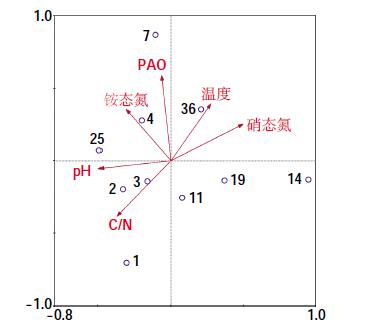
|
| 图 5 氨氧化细菌群落结构的冗余分析(RDA) Figure 5 Redundancy analysis(RDA)of ammonia oxidizing bacterial community structure during composting |
通过冗余分析可知,温度、PAO、铵态氮、硝态氮和pH 对氨氧化细菌群落结构的影响较大,基于手动选择的冗余分析表明温度和铵态氮的影响较为显著(P<0.05),这与Zhang 等[30]研究结果相似,即环境因子显著影响着氨氧化细菌的群落结构变化。温度能够影响氨氧化细菌的生长速度、酶反应动力学以及化合物的溶解度等[31],会对氨氧化细菌群落结构产生明显的影响。在本研究的堆肥体系中,随着温度的升高,氨氧化细菌的群落结构发生了明显的变化,表明氨氧化细菌对温度的适应情况各不相同,仅部分耐高温氨氧化细菌存活。这与Yamamoto等[32]的研究结果一致,即在堆肥高温期氨氧化细菌是唯一的耐热硝化细菌。
铵态氮作为氨氧化反应的直接底物,其浓度与氨氧化细菌的数量与种类有直接关系[33]。宋亚娜等[34]的研究指出,提高铵浓度可以促进间作土氨氧化细菌的生长,对氨氧化细菌的群落结构和多样性产生影响。在本研究中,堆肥前期铵态氮不断积累,营养物质丰富,氨氧化细菌生长旺盛,而在堆肥后期,由于铵态氮浓度逐渐下降,氨氧化细菌因底物浓度减小而衰退,表明铵态氮对氨氧化细菌的群落结构的变化有十分显著的影响。
3 结论(1) 堆肥样品氨氧化细菌16S rRNA 基因丰度的变化范围是1.62×106~3.80×107 copies·g-1,在第14 d氨氧化细菌的基因拷贝数达到最大值。
(2) 不同时期堆肥样品氨氧化细菌多样性指数存在明显的差异,随温度达到最高值,氨氧化细菌的多样性指数也达到最大。
(3) 通过序列比对和系统发育分析可知,氨氧化细菌均隶属于茁-变形菌纲,多数序列与UnculturedNitrosospira sp. 和Uncultured Nitrosomonas sp. 属的细菌相似度较高,与Uncultured Nitrosospira sp.属相似度高的氨氧化细菌为优势菌。
(4) 冗余分析表明,堆肥环境因子对氨氧化细菌群落结构的影响显著,其相关性大小为PAO>硝态氮>铵态氮>C/N>温度>pH,其中温度和铵态氮对氨氧化细菌群落结构的影响更为显著。
| [1] | 朱兆良, DavidN, 孙波. 中国农业面源污染控制对策[M]. 北京: 中国环境科学出版社, 2006. ZHU Zhao-liang, David N, SUN Bo. Agricultural non-point source pollution control measures in China[M]. Beijing: China Environmental Science Press, 2006. |
| [2] | Wei Y S, Fan Y B, Wang M J, et al. Composting and compost application in China[J]. Resources Conservation & Recycling, 2000, 30(4) : 277–300. |
| [3] | Barrington S C D, Trigui M, Knight W. Effect of carbon source on compost nitrogen and carbon losses[J]. Bioresource Technology, 2002, 83(3) : 189–194. DOI:10.1016/S0960-8524(01)00229-2 |
| [4] | 殷震, 刘景华. 动物病毒学[M]. 北京: 科学出版社, 1997. YIN Zhen, LIU Jing-hua. Animal virology[M]. Beijing: Science Press, 1997. |
| [5] | Könneke M, Bernhard A E, Torre J R D L, et al. Isolation of an autotrophic ammonia-oxidizing marine archaeon[J]. Nature, 2005, 437(7058) : 543–546. DOI:10.1038/nature03911 |
| [6] | Lullo L D, Addesse R, Comegna C, et al. Influence of effluent irrigation on community composition and function of ammonia-oxidizing bacteria in soil[J]. Applied & Environmental Microbiology, 2001, 67(8) : 3426–3433. |
| [7] | Teske A, Alm E, Regan J M, et al. Evolutionary relationships among ammonia-and nitrite-oxidizing bacteria[J]. Journal of Bacteriology, 1994, 176(21) : 6623–6630. DOI:10.1128/jb.176.21.6623-6630.1994 |
| [8] | Zeng J, Zhao D Y, Huang R, et al. Abundance and community composition of ammonia-oxidizing archaea and bacteria in two different zones of Lake Taihu[J]. Canadian Journal of Microbiology, 2012, 58(8) : 1018–1026. DOI:10.1139/w2012-078 |
| [9] | Yamada T, Araki S, Ikeda-Ohtsubo W, et al. Community structure and population dynamics of ammonia oxidizers in composting processes of ammonia-rich livestock waste[J]. Systematic & Applied Microbiology, 2013, 36(5) : 359–367. |
| [10] | 邱珊莲, 张少平, 翁伯琦, 等. 鸡粪菌渣好氧堆肥过程中氨氧化古菌及氨氧化细菌群落的动态变化[J]. 农业环境科学学报, 2016, 35(3) : 573–583. QIU Shan-lian, ZHANG Shao-ping, WENG Bo-qi, et al. Dynamics of ammonia-oxidizing archaea and ammonia-oxidizing bacteria during composting of chicken manure and mushroom cultural waste[J]. Journal of Agro-Environment Science, 2016, 35(3) : 573–583. |
| [11] | 鲍士旦. 土壤农化分析[M]. 北京: 中国农业出版社, 2008: 39-89. BAO Shi-dan. Soil and agriculture chemistry analysis[M]. Beijing: China Agriculture Press, 2008: 39-89. |
| [12] | He J Z, Shen J P, Zhang L M, et al. Quantitative analyses of the abundance and composition of ammonia-oxidizing bacteria and ammonia-oxidizing archaea of a Chinese upland red soil under long-term fertilization practices[J]. Environmental Microbiology, 2007, 9(9) : 2364–2374. DOI:10.1111/emi.2007.9.issue-9 |
| [13] | Liu J, Xu X H, Li H T, et al. Effect of microbiological inocula on chemical and physical properties and microbial community of cow manure compost[J]. Biomass & Bioenergy, 2011, 35(8) : 3433–3439. |
| [14] | Innerebner G, Knapp B, Vasara T, et al. Traceability of ammonia-oxidizing bacteria in compost-treated soils[J]. Soil Biology & Biochemistry, 2006, 38(5) : 1092–1100. |
| [15] | Li A J, Yang S F, Li X Y, et al. Microbial population dynamics during aerobic sludge granulation at different organic loading rates[J]. Water Research, 2008, 42(13) : 3552–3560. DOI:10.1016/j.watres.2008.05.005 |
| [16] | Yang Q, Zhang J Z, Wang S, et al. Bacterial community structure and diversity in a black soil as affected by long-term fertilization[J]. Pedosphere, 2008, 18(5) : 582–592. DOI:10.1016/S1002-0160(08)60052-1 |
| [17] | 唐珠. 猪粪堆肥的理化特征及腐熟度评价研究[D]. 南京农业大学, 2011. TANG Zhu. Characterization and identification of maturity of pig manure composts[D]. Nanjing Agricultural University, 2011. |
| [18] | 张亚宁. 堆肥腐熟度快速测定指标和方法的建立[D]. 北京:中国农业大学, 2004. ZHANG Ya-ning. Studies on the foundation of simple indexes and mensuration of compost maturity[D]. Beijing:China Agriculture University, 2004. |
| [19] | 廖新俤, 吴银宝, 汪植三, 等. 堆体大小对猪粪堆肥影响和袋装堆肥的研究[J]. 农业工程学报, 2003, 19(4) : 287–290. LIAO Xin-di, WU Yin-bao, WANG Zhi-san, et al. Effects of pile size and packaging on swine manure composting[J]. Transactions of the Chinese Society of Agricultural Engineering, 2003, 19(4) : 287–290. |
| [20] | 徐大勇, 黄为一. 人工接种堆肥和自然堆肥微生物区系与分子多态性的变化[J]. 生态与农村环境学报, 2006, 22(1) : 29–33. XU Da-yong, HUANG Wei-yi. Variation of microflora and molecular polymorphism in inoculated and natural composts[J]. Journal of Ecology and Rural Environment, 2006, 22(1) : 29–33. |
| [21] | 李海波, 廉静, 李敏, 等. 反硝化菌株GW1的筛选及特性研究[J]. 河北科技大学学报, 2012, 33(2) : 184–189. LI Hai-bo, LIAN Jing, LI Min, et al. Isolation and characteristics of denitrifying bacterium strain GW1[J]. Journal of Hebei University of Science and Technology, 2012, 33(2) : 184–189. |
| [22] | Lu S M, Liao M J, Xie C X, et al. Seasonal dynamics of ammonia-oxidizing microorganisms in freshwater aquaculture ponds[J]. Annals of Microbiology, 2014, 65(2) : 651–657. |
| [23] | 诸葛健, 李华钟. 微生物学[M]. 北京: 科学出版社, 2009. ZHU Ge-jian, LI Hua-zhong. Microbiology[M]. Beijing: Science Press, 2009. |
| [24] | 董莲华, 杨金水, 袁红莉. 氨氧化细菌的分子生态学研究进展[J]. 应用生态学报, 2008, 19(6) : 1381–1388. DONG Lian-hua, YANG Jin-shui, YUAN Hong-li. Research advances in molecular ecology of ammonia oxidizing bacteria[J]. Chinese Journal of Applied Ecology, 2008, 19(6) : 1381–1388. |
| [25] | Wuchter C, Abbas B, Coolen M J, et al. Archaeal nitrification in the ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(33) : 12317–12322. DOI:10.1073/pnas.0600756103 |
| [26] | 杜雪晴, 廖新俤, 吴银宝, 等. 有机废弃物好氧堆肥系统中氨氧化微生物的研究进展[J]. 家畜生态学报, 2014(9) : 1–7. DU Xue-qing, LIAO Xin-di, WU Xin-bao, et al. Advances of ammonia oxidizing microorganisms in organic waste aerobic composting system[J]. Acta Ecologae Animalis Domastici, 2014(9) : 1–7. |
| [27] | 虞泳, 曾光明, 陈耀宁, 等. 农业废物好氧堆肥中氨氧化细菌的群落结构[J]. 环境科学, 2011, 32(10) : 3067–3072. YU Yong, ZENG Guang-ming, CHEN Yao-ning, et al. Succession of ammonia-oxidizing bacteria community structure during agricultural waste composting[J]. Chinese Journal of Environment Science, 2011, 32(10) : 3067–3072. |
| [28] | Enwall K, Nyberg K, Bertilsson S, et al. Long-term impact of fertilization on activity and composition of bacterial communities and metabolic guilds in agricultural soil[J]. Soil Biology & Biochemistry, 2007, 39(1) : 106–115. |
| [29] | Valentín-Vargas A, Root R A, Neilson J W, et al. Environmental factors influencing the structural dynamics of soil microbial communities during assisted phytostabilization of acid-generating mine tailings:A mesocosm experiment[J]. Science of the Total Environment, 2014, 500/501 : 314–324. DOI:10.1016/j.scitotenv.2014.08.107 |
| [30] | Zhang J C, Zeng G M, Chen Y N, et al. Effects of physico-chemical parameters on the bacterial and fungal communities during agricultural waste composting[J]. Bioresource Technology, 2011, 102(3) : 2950–2956. DOI:10.1016/j.biortech.2010.11.089 |
| [31] | 曾光明, 黄国和, 袁兴中, 等. 堆肥环境生物与控制[M]. 北京: 科学出版社, 2006. ZENG Guang-ming, HUANG Guo-he, YUAN Xing-zhong, et al. Compost environmental biology and control[M]. Beijing: Science Press, 2006. |
| [32] | Yamamoto N, Oishi R, Suyama Y, et al. Ammonia-oxidizing bacteria rather than ammonia-oxidizing archaea were widely distributed in animal manure composts from field-scale facilities[J]. Microbes and Environments, 2012, 27 : 519–524. DOI:10.1264/jsme2.ME12053 |
| [33] | Wang S Y, Wang Y, Feng X J, et al. Quantitative analyses of ammonia-oxidizing archaea and bacteria in the sediments of four nitrogen-rich wetlands in China[J]. Applied Microbiology & Biotechnology, 2011, 90(2) : 779–787. |
| [34] | 宋亚娜, MarschnerP, 张福锁, 等. 小麦/蚕豆、玉米/蚕豆和小麦/玉米间作对根际细菌群落结构的影响[J]. 生态学报, 2006, 26(7) : 2268–2274. SONG Ya-na, Marschner P, ZHANG Fu-suo, et al. Effect of intercropping on bacterial community composition in rhizoshpere of wheat(Triticum aestivum L.), maize(Zea mays L.) and faba bean(Vicia fata L.)[J]. Actaecologica Sinica, 2006, 26(7) : 2268–2274. |
 2017, Vol. 36
2017, Vol. 36