文章信息
- 王晶, 王蕊, 朱珂, 修伟明, 赵建宁, 杨殿林, 李刚, 田秀平
- WANG Jing, WANG Rui, ZHU Ke, XIU Wei-ming, ZHAO Jian-ning, YANG Dian-lin, LI Gang, TIAN Xiu-ping
- 转cry1Ab和epsps基因玉米C0030.3.5对土壤古菌丰度和多样性的影响
- Effects of transgenic maize containing cry1Ab and epsps gene C0030.3.5 on the abundance and diversity of soil archaea
- 农业环境科学学报, 2017, 36(10): 2048-2057
- Journal of Agro-Environment Science, 2017, 36(10): 2048-2057
- http://dx.doi.org/10.11654/jaes.2017-0446
文章历史
- 收稿日期: 2017-03-28
2. 天津农学院农学与资源环境学院, 天津 300384
2. College of Agronomy & Resources and Environment, Tianjin Agricultural University, Tianjin 300384, China
转基因作物的研究和应用,在全球范围内得到了蓬勃的发展,已成为“现代农业史上采用最为迅速的作物技术”。据ISAAA最新报告,1996—2016年的21年间,全球转基因作物累计种植面积达到空前的21亿hm2,市场价值达到158亿美元[1]。在转基因作物种植带来可观经济效益的同时,其引发的生态环境安全性问题受到了国际上的广泛关注[2-3]。
土壤微生物是土壤生态系统中物质能量循环的主要驱动力,能够表征土壤质量变化,决定着土壤的健康和稳定性[4]。转基因作物种植是否会对土壤生态系统产生影响,尤其是对土壤微生物的群落结构和功能产生影响,成为评价转基因作物生态安全性的一个重要指标[5]。20世纪80年代,古菌(Archaea)、细菌(Bacteria)和真核生物(Eukarya)的三域分类系统被提出[6]。然而,由于古菌一直被认为仅存在于极端条件下(如高盐、强碱或强酸环境),对古菌的研究非常有限。随着在陆地生态系统中发现大量泉古菌的存在,其在陆地生态系统中的地位引发了科学界的重视,研究发现古菌数量占微生物总数量的比例高达12%~38%[7],参与土壤元素的转化过程[8]。土壤古菌与细菌一样,在碳和氮的生物地球化学循环中发挥重要作用。产甲烷古菌在厌氧条件下能够将有机物甲烷化产生甲烷气体[9-11],氨氧化古菌则会参与硝化作用的氨氧化过程[12-13]。因此,充分认识古菌对环境变化的响应将为研究环境变化对生态系统的影响提供重要依据。
抗虫转基因玉米的培育,成为解决害虫危害难题的“绿色”途径,也为解决我国玉米大面积虫害而造成减产的难题提供了新的思路。具有复合性状的抗虫抗除草剂转基因玉米兼具抗虫性和除草剂耐受性,成为转基因玉米发展的新方向。转基因玉米种植后,其根系分泌物及外源基因表达产物进入土壤可能会引起土壤微生物以及酶活性等的改变,进而影响土壤营养元素转化过程[14]。因此,在转基因玉米推广前,应进行全面的生态环境安全性评价研究。目前转基因玉米生态环境安全性评价研究主要集中在对非靶标生物[15-16]以及土壤细菌群落[17-18]、土壤真菌多样性[19]和放线菌等的影响[20],基因漂流[21-22]和外源蛋白残留情况及动态变化[23]等方面,但对土壤古菌群落的影响研究不足。本研究采用荧光定量PCR和T-RFLP技术分析转cry1Ab和epsps基因玉米C0030.3.5种植后土壤古菌丰度和多样性的变化,探讨其对古菌丰度和群落结构的影响,丰富转基因玉米的环境风险性研究内容,为综合评价转基因玉米的环境安全性提供科学的理论依据。
1 材料与方法 1.1 试验地概况试验地点位于河北省唐山市玉田县陈家铺乡北京大北农生物技术有限公司试验基地,地处东经117°43′、北纬39°47′。该地区属北温带大陆性季风气候,年均降水量693.1 mm,年平均气温11.2 ℃,无霜期约为193 d,土壤为潮土。试验始于2015年,试验开始时,供试土壤0~20 cm土层含有机质13.09 g·kg-1,全氮2.80 g·kg-1,全磷0.53 g·kg-1,硝态氮15.96 mg·kg-1,铵态氮0.2 mg·kg-1,速效磷1.25 mg·kg-1,pH 8.28。
试验以转cry1Ab和epsps基因玉米C0030.3.5(TM)及受体玉米DBN318(PM)为研究对象,均由北京大北农生物技术有限公司提供。试验采用随机区组设计,转cry1Ab和epsps基因玉米C0030.3.5和受体玉米DBN318各设置3次重复,共6个小区,小区面积为10 m×15 m,小区间设有5 m的非转基因玉米隔离带,试验基地四周有围墙隔离,周边为设施蔬菜,无玉米种植。采取春播单季玉米的种植模式,试验前此样地的种植模式为小麦和玉米轮作,小麦和玉米均为普通非转基因品种。试验田施复合肥,其中基肥占60%,拔节期追肥占40%,试验期间的田间管理措施均按照当地常规操作进行,全生育期不施用农药。
1.2 土壤样品采集根际土(GJ)和非根际土(FGJ)样品分别采集于2015年6月28日(拔节期,J)、2015年8月10日(抽雄期,T)、2015年8月31日(乳熟期,M)和2015年9月28日(完熟期,R)。根际土采用十字交叉法采集,在每个小区随机选取5~10株玉米,采用抖落法收集紧紧附着在根上的土壤;非根际土采用W形多点采样法采集,在每个小区随机选取15个采样点,距离玉米主茎20 cm处利用土壤采样器采集0~15 cm的表层土壤。在每个生长时期,将同一处理小区同一采样区域的土壤充分混匀作为1个样品,装入自封袋中。所有土壤样品混匀后放入低温样品储藏箱中带回实验室。土壤样品过2 mm筛剔除植物残体、根系、石头及其他杂物后分为2份,一份保存于-70 ℃冰箱中用于分子实验分析,另一份用于土壤理化性质测定。
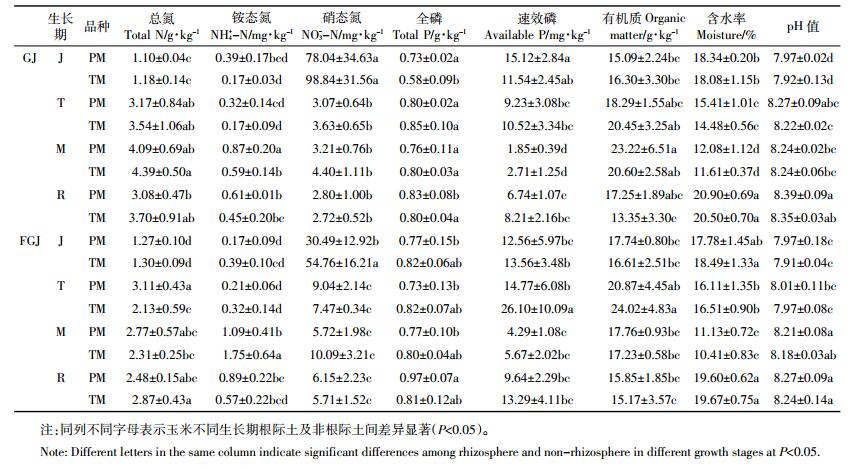
1.3 土壤基本理化性质测定采用连续流动分析仪测定土壤全氮、硝态氮和铵态氮含量。采用重铬酸钾容量法测定土壤有机质含量,硫酸-高氯酸消煮法测定土壤全磷含量,碳酸氢钠浸提-钼锑抗比色法测定土壤速效磷含量,采用烘干法测定土壤含水量(H2O),电位法(水土比2.5:1)测定土壤pH值,具体操作步骤参见文献[24]的方法。土壤基本理化性质的测定结果见表 1。
|
采用PowerSoilTM Total DNA Isolation试剂盒(Mo Bio Laboratories,Solana Beach,CA,USA)提取于-70 ℃冰箱中保存的土壤样品的总DNA。共提取48个土壤样品(2种玉米,2个土壤样品采集区域,4个生长时期,3次重复)的总DNA。提取步骤参考试剂盒说明书,最后用100 μL ddH2O洗脱。提取产物采用1.0%琼脂糖凝胶电泳分析完整性后保存于-70 ℃冰箱中待用。
1.5 qPCR分析土壤古菌16S rRNA基因的qPCR在Stratagene Mx3005P上进行,引物序列信息和PCR反应条件见表 2。25 μL的qPCR反应体系如下:1×SYBR Premix Ex TaqTM Buffer(TaKaRa),0.2 μmol·L-1上下游引物,1×ROX Reference dye Ⅱ(TaKaRa),0.01 g·L-1 T4 gene 32 protein(Roche),1.0 μL 20倍稀释的DNA模板,用灭菌水补足至总体积为25 μL。设置无模板对照(No Template Control,NTC),每个DNA样品和NTC各3次重复。所有qPCR反应均于72 ℃延伸时收集荧光信号,并绘制熔解曲线(60~95 ℃,每扫描1次温度增加0.5 ℃)。
标准曲线的制作:将采用ARC344F和ARC915R引物对扩增获得的片段进行克隆测序,用阳性克隆质粒以10倍梯度逐步稀释制备成的标准样品进行qPCR,绘制标准曲线。本研究中标准曲线的R2在0.998~1.000之间,扩增效率在90.0%~92.8%之间。
1.6 T-RFLP分析土壤古菌16S rRNA基因采用引物对Ar109F和Ar915R进行扩增,其中引物Ar915R的5′端采用6′-FAM荧光标记,引物序列信息和PCR反应条件见表 2。扩增体系如下:50 μL的PCR反应体系包括:1×Buffer(Promga,USA),1.5 mmol·L-1 MgCl2,0.4 mmol·L-1 dNTP(TaKaRa),0.4 μmol·L-1上下游引物,0.4 g·L-1 BSA(TaKaRa),1.25U的Go TaqTM Hot Start Polymerase(Promga,USA),DNA模板1.0 μL,用灭菌水补足至总体积为50 μL,每个样品各3次重复。PCR反应设置无模板对照(NTC)。每个样品和NTC各3次重复。将3次重复的扩增产物混合均匀经切胶纯化后,用限制性内切酶TaqⅠ(TaKaRa)65 ℃下酶切3 h,后加入10×loading buffer终止反应。酶切产物的T-RFLP分析由生工生物工程(上海)股份有限公司完成。
1.7 数据分析T-RFLP数据分析时将相差为±1 bp的片段归为同一T-RF[27],参照Lukow等[28]的方法计算相对丰度(Relative abundance,Ra),所有Ra大于1%的T-RFs为有义片段并用于T-RFLP分析,Ra大于10%的T-RFs为该样品的优势种群[29]。生物多样性用Shannon指数(H)和Evenness指数(EH)评价。计算公式如下:
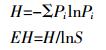
式中:H为Shannon指数;EH为Evenness指数;S为不同片段的种类数;Pi为第i条片段峰面积占该样品总峰面积的比率。
数据的显著性分析采用SPSS 22.0单因素方差分析(One-Way ANOVA)检验,并进行多重比较(Duncan法),2个处理间的数据比较采用独立样本的t检验(Independent-Samplet Test)。PCA分析采用SPSS 22.0软件进行,并通过Origin 9.0软件作图。土壤古菌群落结构与土壤环境因子的关联关系采用RDA方法(CANOCO for Windows 4.5.1)分析,并应用蒙特卡洛检验进行显著性分析(P < 0.05)。
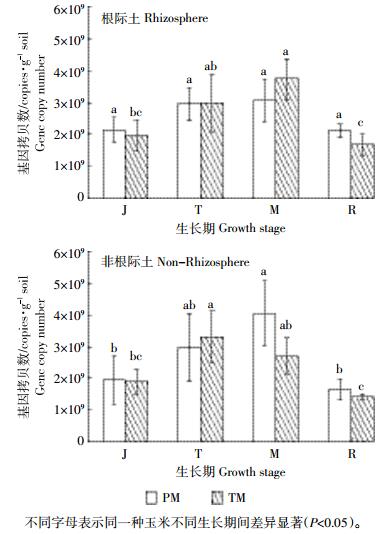
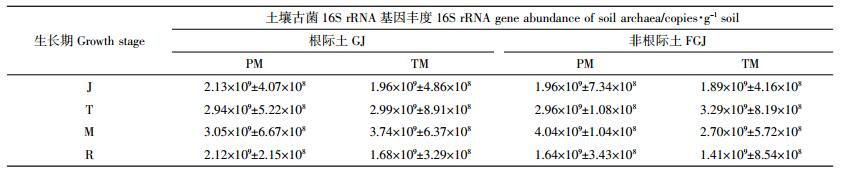
2 结果与分析 2.1 转cry1Ab和epsps基因玉米C0030.3.5对土壤古菌16S rRNA基因丰度的影响土壤古菌16S rRNA基因丰度的变化如图 1所示。PM和TM的根际土和非根际土古菌16S rRNA基因丰度随生长时期均呈现先升高后降低的变化趋势,PM根际土和非根际土古菌16S rRNA基因丰度范围分别为2.12×109~3.05×109 copies·g-1土和1.64×109~4.04×109 copies·g-1土,TM根际土和非根际土古菌16S rRNA基因丰度范围分别为1.68×109~3.74×109 copies·g-1土和1.41×109~3.29×109 copies·g-1土,最低值均出现在完熟期,而最高值除TM非根际土出现在抽雄期外,均出现在乳熟期,不同生长时期土壤古菌16S rRNA基因丰度间的显著性分析表明,PM根际土古菌16S rRNA基因丰度在4个生长期间无显著性差异(P>0.05);PM非根际土乳熟期古菌16S rRNA基因丰度显著高于拔节期和完熟期(P < 0.05),与抽雄期差异不显著(P>0.05),其他3个生长期间差异不显著(P>0.05);与PM非根际土相似,TM根际土古菌16S rRNA基因丰度乳熟期显著高于拔节期和完熟期(P < 0.05),与抽雄期差异不显著(P>0.05),但抽雄期显著高于完熟期(P < 0.05),而与拔节期无显著差异(P>0.05),拔节期与完熟期无显著差异(P>0.05)。TM非根际土古菌16S rRNA基因丰度抽雄期显著高于拔节期和完熟期(P < 0.05),与乳熟期差异不显著(P>0.05),乳熟期显著高于完熟期(P < 0.05),但与拔节期差异不显著(P>0.05),拔节期与完熟期间无显著差异(P>0.05)。
|
| 不同字母表示同一种玉米不同生长期间差异显著(P < 0.05)。 图 1 土壤古菌16S rRNA基因丰度变化 Figure 1 Abundance changes of soil archaeal 16S rRNA gene |
对比同一生长时期同一土壤样品采集区域PM和TM根际土古菌16S rRNA基因丰度(表 3),发现PM根际土在拔节期和完熟期高于TM,而在抽雄期和乳熟期相反,非根际土的分析结果与根际土基本一致(除乳熟期PM高于TM)。同时显著性分析结果表明,在4个生长时期,PM和TM根际土古菌16S rRNA基因丰度之间均无显著差异(P>0.05),非根际土的分析结果与根际土一致。同一种玉米同一生长时期根际土和非根际土古菌16S rRNA基因丰度间的对比结果表明,根际土和非根际土之间均无显著差异(P>0.05)。
|
以上研究结果表明,转cry1Ab和epsps基因玉米C0030.3.5与受体玉米DBN318土壤古菌16S rRNA基因丰度无显著差异,土壤古菌16S rRNA基因丰度变化主要受到玉米生长时期的影响,而受土壤样品采集区域的影响并不显著。
对土壤古菌16S rRNA基因丰度与土壤理化因子进行相关性分析(表 4),结果显示古菌16S rRNA基因丰度与土壤全氮、速效磷、有机质和含水率呈正相关,而与铵态氮、硝态氮和pH值呈负相关,但均未达到显著水平。

|
T-RFLP图谱(图 2)显示,不同生长时期土壤中共获得15种不同长度的T-RFs,其中89 bp(相对丰度19.34%~30.28%)和184 bp(相对丰度60.20%~74.47%)的片段所代表的物种为所有土壤共有的优势种群,它们的相对丰度显著高于其他T-RFs。PM根际土中,89 bp片段的相对丰度在抽雄期最低(19.58%),拔节期最高(25.00%);184 bp片段的相对丰度最低值出现在拔节期(67.21%),抽雄期最高(74.47%)。PM非根际土中,89 bp片段的相对丰度最低值和最高值同样出现在抽雄期(19.34%)和拔节期(27.72%);184 bp片段的相对丰度则在拔节期最低(63.67%)和抽雄期最高(71.80%)。TM根际土中,89 bp片段的相对丰度均高于20%,乳熟期最低(21.02%),拔节期最高(30.28%);184 bp片段的相对丰度均大于60%,拔节期最低(60.2%),乳熟期最高(73.63%)。TM非根际土89 bp片段的相对丰度拔节期(25.79%)最高,与根际土相同,但最低值出现在抽雄期(20.21%);184 bp片段则与根际土一致,拔节期最低(66.33%),乳熟期最高(71.92%)。
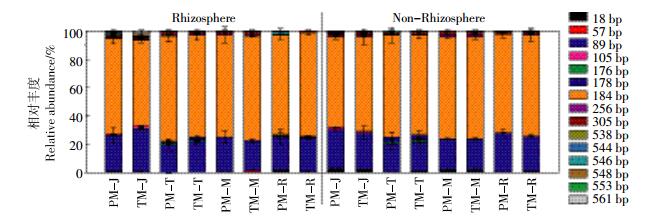
|
| 图 2 土壤古菌16S rRNA基因的限制性酶切片段和相对丰度 Figure 2 Average relative abundance of T-RFs of soil archaeal 16S rRNA gene |
研究同时发现,176 bp和256 bp片段在TM完熟期非根际土中存在,而在PM中缺失;178 bp片段在PM完熟期非根际土中存在,而在TM中缺失;538 bp片段在PM乳熟期根际土和拔节期非根际土中存在,在TM中缺失,而在TM乳熟期非根际土中存在,在PM中缺失;544 bp仅存在于PM拔节期根际土和非根际土中;546 bp片段在TM抽雄期根际土和拔节期非根际土中存在,相应时期的PM中缺失,而在PM完熟期根际土和非根际土中存在,TM中缺失;548 bp和561 bp片段存在于TM拔节期根际土和PM完熟期非根际土中,而相应的PM和TM中缺失;553 bp片段存在于PM拔节期和抽雄期根际土和TM拔节期及乳熟期非根际土中,相应的TM和PM中缺失。这些表现存在与缺失的非优势T-RFs的相对丰度均非常低,虽然它们表现为缺失,但并不说明在该样品中不存在,很可能是它们在各自样品中的含量低于所采用的T-RFLP方法的检出限。
同一生长时期和土壤样品采集区域2种玉米相同优势种群(89 bp和184 bp)的相对丰度间显著性分析表明,PM和TM间均无显著差异(P>0.05),TM和PM共有的非优势种群与优势种群分析结果一致。不同T-RFs所代表土壤古菌种群在各生长时期的土壤中所占比例发生变化,说明土壤古菌的群落结构发生一定的改变,但89 bp和184 bp 2种T-RFs所代表的优势群变化很小,表明土壤古菌整体的群落组成未发生显著变化,转cry1Ab和epsps基因玉米C0030.3.5与受体玉米DBN318间土壤古菌群落组成无显著差异。
2.3 转cry1Ab和epsps基因玉米C0030.3.5对土壤古菌16S rRNA基因多样性指数的影响由表 5可见,PM根际土古菌16S rRNA基因的Shannon指数表现为降低-升高-降低的变化趋势,抽雄期最低,乳熟期最高,而非根际土的Shannon指数表现为降低-升高的变化趋势,最低值同样出现在抽雄期,而最高值出现在完熟期;TM根际土和非根际土古菌16S rRNA基因的Shannon指数,均呈现先降低,后升高,再降低,抽雄期最低,乳熟期最高。PM和TM根际土古菌16S rRNA基因的Evenness指数变化均表现为先降低后逐渐升高的趋势,抽雄期最低,完熟期最高;而PM和TM非根际土的Evenness指数变化趋势均与根际土不同,且呈现降低-升高-降低的趋势,其中最小值均出现在抽雄期,PM最大值出现在乳熟期,TM最大值出现在拔节期。对同一生长时期和土壤样品采集区域PM和TM的Shannon指数间及Evenness指数间进行显著性分析,结果表明,PM和TM间均无显著差异(P>0.05),多样性指数的变化主要受到玉米生长时期的影响,受土壤样品采集区域影响较小。
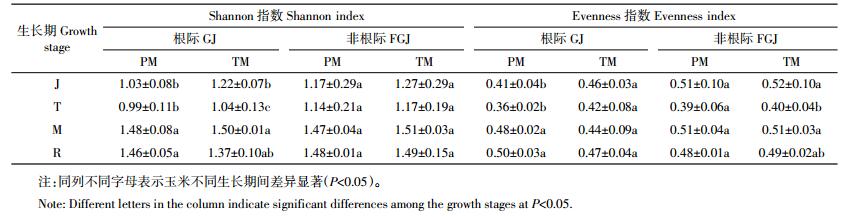
|
采用T-RFLP结果中相对丰度大于1%的T-RFs进行PCA分析,结果表明,不同生长时期,PM和TM根际土古菌群落组成在PC1和PC2轴上未发生明显分离(图 3A),非根际土古菌群落同样未发生明显分离(图 3B)。这说明转cry1Ab和epsps基因玉米C0030.3.5土壤古菌群落组成与受体玉米DBN318无显著差异。
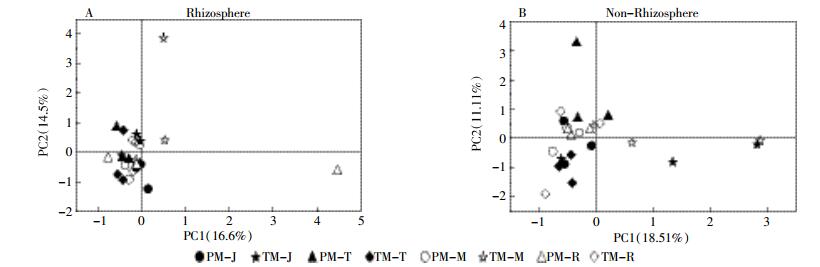
|
| 图 3 土壤古菌群落组成的PCA分析 Figure 3 PCA analysis of the composition of soil archaea |
对不同生长时期根际土古菌16S rRNA基因的T-RFLP结果进行RDA分析,结果显示(图 4A),前两轴可解释古菌16S rRNA基因多样性变化的90.3%。蒙特卡洛检验表明,土壤硝态氮含量(P=0.002)、pH值(P=0.002)、总氮含量(P=0.008)和总磷含量(P=0.014)与根际土古菌群落组成显著相关。不同生长时期非根际土古菌16S rRNA基因的RDA分析结果显示(图 4B),前两轴可解释古菌16S rRNA基因多样性变化的89.9%,土壤总氮含量(P=0.010)和硝态氮含量(P=0.014)与非根际土古菌群落组成显著相关。综合分析认为,土壤总氮和硝态氮是调控土壤古菌群落的关键因子。由图 4还可见,无论是根际土还是非根际土,PM和TM均未发生明显分离,表明转cry1Ab和epsps基因玉米C0030.3.5与受体玉米DBN318的土壤古菌群落组成无显著差异,与PCA分析结果一致。
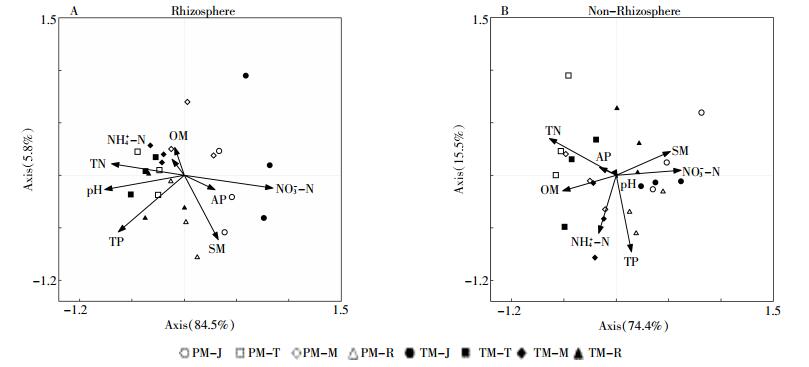
|
| 图 4 土壤古菌群落组成的RDA分析 Figure 4 RDA analysis of the composition of soil archaea |
随着转基因作物的大面积种植,其对土壤生态系统,尤其是对土壤微生物的影响受到广泛关注[30]。土壤微生物作为土壤生态系统的重要组成成分,会对作物种植产生敏感的响应,进而影响作物生长[31]。古菌作为一种可以适应极端环境如高温、强酸的生物,在整个生态系统中起着不可估量的作用[32-36]。本研究采用qPCR和T-RFLP方法分析了土壤古菌的丰度和多样性的变化,探讨了转cry1Ab和epsps基因玉米C0030.3.5种植对土壤古菌丰度和群落结构的影响。
qPCR分析结果表明,TM和PM根际土中古菌16S rRNA基因丰度均呈先升高后降低的趋势,说明玉米种植后,玉米的根系活动及其产生的根系分泌物,为微生物营造了更适应其生长和繁殖的微环境。在玉米生长前期(营养生长阶段),根系不断生长,根系活动增强,其分泌物和脱落物都不断增加,为古菌提供了可利用的碳源、氮源及其他营养物质,因此古菌的数量也随之增加,在乳熟期达到最大值,而在玉米生长后期,其根系生长缓慢,根系活动减弱,古菌可以利用的碳源和氮源也相应减少,其生长受到限制,数量也随之减少。在4个生长期中,同一时期同一土壤样品采集区域PM和TM根际土古菌16S rRNA基因丰度之间均无显著差异(P>0.05),并且同一玉米品种同一生长时期根际土和非根际土古菌16S rRNA基因丰度间同样均无显著差异(P>0.05)。
相关分析发现,古菌16S rRNA基因丰度与土壤总氮、速效磷、有机质和含水量呈正相关,而与铵态氮、硝态氮和pH值呈负相关,但均未达到显著水平。土壤有机质和总氮可以提供古菌生长需要的碳源和氮源。当土壤中总氮和有机质含量满足古菌生长所需时,古菌会大量繁殖,数量会随之增加。水分作为土壤中各种元素的溶质,可以通过溶解土壤中的营养物质影响古菌对养分的利用[37],同时,土壤水分会影响土壤的通气状况,进而影响微生物的呼吸作用,对土壤古菌数量产生影响。土壤古菌能够耐受极端环境,如强酸等,但极端条件会对古菌数量产生抑制作用,因此古菌数量与pH值表现为负相关关系;同时pH值又会影响土壤中铵态氮与硝态氮的转化过程[38],也造成速效氮素与古菌数量呈负相关关系。
T-RFLP数据表明,不同T-RFs所代表土壤古菌种群在不同生长时期的土壤中所占比例发生变化。虽然土壤古菌的群落结构在各生长时期间发生一定的改变,但89 bp和184 bp 2种T-RFs所代表的优势群的相对丰度变化很小,表明土壤古菌整体的群落结构组成未发生显著变化。土壤古菌多样性指数分析数据表明,TM土壤古菌Shannon指数和Evenness指数与PM无显著差异,与吴元凤等[39]对转基因抗虫棉种植后土壤氨氧化古菌群落多样性指数变化的研究结果一致。Shannon指数是用来估计群落多样性的高低的指标[40],多样性指数越高则群落结构越稳定,抗外界干扰的能力就越强,在2种玉米的4个生长期中,无论根际土还是非根际土古菌Shannon指数均呈现先升高后降低的趋势,并在乳熟期达到最高值。该结果说明玉米乳熟期时土壤群落结构最稳定,而到完熟期古菌Shannon指数降低,可能是由于玉米生长末期时根系活动程度降低,根系老化,分泌物减少所致。Evenness指数通常是用来描述物种中个体的相对丰富度或所占比例的一个指标,土壤Evenness指数呈先降低后升高再降低的趋势,在乳熟期古菌的Evenness指数达到最大值,说明在此时期某些古菌类群的相对丰度高于其他时期。
PCA分析结果表明,不同生长时期,PM和TM根际土和非根际土古菌群落未发生明显分离,说明古菌群落组成相对稳定,受转cry1Ab和epsps基因玉米C0030.3.5种植的影响不显著。Fang等[41]研究认为,转Bt基因玉米并未显著影响土壤微生物群落结构,反而是土壤质地和采样时间对其产生了影响,与本文结论基本一致。RDA分析发现古菌群落结构与硝态氮、总氮含量变化显著相关。氮素是所有生命体最重要的元素,植物吸收无机态氮在体内转化为有机态氮,而微生物兼具吸收无机态氮和有机态氮[42],在种植玉米的旱地土壤中,对氮素的利用主要以氧化态的硝态氮为主。因此,土壤中无机态氮和有机态氮都会对土壤古菌群落结构产生影响。氨氧化古菌作为古菌的主要组成类群,参与土壤氮循环,与土壤总氮和硝态氮关系密切。本课题组研究发现,氨氧化古菌在古菌中所占比例较高,且其群落结构随生长时期发生变化。这种变化也在土壤古菌总体群落变化中得以体现。
本研究从土壤古菌数量和群落结构变化2个方面探讨了转基因玉米种植对土壤古菌群落的影响。但本研究仅对1年和1个地点的土壤古菌群落进行了分析,不能全面地评价转基因玉米种植对土壤古菌群落的影响,并且由于方法的局限性,未能从古菌物种角度细致分析转基因玉米种植后土壤古菌群落结构的变化,因此在今后的研究中将进行长期多地区监测,并结合多种先进的分子生物学方法[43-44],如高通量测序等进行更全面的研究,为转基因作物的生态安全性评价提供更加全面的理论基础。
4 结论(1)转cry1Ab和epsps基因玉米C0030.3.5土壤古菌16S rRNA基因丰度与受体玉米DBN318无显著差异。玉米生长时期是影响土壤古菌16S rRNA基因丰度的主要因素。
(2)土壤古菌16S rRNA基因丰度与土壤总氮含量、速效磷含量、含水量和有机质含量呈正相关关系,而与pH值以及铵态氮和硝态氮呈负相关关系,但相关性均不显著。
(3)转cry1Ab和epsps基因玉米C0030.3.5土壤古菌16S rRNA基因的多样性与受体玉米DBN318无显著差异。玉米生长时期是古菌群落结构变化的主要影响因素,土壤总氮和硝态氮是调控土壤古菌群落结构变化的关键因子。
| [1] |
国际农业生物技术服务组织. 2016年全球生物技术/转基因作物商业化发展态势[J]. 中国生物工程杂志, 2017, 37(4): 1-8. International Service for the Acquisition of Agri-biotech Applications. Global trends in commercialization of global biotechnology/Transgenic crops in 2016[J]. China Biotechnology, 2017, 37(4): 1-8. |
| [2] |
Wolt J D, Peterson R K D. Prospective formulation of environmental risk assessment:Probabilistic screening for Cry1A(b) maize risk to aquatic insects[J]. Ecotoxicology and Environmental Safety, 2010, 73(6): 1182-1188. DOI:10.1016/j.ecoenv.2010.06.001 |
| [3] |
Neher D, Muthumbi A W N, Dively G P. Impact of coleopteran-active Bt corn on non-target nematode communities in soil and decomposing corn roots[J]. Soil Biology and Biochemistry, 2014, 76: 127-135. DOI:10.1016/j.soilbio.2014.05.019 |
| [4] |
Groffman P, Bohlen P. Soil and sediment biodiversity-cross-system comparisons and large-scale effects[J]. Bioscience, 1999, 49: 139-148. DOI:10.2307/1313539 |
| [5] |
杨永华. 转基因作物对土壤微生物群落的影响及主要研究策略[J]. 农业生物技术学报, 2011, 19(1): 1-8. YANG Yong-hua. Advances on the effects of genetically modified crops on soil microbial community and main countermeasures of their approaches[J]. Journal of Agricultural Technology, 2011, 19(1): 1-8. |
| [6] |
Woese C R, Fox G E. Phylogenetic structure of prokaryotic domain:The primary kingdoms[J]. Proceedings of the National Academy of Sciences of the United States of America, 1977, 74: 5088-5090. DOI:10.1073/pnas.74.11.5088 |
| [7] |
Kemnitz D, Kolb S, Conrad R. High abundance of crenarchaeota in a temperate acidic forest soil[J]. FEMS Microbiology Ecology, 2007, 60(3): 442-448. DOI:10.1111/fem.2007.60.issue-3 |
| [8] |
包丽君, 贾仲君. 模拟干湿交替对水稻土古菌群落结构的影响[J]. 土壤学报, 2017, 54(1): 191-203. BAO Li-jun, JIA Zhong-jun. Changes of archaeal community in a paddy soil under drying and re-wetting cycles[J]. Acta Pedologica Sinica, 2017, 54(1): 191-203. DOI:10.11766/trxb201603140046 |
| [9] |
Erkel C, Kube M, Reinhardt R, et al. Genome of rice cluster I archaea-the key methane producers in the rice rhizosphere[J]. Science, 2006, 313: 370-372. DOI:10.1126/science.1127062 |
| [10] |
Shimizu S, Upadhye R, Ishijima Y, et al. Methanosarcina horonobensis sp. nov., a methanogenic archaeon isolated from a deep subsurface Miocene formation[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61: 2503-2507. DOI:10.1099/ijs.0.028548-0 |
| [11] |
Yashiro Y, Sakai S, Ehara M, et al. Methanoregula formicica sp. nov., a methane-producing archaeon isolated from methanogenic sludge[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61: 53-59. DOI:10.1099/ijs.0.014811-0 |
| [12] |
Konneke M, Bernhard A E, Torre J R, et al. Isolation of an autotrophic ammonia-oxidizing marine archaeon[J]. Nature, 2005, 437: 543-546. DOI:10.1038/nature03911 |
| [13] |
Hatzenpichler R, Lebedeva E V, Spieck E, et al. A moderately thermophilic ammonia oxidizing crenarchaeote from a hot spring[J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105: 2134-2139. DOI:10.1073/pnas.0708857105 |
| [14] |
左娇, 郭运玲, 孔华, 等. 转基因玉米安全性评价研究进展[J]. 玉米科学, 2014, 22(1): 73-78. ZUO Jiao, GUO Yun-ling, KONG Hua, et al. Progress on safety evaluation of transgenic corn[J]. Journal of Maize Sciences, 2014, 22(1): 73-78. |
| [15] |
郭艳艳. 转基因抗虫玉米和棉花对非靶标生物的影响评价[D]. 北京: 中国农业大学, 2014. GUO Yan-yan. The impact of Bt transgenic corn and cotton on non-target organisms[D]. Beijing:Chinese Agricultural University, 2014. |
| [16] |
王柏凤, 宋新元, 常亮, 等. 转基因玉米C63-1种植对土壤跳虫的影响[J]. 应用昆虫学报, 2014, 51(5): 1215-1221. WANG Bai-feng, SONG Xin-yuan, CHANG Liang, et al. Effect of transgenic corn(C63-1) cultivation on soil collembola[J]. Chinese Journal of Applied Entomology, 2014, 51(5): 1215-1221. DOI:10.7679/j.issn.2095-1353.2014.144 |
| [17] |
邹雨坤, 张静妮, 杨殿林, 等. 转Bt基因玉米对根际土壤细菌群落结构的影响[J]. 生态学杂志, 2011, 30(1): 98-105. ZOU Yu-kun, ZHANG Jing-ni, YANG Dian-lin, et al. Effects of transgenic Bt maize on bacterial community structure in rhizosphere soil[J]. Chinese Journal of Ecology, 2011, 30(1): 98-105. |
| [18] |
崔红娟, 束长龙, 宋福平, 等. 转cry1Ah基因玉米对根际土壤微生物群落结构的影响[J]. 东北农业大学学报, 2011, 42(7): 30-38. CUI Hong-juan, SHU Chang-long, SONG Fu-ping, et al. Effects of transgenic cry1Ah maize on bacterial community structure in rhizosphere soil[J]. Journal of Northeast Agricultural University, 2011, 42(7): 30-38. |
| [19] |
Xie M, Yan J, Zhang D L, et al. Field studies show no significant effect of a Cry1Ab/Ac producing transgenic cotton on the fungal community structure in rhizosphere soil[J]. European Journal of Soil Biology, 2016, 73: 69-76. DOI:10.1016/j.ejsobi.2016.01.006 |
| [20] |
刘凯, 李永山, 范巧兰, 等. 转基因玉米对土壤微生物群系的影响[J]. 山西农业科学, 2015, 43(9): 1138-1140. LIU Kai, LI Yong-shan, FAN Qiao-lan, et al. Effects of transgenic corn on soil microbial population[J]. Journal of Shanxi Agricultural Sciences, 2015, 43(9): 1138-1140. |
| [21] |
尹昭坤, 乔建, 袁潜华. 转基因水稻向普通野生稻及其它近缘植物的基因漂流研究进展[J]. 热带生物, 2011, 4(2): 374-377. YIN Zhao-kun, QIAO Jian, YUAN Qian-hua. Progress on the gene flow from transgenic rice to common wild rice and its close relatives[J]. Journal of Tropical Organisms, 2011, 4(2): 374-377. |
| [22] |
Liu Y B, Chen F J, Guan X, et al. High crop barrier reduces gene flow from transgenic to conventional maize in large fields[J]. European Journal of Agronomy, 2015, 71: 135-140. DOI:10.1016/j.eja.2015.09.005 |
| [23] |
Xue K, Benjamin R D, Janice E Thies. Stability of Cry3Bb1 protein in soils and its degradation in transgenic corn residues[J]. Soil Biology & Biochemistry, 2014, 76: 119-126. |
| [24] |
鲍士旦. 土壤农化分析[M]. 第三版. 北京: 中国农业出版社, 2008. BAO Shi-dan. Soil analysis[M]. The third edition. Beijing: China Agriculture Press, 2008. |
| [25] |
Lueders T, Manefield M, Friedrich M W. Enhanced sensitivity of DNA and rRNA-based stable isotope probing by fractionation and quantitative analysis of isopycnic centrifugation gradients[J]. Environmental Microbiology, 2004(6): 2273-2278. |
| [26] |
Nakaya A, Onodera Y, Nakagawa T, et al. Analysis of ammonia monooxygenase and archaeal 16S rRNA gene fragments in nitrifying acid-sulfate soil microsms[J]. Microbes and Environments, 2009, 24(2): 168-174. DOI:10.1264/jsme2.ME09104 |
| [27] |
张慧, 袁红朝, 朱亦君, 等. 不同利用方式对红壤坡地微生物多样性和硝化势的影响[J]. 生态学杂志, 2011, 30(6): 1169-1176. ZHANG Hui, YUAN Hong-zhao, ZHU Yi-jun, et al. Microbial diversity and nitrification potential of sloping land red soil under different land use patterns[J]. Chinese Journal of Ecology, 2011, 30(6): 1169-1176. |
| [28] |
Lukow T, Dunfield P F, Liesack W. Use of the T-RFLP technique to access spatial and temporal changes in the bacterial community structure with in an agricultural soil with transgenic and no transgenic potato plants[J]. FEMS Microbiology Ecology, 2000, 32: 241-247. DOI:10.1111/fem.2000.32.issue-3 |
| [29] |
Yuan H Z, Ge T D, Wu X H, et al. Long-term field fertilization alters the diversity of autotrophic bacteria based on the ribulose-1, 5-biphosphate carboxylase/oxygenase(Rubisco) large-subunit genes in paddy soil[J]. Applied Microbiology and Biotechnology, 2012, 95: 1061-1071. DOI:10.1007/s00253-011-3760-y |
| [30] |
Hannula S E, de Boerw, Van J A. Do genetic modifications in crops affect soil fungi? A review[J]. Biology and Fertility of Soils, 2014, 50(3): 433-446. DOI:10.1007/s00374-014-0895-x |
| [31] |
Eilers K G, Debenport S, Anderson S, et al. Digging deeper to find unique microbial communities:The strong effect of depth on the structure of bacterial and archaeal communities in soil[J]. Soil Biology and Biochemistry, 2012, 50: 58-65. DOI:10.1016/j.soilbio.2012.03.011 |
| [32] |
Delong E F. Archaea in coastal marine environments[J]. Proceedings of the National Academy of Sciences of the United States of America, 1992, 89: 5685-5689. DOI:10.1073/pnas.89.12.5685 |
| [33] |
Karner M B, Delong E F, Karl D M. Archaeal dominance in the mesopelagic zone of the Pacific Ocean[J]. Nature, 2001, 409: 507-510. DOI:10.1038/35054051 |
| [34] |
Keough B P, Schmidt T M, Hicks R E. Archaeal nucleic acids in picoplankton from great lakes on three continents[J]. Microbial Ecology, 2003, 46: 238-248. DOI:10.1007/s00248-003-1003-1 |
| [35] |
Jurgens G, Lindstrom K, Saano A. Novel group within the kingdom Crenarchaeota from boreal forest soil[J]. Applied and Environmental Microbiology, 1997, 63: 803-805. |
| [36] |
Ochsenreiter T, Selezi D, Quaiser A, et al. Diversity and abundance of Crenarchaeota in terrestrial habitats studied by 16S RNA surveys and real time PCR[J]. Environmental Microbiology, 2003(5): 787-797. |
| [37] |
冯帅, 刘小利, 吴小丽, 等. 不同水分条件对玉米根际微生物群落的影响[J]. 作物杂志, 2017(1): 127-134. FENG Shuai, LIU Xiao-li, YU Xiao-li, et al. Effects of different water conditions on rhizosphere microbial community of maize[J]. Crop Journal, 2017(1): 127-134. |
| [38] |
马永玉. 酸性土壤颗粒表面电场对氨氧化细菌和古菌群落结构的影响[D]. 重庆: 西南大学, 2011. MA Yong-yu. Effects of surface electro-chemical property on community structure of ammonina-oxdizing bacteria and archaea in an acid soil[D]. Chongqing:Southwest University, 2011. http://d.wanfangdata.com.cn/Thesis/Y1882821 |
| [39] |
吴元凤, 李刚, 修伟明, 等. 双价转基因抗虫棉花对土壤氨氧化细菌和氨氧化古菌群落结构及丰度的影响[J]. 农业环境科学学报, 2014, 33(11): 2155-2163. WU Yuan-feng, LI Gang, XIU Wei-ming, et al. Community structure and abundance of soil ammonia-oxidizing bacteria and ammonia-oxidizing archea as influenced by insect-resistant bivalent transgenic cotton[J]. Journal of Agro-Environment Science, 2014, 33(11): 2155-2163. DOI:10.11654/jaes.2014.11.013 |
| [40] |
马琳, 孙本华, 孙瑞, 等. 长期不同施肥对土娄土细菌群落多样性的影响[J]. 西北农业学报, 2015, 24(6): 162-170. MA Lin, SUN Ben-hua, SUN Rui, et al. Effects of long-term different fertilization on bacterial community diversity in an anthropogenic Lou soil[J]. Acta Agriculturae Boreali-occidentalis Sinica, 2015, 24(6): 162-170. DOI:10.7606/j.issn.1004-1389.2015.06.026 |
| [41] |
Fang M, Kremer R J, Motavalli P P, et al. Bacterial diversity in rhizospheres of nontransgenic and transgenic corn[J]. Applied and Environmental Microbiology, 2005, 71: 4132-4136. DOI:10.1128/AEM.71.7.4132-4136.2005 |
| [42] |
奚振邦. 硝态氮与铵态氮[J]. 磷肥与复肥, 2011, 26(1): 62-65. XI Zhen-bang. Nitrate and ammonium nitrogen[J]. Phosphate Fertilizer and Compound Fertilizer, 2011, 26(1): 62-65. |
| [43] |
Lauber C L, Ramirez K S, Aanderud Z, et al. Temporal variability in soil microbial communities across land-use types[J]. The ISME Journal, 2013, 7(8): 1641-1650. DOI:10.1038/ismej.2013.50 |
| [44] |
Dumont M G, Pommerenke B, Casper P. Using stable isotope probing to obtain a targeted metatranscriptome of aerobic methanotrophs in lake sediment[J]. Environmental Microbiology Reports, 2013, 5(5): 757-764. |
 2017, Vol. 36
2017, Vol. 36





