文章信息
- 王志刚, 陈文晶, 胡影, 徐伟慧, 王春龙, 吕智航, 刘泽平
- WANG Zhi-gang, CHEN Wen-jing, HU Ying, XU Wei-hui, WANG Chun-long, LÜ Zhi-hang, LIU Ze-ping
- DNA在黑土胶体微界面的吸附与解吸特性
- Characteristics of DNA adsorption and desorption on micro-interfaces of black soil colloids
- 农业环境科学学报, 2017, 36(10): 2058-2062
- Journal of Agro-Environment Science, 2017, 36(10): 2058-2062
- http://dx.doi.org/10.11654/jaes.2017-0619
文章历史
- 收稿日期: 2017-04-27
土壤中包含大量的生命有机体,富含DNA和酶等生物活性物质。土壤环境中的胞外DNA主要来源于微生物、动植物细胞的主动分泌或细胞裂解释放,以往认为环境中的DNA会立即被存在的DNase Ⅰ所降解[1],而根据大量研究表明,生物释放到环境中的DNA能被其周围的土壤胶体吸附固定[2-3],使DNA对DNase Ⅰ的反应特性以及敏感度发生改变,进而抵御DNase Ⅰ的降解而长期保存于环境中,直至被合适的宿主细胞所吸收利用[4-5],在特定条件下,在合适宿主细胞内发生遗传转化,DNA基因编码就能得到表达。土壤胞外DNA可以通过重组、转导、转化和转位等途径在微生物之间转移,甚至在植物与微生物之间发生转移[6],在微生物的遗传进化和生物多样性等方面起着重要作用[7]。这种环境中的“隐性基因”已经引起医学、生物学、土壤学、生命起源学等相关领域的广泛关注。
土壤胶体对DNA的固定,使土壤成为DNA活性保存的载体和基因转移的重要媒介[8]。土壤胶体包括有机胶体和无机胶体,它是土壤肥力的重要物质基础[9]。东北黑土为世界上仅有的四大黑土区之一,有机质含量高达5%~7%,是我国重要的自然资源。在黑土中栖息着大量的微生物[10],这些生物的新陈代谢势必要向土壤环境释放大量的胞外DNA,而胶体与DNA的吸附与解吸特性直接影响着DNA在宿主细胞中的水平遗传转化过程[11]。因此,研究DNA在黑土胶体中的吸附与解吸过程,对揭示DNA在黑土微生物的遗传进化和基因转移机制等方面的作用具有重要意义。为此,本文研究了黑土有机与无机胶体对DNA的吸附与解吸特性,以期为揭示土壤胞外DNA分子存在状态和归宿以及生态安全评价提供科学依据。
1 材料与方法 1.1 实验材料 1.1.1 供试DNA使用德国Sigma公司生产的鲑鱼精DNA为供试材料。用双蒸水溶解后使用,配制成500 μg·mL-1的储备液,使用超微量分光光度计2000 C(Thermo Fisher Scientific,美国)测定其A260/A280值为1.86,于4 ℃冰箱保存备用。
1.1.2 黑土胶体的制备实验所用黑土采自黑龙江省克山县(126°4′57″E,48°21′43″N)农田,室温条件下自然风干,剔除植物根系、碎石块与草屑等异物,研磨并全部过200目筛。
土壤胶体的提取方法参照Li等[12]实验方法并加以改进,具体步骤为:称取50 g黑土样品两份,分别置于500 mL烧杯中,加100~150 mL纯水搅拌均匀,其中一份加入适量的双氧水(30%),不断搅拌直至没有明显气泡产生,除净有机质后,用于制备去有机质胶体(Coarse inorganic colloid,CIC),另一份不需要双氧水处理,用于制备含有机质胶体(Coarse organic colloid,COC),两份悬液超声分散1 h,期间不断搅拌,然后把土壤悬液浇注于320目筛,以除去砂粒。过筛后的土壤悬液转入2000 mL烧杯,用超纯水定容至2000 mL,上下搅拌(避免产生旋涡)后静置。根据Stokes定律计算沉淀粒径<2 μm胶体颗粒所需时间为2 h,反复多次提取胶体,直至悬液澄清透亮。在收集的胶体溶液中加入250 mL的0.5 mol·L-1 CaCl2溶液絮凝胶体,倾去上清液。用20 mL去离子水与20 mL 95%的无水乙醇清洗絮凝物,采用AgNO3滴定法检测Cl-,直至无Cl-。最后将黑土胶体冷冻干燥后,用玛瑙研钵研磨过200目筛后,置于干燥器中保存备用。
1.1.3 黑土胶体悬液的制备准确称取1.000 g黑土胶体于50 mL容量瓶中,加入灭菌的超纯水,超声波分散1 h并充分摇匀,用0.1 mol·L-1的NaOH与HCl溶液多次调节pH值使其稳定在7.0,补充超纯水使总体积为50 mL,得到COC和CIC胶体悬液,胶体浓度为20 mg·mL-1,避光保存备用。矿物组成用X-射线衍射法(XRD)进行测定。总有机质含量(Total organic carbon,TOC)和阳离子交换量(Cation exchange capacity,CEC)分别用KCr2O7-H2SO4外加热法和中性乙酸铵法测定。比表面积(Specific surface area,SSA)采用BET氮气吸附法进行测定,测定条件:80 ℃真空脱气3 h。胶体电荷零点(Point of zero charge,PZC)用zeta电位仪测定。供试黑土胶体的形貌见图 1,理化性质见表 1。如图 1所示,含有机质胶体与去有机质胶体均呈现层状结构。

|
| 图 1 扫描电镜下的土壤胶体形貌图(5 μm) Figure 1 Scanning electron microscope images of COC and CIC (5 μm) |
在10 mL DNA LoBind低吸离心管(Eppendorf)中分别加入1.6 mL Tris-HCl缓冲液(0.05 mol·L-1,pH 7.4)、0.4 mL DNA溶液、0.5 mL黑土胶体悬液,控制总体系为2.5 mL;25 ℃条件下,旋转振荡10、20、40、60、80、120、180 min;振荡完毕后取出,14 500 r·min-1离心20 min,上清液用超微量分光光度计在260 nm波长比色测定;为避免胶体的不完全沉淀对实验结果造成影响,上清液中的DNA含量等于“胶体+DNA”上清的DNA量减去胶体吸附后的上清DNA量,获得DNA在土壤胶体上的吸附量。实验设置清水、胶体和DNA溶液3个处理,每个实验处理3组平行。
1.3 等温吸附曲线10 mL DNA LoBind低吸离心管(Eppendorf)中分别加入1.6 mL 0.05 mol·L-1 Tris-HCl缓冲液(pH 7.4)、0.5 mL黑土胶体悬液,以及0.4 mL DNA溶液,控制总体系为2.5 mL。体系中的DNA终浓度为10、20、40、60、80、100、120、140 μg·mL-1。后续步骤同1.2所示。
1.4 pH值对DNA吸附的影响在10 mL离心管中分别加入1.6 mL不同pH值(2、3、4、5、6、7、8、9)的TE缓冲液、0.4 mL DNA溶液、0.5 mL黑土胶体悬液,控制总体系为2.5 mL。后续步骤同1.2所示。
1.5 解吸实验解吸实验是根据等温吸附实验结果,在DNA吸附平衡后,在体系中加入10 mmol·L-1 Tris-HCl缓冲液(0.05 mol·L-1,pH 7.4),振荡30 min,然后测缓冲液的OD260nm值,反复解吸3~5次,直到上清液中检测不到DNA为止。DNA的解吸率由下方公式求得:
DNA解吸率=(DNA解吸量/DNA吸附量)×100%
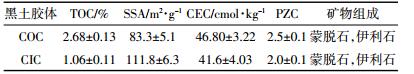
2 结果与分析 2.1 黑土胶体对DNA的吸附过程由图 2可知,DNA在COC和CIC表面均可以发生吸附过程,但是吸附过程规律却完全不同。COC对DNA的吸附过程呈现明显的三个阶段:快速吸附期(0~10 min)、平缓吸附期(10~80 min)和吸附平衡期(>80 min);CIC对DNA的吸附过程分为两个阶段:快速吸附期(0~40 min)和吸附平衡期(>40 min)。在快速吸附期,COC吸附曲线斜率明显高于CIC,说明COC吸附速率高于CIC。在整个吸附过程中,CIC表面吸附DNA的量小于COC,且更快达到吸附平衡。
|
| 图 2 DNA在黑土胶体上的吸附动力学特征 Figure 2 Kinetic characteristics of DNA adsorption on black soil colloids |
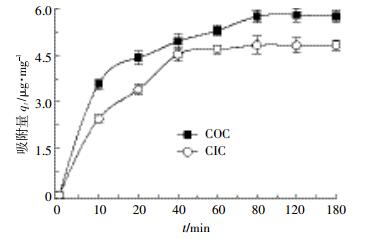
pH是影响DNA在土壤胶体表面吸附的重要因素,由图 3可知,黑土胶体表面的DNA吸附量随着体系pH值的上升而降低。pH<5时,COC对DNA的吸附量从16.62 μg·mg-1降低到6.25 μg·mg-1,CIC表面DNA的吸附量从15.52 μg·mg-1降低到9.54 μg·mg-1;pH≥6时,随着pH升高,COC和CIC对DNA的吸附量持续降低,但COC的吸附量高于CIC,在pH=9时,CIC的吸附量仅为1.5~2 μg·mg-1。
|
| 图 3 pH对DNA在黑土胶体表面吸附的影响 Figure 3 Effect of pH on DNA adsorption on black soil colloids |
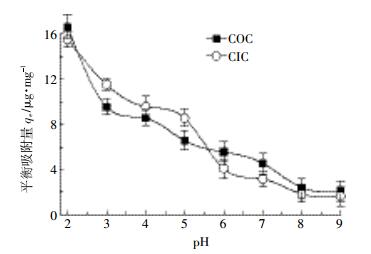
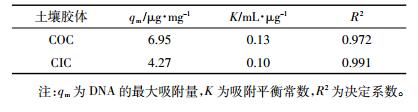
由图 4可知,在黑土胶体吸附体系中,当DNA浓度低于30 μg·mL-1时,吸附等温线的斜率较大,吸附量随DNA浓度的增加快速上升;而当DNA浓度大于30 μg·mL-1时,等温吸附曲线变得较为平缓,表明吸附逐渐趋于饱和。在所有DNA浓度下,CIC的吸附量小于COC,且较COC的DNA饱和浓度低。通过Langmuir吸附方程对黑土胶体等温吸附DNA曲线的拟合发现(表 2),COC的决定系数(R2)为0.972,最大吸附量(qm)为6.95 μg·mg-1,CIC的R2为0.991,qm为4.27 μg·mg-1。COC的吸附平衡常数(K)大于CIC,说明COC对DNA的吸附亲和能力高于CIC。
|
| 图 4 黑土胶体对DNA的等温吸附曲线 Figure 4 Curves of isotherm adsorption of DNA on black soil colloids |
|
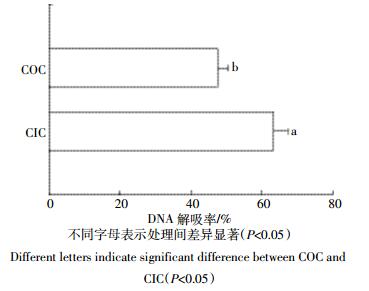
吸附在黑土胶体表面的DNA用0.05 mol·L-1 Tris-HCl缓冲液进行解吸,解吸2~3次后,溶液中DNA解吸完全,由图 5可看出,CIC表面DNA的解吸率为63.5%,而COC表面DNA的解吸率为47.4%,CIC表面的DNA解吸率较高,也印证了Langmuir等温吸附模型中,COC对DNA的亲和力大于CIC的结论。
|
| 不同字母表示处理间差异显著(P < 0.05) Different letters indicate significant difference between COC and CIC (P < 0.05) 图 5 黑土胶体表面DNA的解吸 Figure 5 Desorption of DNA on black soil colloids |
土壤胶体具有巨大的比表面积与表面电荷特性,土壤胶体与进入其中的物质间有很高的亲和性,其微界面过程是土壤研究的热点。DNA分子质子化后可携带电荷,能与携带相反电荷的土壤胶体表面结合[13]。土壤胶体界面吸附的DNA可以抵御核酸酶降解和其他物质损伤[14],增加DNA在环境中的存活周期,提升其发生遗传转化的几率[15-16],对环境生物水平遗传转化具有重要意义。
通过X-射线衍射法研究发现,黑土胶体的主要矿物成分为高岭石和蒙脱石,蒙脱石或高岭石的断裂边缘具有活性羟基,可作为质子供体或受体而与其他物质形成氢键,DNA分子可通过氢键与土壤颗粒发生吸附作用[17],Cai等[18]证明DNA可以通过静电引力吸附在蒙脱石表面,而DNA分子与高岭石之间还存在配体交换作用,CIC和COC的吸附曲线进一步证明了DNA可以与高岭石和蒙脱石发生吸附作用,并且有机质的存在强化了这种吸附作用。CIC表面DNA快速吸附期的吸附速率和吸附量都小于COC,且更快达到吸附平衡。在Langmuir吸附方程中,COC的K值大于CIC值,说明COC对DNA的吸附亲和能力大于CIC,这可能是由于有机质更容易与DNA形成相对稳定的氢键所导致的[19];解吸实验中,CIC表面DNA的解吸率为63.5%,显著高于COC表面DNA的解吸率47.4%,结果与Langmuir方程的研究结论一致,说明有机质在黑土胶体对DNA的吸附过程中起关键作用,而且有机质含有较多的官能团,可通过氢键等作用而增强对DNA的吸附。
在不同pH条件下,黑土胶体吸附DNA的规律与火山土胶体的特征相反[20],可能是由于DNA的等电点约为5,当体系pH<5时,DNA表面带正电荷,DNA与黑土胶体之间的静电引力作用增强,因此DNA的吸附量较大;而pH>5时,DNA分子中的磷酸基团发生去质子化,DNA带负电荷。同时,由于黑土胶体的电荷零点为2.0左右,当体系的pH值上升,DNA与胶体表面的负电荷均增加,静电排斥作用增强,因此吸附量降低。这表明,DNA与胶体表面存在静电作用[21],但低pH可能会导致DNA沉淀,也可能是吸附量增加的原因[22],但是其作用机制仍需要进一步研究。
4 结论(1)DNA可在黑土胶体表面发生吸附作用,快速吸附期COC对DNA的吸附速率和吸附量都高于CIC。
(2)COC对DNA的吸附亲和能力大于CIC,说明有机质在黑土胶体对DNA的吸附过程中起关键作用。
(3)黑土胶体对DNA的吸附量随着pH值的升高而降低,pH是影响黑土胶体吸附DNA的重要因子。
致谢 Kui Chen教授在论文写作过程中提供了宝贵意见,谨致谢忱。| [1] |
Stotzky G. Persistence and biological activity in soil of insecticidal proteins from Bacillus thuringiensis and of bacterial DNA bound on clays and humic acids[J]. Journal of Environmental Quality, 2000, 29(3): 691-705. |
| [2] |
Crecchio C, Ruggiero P, Curci M, et al. Binding of DNA from Bacillus subtilis on montmorillonite-humic acids-aluminum or iron hydroxy polymers[J]. Soil Science Society of America Journal, 2005, 69(3): 834-841. DOI:10.2136/sssaj2004.0166 |
| [3] |
James C, Ellen C P, Caroline M J, et al. The adsorption of short single-stranded DNA oligomers to mineral surfaces[J]. Chemosphere, 2011, 83(11): 1560-1567. DOI:10.1016/j.chemosphere.2011.01.023 |
| [4] |
Pietramellara G, Ascher J, Borgogni F, et al. Extracellular DNA in soil and sediment:Fate and ecological relevance[J]. Biology and Fertility of Soils, 2009, 45(3): 219-235. DOI:10.1007/s00374-008-0345-8 |
| [5] |
Rizzi A, Raddadi N, Sorlini C, et al. The stability and degradation of dietary DNA in the gastrointestinal tract of mammals:Implications for horizontal gene transfer and the biosafety of GMOs[J]. Critical Reviews in Food Science and Nutrition, 2012, 52(2): 142-161. DOI:10.1080/10408398.2010.499480 |
| [6] |
Hoffmann T, Golz C, Scheider O. Foreign DNA sequences are received by a wild-type strain of Aspergillus niger after co-culture with transgenic higher plants[J]. Current Genetics, 1994, 27(1): 70-76. DOI:10.1007/BF00326581 |
| [7] |
Stotzky G. Persistence and biological activity in soil of insecticidal proteins from Bacillus thuringiensis and of bacterial DNA bound on clays and humic acids[J]. Journal of Environmental Quality, 2000, 29(3): 691-705. |
| [8] |
廖敏, 谢晓梅, 方舒, 等. 不同粒径红壤胶体颗粒对DNA的吸附特性[J]. 应用生态学报, 2013, 24(3): 764-770. LIAO Min, XIE Xiao-mei, FANG Shu, et al. Characteristics of DNA adsorption on different sizes red soil colloidal particles[J]. Chinese Journal of Applied Ecology, 2013, 24(3): 764-770. |
| [9] |
刘淑霞, 王宇, 周平, 等. 不同施肥对黑土有机无机复合及腐殖质结合形态的影响[J]. 南京农业大学学报, 2008, 31(2): 76-80. LIU Shu-Xia, WANG Yu, ZHOU Ping, et al. Effect of various fertilization on the organo-mineral complexation and the combined forms of humus of Phaeozem[J]. Journal of Nanjing Agriculture University, 2008, 31(2): 76-80. |
| [10] |
Ding J L, Jiang X, Ma M C, et al. Effect of 35 years inorganic fertilizer and manure amendment on structure of bacterial and archaeal communities in black soil of Northeast China[J]. Applied Soil Ecology, 2016, 105: 187-195. DOI:10.1016/j.apsoil.2016.04.010 |
| [11] |
Beall G W, Sowersby D S, Roberts R D, et al. Analysis of oligonucleotide DNA binding and sedimentation properties of montmorillonite clay using ultraviolet light spectroscopy[J]. Biomacromolecules, 2009, 10(1): 105-112. DOI:10.1021/bm800970v |
| [12] |
Li W, He Y, Wu J, et al. Extraction and characterization of natural soil nanoparticles from Chinese soils[J]. European Journal of Soil Science, 2012, 63(5): 754-761. DOI:10.1111/ejs.2012.63.issue-5 |
| [13] |
Nguyen T H, Elimelech M. Plasmid DNA adsorption on silica:Kinetics and conformational changes in monovalent and divalent salts[J]. Biomacromolecules, 2007, 8(1): 24-32. DOI:10.1021/bm0603948 |
| [14] |
Wu P X, Li W, Zhu Y J, et al. The protective effect of layered double hydroxide against damage to DNA induced by heavy metals[J]. Applied Clay Science, 2014, 100(10): 76-83. |
| [15] |
Recothet G, Picard C, Mormand P, et al. Kinetics of the persistence of chromosomal DNA from genetically engineered Escherichia coli introduced into soil[J]. Applied and Environmental Microbiology, 1993, 59(12): 4289-4294. |
| [16] |
Gallori E, Bazziealupo M, Canto D L, et al. Transformation of Bacillus subtilis by DNA bound on clay in non-sterile soil[J]. FEMS Microbiology Ecology, 1994, 15(1/2): 119-126. |
| [17] |
Adélia J A, Daniel T, Georg H, et al. Solvent effects on hydrogen bonds:A theoretical study[J]. Journal of Physical Chemistry A, 2002, 106(9): 1862-1871. DOI:10.1021/jp013677x |
| [18] |
Cai P, Zhu J, Huang Q Y, et al. Role of bacteria in the adsorption and binding of DNA on soil colloids and minerals[J]. Colloids & Surfaces B:Biointerfaces, 2009, 69(1): 26-30. |
| [19] |
Saeki K, Sakai M. The influence of soil organic matter on DNA adsorptions on andosols[J]. Microbes and Environments, 2009, 24(2): 175-179. DOI:10.1264/jsme2.ME09117 |
| [20] |
Saeki K, Kunito T, Sakai M. Effects of pH, ionic strength, and solutes on DNA adsorption by andosols[J]. Biology and Fertility of Soils, 2010, 46(5): 531-535. DOI:10.1007/s00374-010-0447-y |
| [21] |
Lee G U, Chrisey L A, Colton R J. Direct measurement of the forces between complementary strands of DNA[J]. Science, 1994, 266(5186): 771-773. DOI:10.1126/science.7973628 |
| [22] |
Cai P, Huang Q Y, Zhang X, et al. Adsorption of DNA on clay minerals and various colloidal particles from an Alfisol[J]. Soil Biology & Biochemistry, 2006, 38(3): 471-476. |
 2017, Vol. 36
2017, Vol. 36