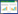文章信息
- 陈凯丽, 吴蔓莉, 叶茜琼, 李炜, 袁婧
- CHEN Kai-li, WU Man-li, YE Xi-qiong, LI Wei, YUAN Jing
- 生物修复对石油污染土壤微生物活性的影响
- Impacts of bioremediation on microbial activities in petroleum contaminated soil
- 农业环境科学学报, 2017, 36(2): 279-285
- Journal of Agro-Environment Science, 2017, 36(2): 279-285
- http://dx.doi.org/10.11654/jaes.2016-0991
文章历史
- 收稿日期: 2016-08-01
利用微生物修复法处理土壤具有成本低、无二次污染等优点,目前已成为一种广泛采用的土壤修复技术[1-2]。修复过程中土壤的微生物活性,尤其是降解菌的代谢能力对石油烃的降解起着关键作用[3-4]。文献报道的用于表征土壤微生物活性的指标主要包括土壤酶活、腺嘌呤核苷三磷酸(Adenosine-triphosphate,ATP)含量以及耗氧速率(Oxygen uptake rate,OUR)等[5]。由于土壤酶在有机物分解和有毒物质降解等重要代谢过程中起着催化剂的作用,因此可利用土壤酶活性指示土壤微生物活性的变化。
对于石油烃降解菌的研究,一般采用最大可能计数法或者平板计数法[6-7]。这些方法只能用于测定土壤中降解菌数量的多少,不能反映降解菌的实际代谢活性。Biolog法具有灵敏度高、分辨力强、测定简便、无需分离培养纯种微生物即可进行测定等优点,是目前已知的研究微生物代谢功能多样性的重要方法,已被广泛应用于评价土壤及水体中微生物群落功能的多样性[8-11]。但是,以石油烃不同组分作为自定义碳源,利用Biolog法对石油烃降解菌代谢活性进行研究的工作,文献报道相对较少。
本研究利用微生物修复法对陕北某地受石油污染的土壤进行了实验室修复研究,修复过程中沿程测定烷烃和多环芳烃浓度变化以评价修复效果。利用Biolog-MT2板测定降解菌对不同组分石油烃的代谢能力,同时测定了土壤的四种酶活以表征土壤微生物活性;利用SPSS软件分析土壤酶活、降解菌活性及不同组分烃之间的相关关系。研究结果可为明确微生物修复过程中不同组分烃的去除特性、土壤微生物活性变化规律提供一定的理论依据。
1 材料与方法 1.1 供试土壤和石油烃降解菌石油污染土壤取自陕北某油井附近受石油污染的表层土(0~10 cm)。土样经过除杂、研磨、过筛(2 mm),充分混匀后置于自然环境下,备用。土壤的理化性质如表 1所示。土壤中总石油烃(TPH)浓度为40 013 mg·kg-1,其中,烷烃25 988 mg·kg-1,占总石油烃浓度的64.9%,多环芳烃5323 mg·kg-1,占总石油烃浓度的13.3%。
实验研究中所用的12株菌是前期从甘肃庆阳、陕北子长和陕北清涧石油污染土壤中分离出来的石油烃降解菌[12],包括5株假单胞菌属、2株芽孢杆菌、3株无色产碱杆菌、1株黄杆菌,1株不动杆菌。
1.2 实验方案设计及测定方法将12株菌制成OD值为1.0(600 nm处测定值)的混合菌悬液:将实验室保存的12株单株降解菌分别接种至50 mL牛肉膏蛋白胨液体培养基中,置于恒温水浴振荡培养器中30 ℃、130 r·min-1培养1 d,在4 ℃、10 000 r·min-1条件下离心10 min,弃去上清液,向离心管加入30 mL PBS缓冲液,摇匀,在相同条件下离心10 min,弃去上清液。如此反复3次,用PBS调节每株菌的菌悬液OD值为1.0(600 nm处测定值),取等量单株菌悬液混合即制得混合菌悬液[12]。
称取900 g供试土壤装于圆形瓷盆中,向土壤中投加12株混合菌悬液10 mL(OD600=1.0),使土壤中外加降解菌个数为108 cfu·g-1,同时向土壤中加入(NH4)2SO4和KH2PO4调节土壤C/N/P=100/10/1,对土壤进行微生物修复处理(BAS)。另取900 g供试土壤,不进行任何处理作为控制实验(CK)。每种处理3个平行。室温条件下,连续进行11周的修复,修复期间每天翻动土壤保持透气性。
每周取2 g土样,利用超声波萃取法提取土壤中的总石油烃[4],利用重量法测定烷烃和多环芳烃含量的变化[13]。
土壤酶活性的测定参照《土壤酶及其研究法》[14]:利用三苯基甲胺(TF)法测定脱氢酶活性,酶活性以1 g土壤产生1 μg TF的量表示;脂肪酶活性测定采用对硝基苯基丁酸-异丙醇法,酶活性以μg PNP·g-1干土表示;多酚氧化酶测定采用比色法,酶活性以1 g土壤中紫色没食子素的毫克数表示;利用高锰酸钾滴定法测定过氧化氢酶,酶活性以1 g干土消耗的高锰酸钾(0.1 mol·L-1)体积数表示。
降解菌代谢活性的测定:利用Biolog MT2板分析土壤中可降解烃类微生物的代谢活性时,共设2种碳源,分别是正十六烷烃和混合多环芳烃(菲1 g、蒽0.1 g、芘0.1 g,用二氯甲烷溶解并定容至100 mL)。
取修复第1~11周的土壤样品1 g,加入99 mL无菌蒸馏水,振荡20 min后沉降30 min,取150 μL上清液接种到MT2板的微孔中,在微孔内分别加入5 μL正十六烷烃、3种多环芳烃混合物作为碳源,其中第1个孔不加任何碳源,作为对照。将接种好的MT2板放入恒温培养箱中(25 ℃),每隔24 h于590 nm处测定光度值,测定时间为168 h。根据文献方法计算孔的平均颜色变化率(Average well color development,AWCD)[15]。
AWCD=[Σ(Ci-R)]/95
式中:Ci为第i个反应孔的OD值;R为对照孔的OD值。
1.3 数据统计分析和处理利用SPSS 19.0软件对不同组分烃的降解率、降解菌的活性以及4种土壤酶活的测定数据进行统计分析,利用双变量法进行相关性分析,在P<0.01水平对降解菌代谢活性、土壤微生物活性及烃降解率进行相关分析[12]。
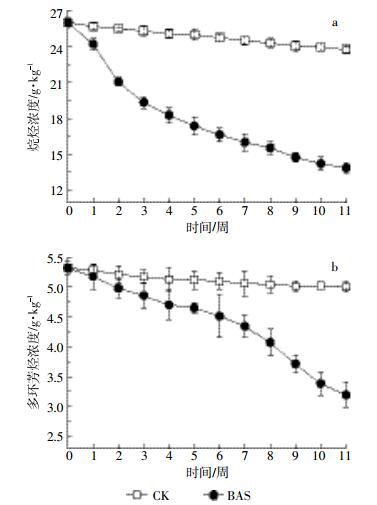
2 结果与讨论 2.1 微生物修复对不同组分烃的去除作用微生物修复法对土壤中不同组分烷烃的去除情况如图 1a所示。经过11周的修复,土壤中烷烃的浓度由25 988 mg·kg-1降低为13 800 mg·kg-1,总去除率为46.8%;在自然条件下修复的土壤(CK)中烷烃浓度由25 988 mg·kg-1降低为23 700 mg·kg-1,总去除率为8.8%。研究结果表明,微生物修复作用可有效去除土壤中大部分烷烃。
|
| 图 1 土壤中不同组分烃的浓度变化 Figure 1 The concentrations of different fractional petroleum hydrocarbons in the soil |
土壤中多环芳烃的降解情况如图 1b所示。修复11周后,土壤中多环芳烃的浓度由5323 mg·kg-1降低为3200 mg·kg-1,去除率为39.9%;自然条件下修复的土壤(CK)中多环芳烃浓度由5323 mg·kg-1降低为5000 mg·kg-1,去除率为5.6%。与控制实验相比,微生物修复作用可有效去除土壤中的多环芳烃。
经过11周的修复,烷烃的总去除率为46.8%,多环芳烃的总去除率为39.9%,表明在利用微生物法修复石油污染土壤时,对土壤中不同组分烃的去除效果存在差异,烷烃比多环芳烃更易降解。实验结果与文献所得的研究结果一致[16-17]。
利用下式计算烷烃、多环芳烃的周降解率,绘制降解率随时间变化的曲线,所得结果如图 2所示。
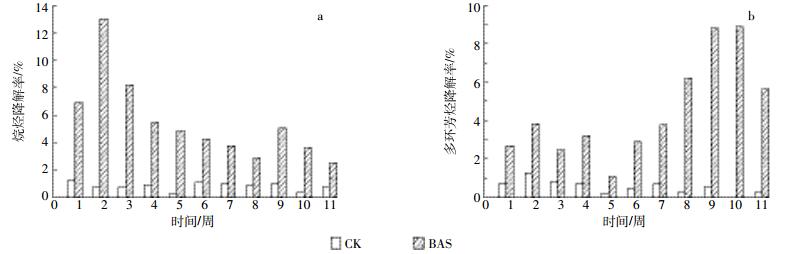
|
| 图 2 土壤中不同组分烃的周降解率 Figure 2 The weekly degradation efficiencies of different fractional hydrocarbons in the soil |

根据图 2可知,烷烃周降解率在修复的前4周较大,多环芳烃周降解率在修复的最后4周较大。
2.2 土壤酶活变化修复过程中土壤酶活的变化情况如图 3所示。利用微生物法进行修复处理的土壤中(BAS)多酚氧化酶活性在第2周时达到最大,随后降低;脱氢酶活性在第4、5周时最大,第6周开始降低;过氧化氢酶活在修复的前4周呈增加趋势,第4周后保持不变;脂肪酶活性在修复第2周急剧增加,此后10周内均保持较高的活性。
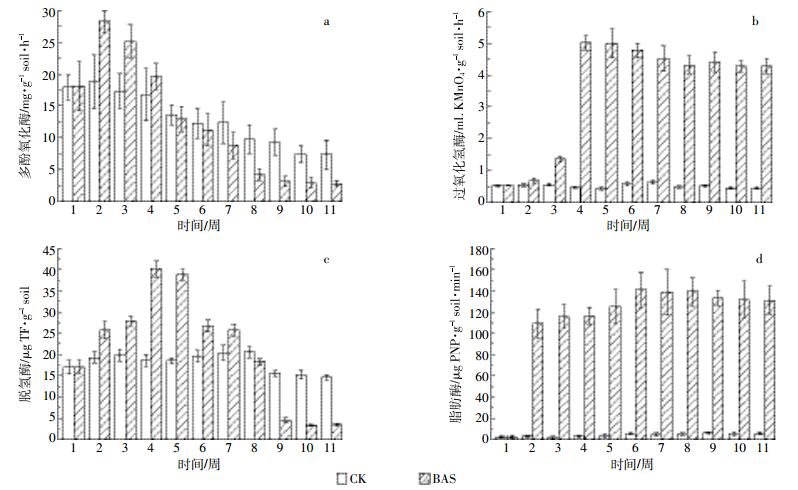
|
| 图 3 修复过程中土壤酶活的变化情况 Figure 3 The changes of four enzyme activities in the different treatment soil (a)多酚氧化酶Polyphenoloxidase;(b)过氧化氢酶Catalase;(c)脱氢酶Dehydrogenase;(d)脂肪酶Lipase |
未经处理的控制土壤(CK)中脱氢酶、过氧化氢酶、脂肪酶活性在修复期间基本保持不变;多酚氧化酶活性呈先降低后稳定的趋势。
与不经修复处理的土壤(CK)相比,经过微生物修复处理的土壤中过氧化氢酶、脂肪酶活性均显著提高;多酚氧化酶、脱氢酶活性都呈先增加后降低的趋势,且在修复后期(7~8周后)这两种酶的活性已明显低于自然放置的土壤中的酶活性。
过氧化氢酶可以促进土壤微生物对过氧化氢的降解作用[18-19],脂肪酶能将土壤中羧酸脂类有机化合物水解为可溶性物质[20-21]。多酚氧化酶、脱氢酶都属于氧化还原酶,可催化有机化合物的氧化还原反应[22-24]。文献报道脱氢酶在石油烃的氧化和转化过程中起着非常重要的作用[25]。土壤脱氢酶和多酚氧化酶在修复前期活性增加,可能是由于投加的降解菌很快适应石油污染物的胁迫环境,对土壤中存在的石油烃存在较好的降解性能;在修复后期两种酶活性降低,可能是由于在修复过程中降解的石油烃组分减少,难降解石油烃组分及有毒代谢产物的积累,导致了土壤微生物活性降低。
2.3 石油烃降解菌代谢活性变化分别以正十六烷烃和多环芳烃为自定义碳源,利用Biolog法对石油烃降解菌的代谢活性进行了测定,所得结果如图 4所示。AWCD值表征了微生物对两种碳源的代谢能力。在分别以两种烃作为碳源的情况下,AWCD值在生物修复处理后的1~2周内达到最大,之后开始下降。以正十六烷烃为碳源时,CK和BAS处理的土壤AWCD值范围分别为0.17~0.49和0.17~1.15;以多环芳烃为碳源时,CK和BAS处理的土壤AWCD值范围分别为0.07~0.17和0.07~0.24,且经过生物修复处理的土壤AWCD值大于未经生物修复处理的土壤。研究结果说明微生物修复作用可提高土壤微生物对不同组分石油烃的代谢能力,同一处理中土壤微生物对正十六烷烃的利用能力强于对多环芳烃的利用能力,且修复前期土壤微生物对碳源的代谢活性较强。
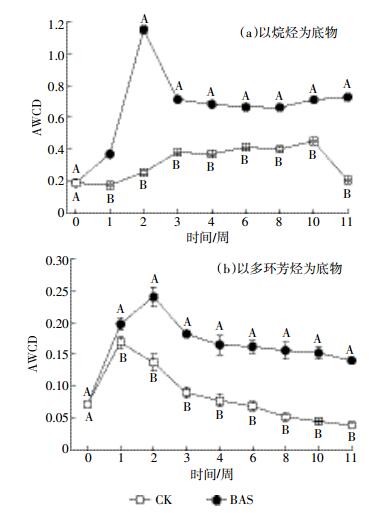
|
| 图 4 土壤微生物对烷烃、 多环芳烃的代谢活性 Figure 4 The hydrocarbon metabolic activities of soil microorganisms in the soil |
利用SPSS 19.0软件对土壤酶活、不同组分烃浓度变化、降解菌的代谢活性进行相关性分析,所得结果如表 2所示。
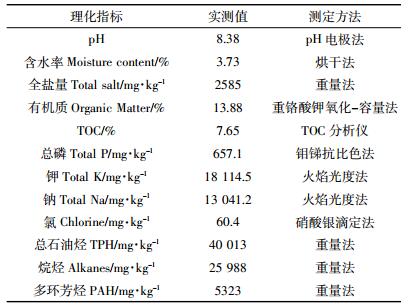
在自然放置的土壤(CK)中,烷烃的降解与多环芳烃浓度、微生物对多环芳烃的代谢活性、多酚氧化酶活性呈显著正相关(相关系数R分别为0.969、0.719、0.901),与微生物对烷烃的代谢活性、脂肪酶活性呈负相关(R分别为-0.474、-0.855);多环芳烃的浓度与土壤微生物对多环芳烃的代谢活性、多酚氧化酶活性正相关(R分别为0.722、0.937),与微生物对烷烃的代谢活性,脂肪酶活性负相关(R分别为-0.610、-0.851)。在自然放置的土壤中,尽管烷烃、多环芳烃的降解与一些生物因素的代谢活性有关,但烷烃、多环芳烃的去除效果较差(图 1)。
在经过生物修复处理的土壤(BAS)中,烷烃的降解与多环芳烃的浓度、总菌数、多酚氧化酶活性、脱氢酶活性正相关(R分别为0.888、0.863、0.978、0.459),与微生物对烷烃的代谢活性、过氧化氢酶、脂肪酶活性负相关(R分别为-0.488、-0.746、-0.892);多环芳烃的降解与总菌数、多酚氧化酶、脱氢酶活性正相关(R分别为0.861、0.876、0.724),与过氧化氢酶、脂肪酶活性负相关(R分别为-0.434、-0.683)。
由于在修复过程中,自然放置的土壤中烷烃、多环芳烃浓度变化不大,而生物修复处理的土壤中烷烃、多环芳烃的去除效率较高(图 1)。因此,在分析影响土壤中烷烃、多环芳烃降解的生物因素时,可以以自然放置土壤的相关性为基准,从经过生物修复处理的土壤中扣除自然放置土壤中的相关性测定结果。通过比较可知,土壤微生物对烷烃的代谢活性、土壤脱氢酶和多酚氧化酶活性变化与烷烃降解存在较强的相关关系,而脱氢酶和多酚氧化酶活性与多环芳烃降解存在较强的相关关系。此外,微生物修复作用可提高土壤中这4种酶活之间的相关性。
3 结论(1) 经过11周的修复,土壤中烷烃、多环芳烃去除率分别为46.8%、39.9%,自然放置的土壤中烷烃、多环芳烃去除率分别为8.8%和5.6%。利用微生物修复技术可有效去除土壤中的不同组分烃。
(2) 微生物修复处理可以改变土壤中多酚氧化酶、脱氢酶、过氧化氢酶、脂肪酶4种酶的活性,土壤中4种酶活之间的相关性增强。土壤微生物对正十六烷烃、多环芳烃的代谢活性显著提高。
(3) 烷烃的降解与微生物对烷烃的代谢活性、土壤脱氢酶和多酚氧化酶活性相关;多环芳烃的生物降解与脱氢酶和多酚氧化酶活性相关。微生物修复作用可提高土壤中这4种酶活与烃代谢活性之间的相关性。
| [1] | 卢晓霞, 李秀利, 马杰, 等. 焦化厂多环芳烃污染土壤的强化微生物修复研究[J]. 环境科学, 2011, 32(3) : 864–869. LU Xiao-xia, LI Xiu-li, MA Jie, et al. Enhanced bioremediation of coking plant soils contaminated with polycyclic aromatic hydrocarbons[J]. Environmental Science, 2011, 32(3) : 864–869. |
| [2] | Chaineau C H, Rougeux G, Yéprémian C, et al. Effects of nutrient concentration on the biodegradation of crude oil and associated microbial populations in the soil[J]. Soil Biology and Biochemistry, 2005, 37(8) : 1490–1497. DOI:10.1016/j.soilbio.2005.01.012 |
| [3] | Miya R K, Firestone M K. Enhanced phenanthrene biodegradation in soil by slender oat root exudates and root debris[J]. Journal of Environmental Quality, 2001, 30(6) : 1911–1918. DOI:10.2134/jeq2001.1911 |
| [4] | 杨茜, 吴蔓莉, 聂麦茜, 等. 石油污染土壤的生物修复技术及微生物生态效应[J]. 环境科学, 2015, 36(5) : 1856–1863. YANG Qian, WU Man-li, NIE Mai-qian, et al. Effects and biological response on bioremediation of petroleum contaminated soil[J]. Environmental Science, 2015, 36(5) : 1856–1863. |
| [5] | Zhou X Q, Chen C R, Wang Y F, et al. Soil extractable carbon and nitrogen, microbial biomass and microbial metabolic activity in response to warming and increased precipitation in a semiarid Inner Mongolian grassland[J]. Geoderma, 2013, 206 : 24–31. DOI:10.1016/j.geoderma.2013.04.020 |
| [6] | 冯庆贤, 陈智宇. 耐高温采油微生物的研究与应用[J]. 石油勘探与开发, 2000, 27(3) : 50–52. FENG Qing-xian, CHEN Zhi-yu. Research and application of high-temperature oil microorganisms[J]. Petroleum Exploration and Development, 2000, 27(3) : 50–52. |
| [7] | Wrenn B A, Venosa A D. Selective enumeration of aromatic and aliphatic hydrocarbon degrading bacteria by a most-probable-number procedure[J]. Canadian Journal of Microbiology, 1996, 42(3) : 252–258. DOI:10.1139/m96-037 |
| [8] | 郑华, 陈法霖, 欧阳志云, 等. 不同森林土壤微生物群落对Biolog-GN板碳源的利用[J]. 环境科学, 2007, 28(5) : 1126–1130. ZHENG Hua, CHEN Fa-lin, OUYANG Zhi-yun, et al. Utilization of different carbon sources types in Biolog-GN microplates by soil microbial communities from four forest types[J]. Environmental Science, 2007, 28(5) : 1126–1130. |
| [9] | 杨永华, 姚健, 华晓梅. 农药污染对土壤微生物群落功能多样性的影响[J]. 微生物学杂志, 2000, 20(2) : 23–25. YANG Yong-hua, YAO Jian, HUA Xiao-mei. Effect of pesticide pollution against functional microbial diversity in soil[J]. Journal of Microbiology, 2000, 20(2) : 23–25. |
| [10] | 周徽, 冯彦房, 薛利红, 等. 自然生物膜对水体中罗丹明B的净化与响应[J]. 环境科学研究, 2014, 27(7) : 726–732. ZHOU Hui, FENG Yan-fang, XUE Li-hong, et al. Purification of rhodamine B in wastewater by periphyton and response of periphyton to dye[J]. Research of Environmental Sciences, 2014, 27(7) : 726–732. |
| [11] | 张燕燕, 曲来叶, 陈利顶. BiologECOPlateTM实验信息提取方法改进[J]. 微生物学通报, 2009, 36(7) : 1083–1091. ZHANG Yan-yan, QU Lai-ye, CHEN Li-ding. An amendment on information extraction of BiologEcoPlateTM[J]. Microbiology, 2009, 36(7) : 1083–1091. |
| [12] | 杨茜, 吴蔓莉, 曹碧霄, 等. 石油降解菌的筛选、降解特性及其与基因的相关性研究[J]. 安全与环境学报, 2014, 14(1) : 187–192. YANG Qian, WU Man-li, CAO Bi-xiao, et al. Isolation of petroleum degrading strains and determination their degrading and gene characteristics[J]. Journal of Safety and Environment, 2014, 14(1) : 187–192. |
| [13] | U.S. Environmental Protection Agency. N-hexane extractable material (HEM; oil and grease) and silica gel treated N-hexane extractable material (SGT-HEM; non-polar material) by extraction and gravimetry[S]. EPA-821-R-98-002, 1999. |
| [14] | 关松荫. 土壤酶及其研究法[M]. 北京: 农业出版社, 1986: 274-300. GUAN Song-yin. Soil enzyme and study method[M]. Beijing: Agricul-tural Press, 1986: 274-300. |
| [15] | Taha M, Kadali K K, Al-Hothaly K, et al. An effective microplate method (Biolog-MT2) for screening native lignocellulosic-straw-degrading bacteria[J]. Annals of Microbiology, 2015, 65(4) : 2053–2064. DOI:10.1007/s13213-015-1044-y |
| [16] | 李广贺, 张旭, 卢晓霞. 土壤残油生物降解性与微生物活性[J]. 地球科学-中国地质大学学报, 2002, 27(2) : 181–185. LI Guang-he, ZHANG Xu, LU Xiao-xia. Biodegradation of residual petrochemicals and microbial activities in polluted soil[J]. Earth Science-Journal of China University of Geosciences, 2002, 27(2) : 181–185. |
| [17] | Liu P W G, Chang T C, Whang L M, et al. Bioremediation of petroleum hydrocarbon contaminated soil:Effects of strategies and microbial community shift[J]. International Biodeterioration & Biodegradation, 2011, 65(8) : 1119–1127. |
| [18] | Margesin R, Zimmerbauer A, Schinner F. Soil lipase activity a usefulindicator of oil biodegradation[J]. Biotechnology Techniques, 1999, 13(12) : 859–863. DOI:10.1023/A:1008928308695 |
| [19] | Dinesh R, Dubey P R, Shyam G P. Soil microbial biomass and enzyme activities as influenced by organic manure incorporation into soils of a rice-rice system[J]. J Agron Crop Sci, 1998, 181(3) : 173–178. DOI:10.1111/j.1439-037X.1998.tb00414.x |
| [20] | 蔺昕, 李培军, 孙铁珩, 等. 石油污染土壤的生物修复与土壤酶活性关系[J]. 生态学杂志, 2005, 24(10) : 1226–1229. LIN Xin, LI Pei-jun, SUN Tie-heng, et al. Bioremediation of petroleum-contaminated soil and its relationship with soil enzyme activities[J]. Chinese Journal of Ecology, 2005, 24(10) : 1226–1229. |
| [21] | Margesin R, Schinner F. Biological decontamination of oil spills in cold environments[J]. Journal of Chemical Technology and Biotechnology, 1999, 74(5) : 381–389. DOI:10.1002/(ISSN)1097-4660 |
| [22] | 范淑秀, 李培军, 巩宗强, 等. 苜蓿对PAHs菲污染土壤的修复作用研究[J]. 环境科学, 2007, 28(9) : 2080–2085. FAN Shu-xiu, LI Pei-jun, GONG Zong-qiang, et al. Study on phytoremediation of phenanthrene-contaminated soil with Alfalfa (Medicago sativa L.)[J]. Chinese Journal of Environment Science, 2007, 28(9) : 2080–2085. |
| [23] | Zou J, Liu X, Zhong C, et al. Effect of palmitic acid on remediation of Scripus triqueter, and enzymes activities of the rhizosphere soil in the simulated diesel-spiked wetland[J]. International Biodeterioration & Biodegradation, 2014, 94 : 109–114. |
| [24] | Gu Y, Wang P, Kong C H. Urease, invertase, dehydrogenase and polyphenoloxidase activities in paddy soil influenced by allelopathic rice variety[J]. European Journal of Soil Biology, 2009, 45(5/6) : 436–441. |
| [25] | Sun T R, Cang L, Wang Q Y, et al. Roles of abiotic losses, microbes, plant roots, and root exudates on phytoremediation of PAHs in a barren soil[J]. Journal of Hazardous Materials, 2010, 176(1) : 919–925. |
 2017, Vol. 36
2017, Vol. 36