文章信息
- 范瑞娟, 郭书海, 李凤梅
- FAN Rui-juan, GUO Shu-hai, LI Feng-mei
- 石油降解菌群的构建及其对混合烃的降解特性
- Construction of petroleum degrading bacteria consortium and its degradation properties of mixed hydrocarbons
- 农业环境科学学报, 2017, 36(3): 522-530
- Journal of Agro-Environment Science, 2017, 36(3): 522-530
- http://dx.doi.org/10.11654/jaes.2016-1266
文章历史
- 收稿日期: 2016-09-30
2. 中国科学院沈阳应用生态研究所, 沈阳 110016
2. Institute of Applied Ecology, Chinese Academy of Sciences, Shenyang 110016, China
在各种石油污染土壤修复方法中,生物修复技术以其操作简单、处理费用低、环境影响小等优点而受到人们的重视。多项研究证明生物修复在脂肪烃和芳香烃的降解中起着重要作用[1-3]。
生物修复效率的高低取决于多方面的因素,如微生物群落结构、污染物类型、营养物质和污染物的生物可利用性,以及土壤理化性质,如pH、温度和水分含量等[4],其中微生物群落组成对石油降解效率和生物利用途径等起着决定性的作用[5]。由于土著微生物降解速度慢,修复周期长,往往需要投加外源性微生物以加速污染物的降解[6]。然而石油是一种组成成分复杂的化合物,其中包含脂肪烃和多环芳烃等多种成分,靠单一的微生物菌种很难实现其完全降解。研究表明,将不同降解菌进行组合作为石油烃的降解微生物,其对石油的降解性能明显高于单菌。Chen等[7]采用Acinetobacter sp. YC-X 2,Kocuria sp. YC-X 4和Kineococcus sp. YC-X 7构建的微生物菌群对稠油污染土壤进行了修复试验,结果表明该混合菌群比单株菌有更好的修复效果。另有研究表明,相对单菌株而言,Mycobacterium sp.和Sphingomonas sp.组合对菲的降解有促进作用[8]。仅仅接种降解菌而不添加营养物质并不能使修复过程顺利进行,必须为微生物提供氮、磷等营养元素,以改善其生长的环境条件,提高微生物的数量、多样性和活性,才可加速污染物的降解[9]。然而有时加入营养物质并不能促进有机污染物的生物降解,需要对氮、磷等营养水平进行适当的调节才有利于生物修复的进行[10]。石油烃可在土壤中形成独立的非水相,不能被微生物直接利用,而使其生物可利用性减小,同时容易产生生物毒害[11]。表面活性剂的加入,可提高石油烃类化合物和其他一些有机化合物的生物可利用性,有助于提高微生物对其利用能力[12]。叶淑红等[13]研究发现,非离子表面活性剂吐温-80对微生物的繁殖和油的降解具有促进作用。
本研究拟从石油污染土壤中筛选具有直链烷烃、环烷烃和芳烃降解能力的高效降解菌,构建微生物降解菌群,并对影响微生物菌群的环境条件进行优化;以正十六烷、环十二烷和芘分别作为直链烷烃、环烷烃和芳烃的代表性污染物,实施污染土壤中总石油烃(TPH)的修复试验。研究结果可为石油烃类污染物的生物降解过程调控提供一定的理论基础。
1 材料与方法 1.1 试验材料 1.1.1 污染物大庆原油,其密度为0.882 g·cm-3(20 ℃),凝固点为25.8 ℃,粘度为18.9 mPa·s-1(50 ℃);以环十二烷(> 99%,东京化成工业株式会社)、正十六烷(>98%,东京化成工业株式会社)、芘(>99%,百灵威科技有限公司)分别作为环烷烃、直链烷烃和芳烃的代表性污染物。
1.1.2 试验土壤石油污染土壤样品采自大庆油田。试验所采用的干净土壤采自沈阳应用生态研究所生态站附近0~30 cm土层,其理化性质为:pH 6.22,有机碳6.43 g·kg-1,阳离子交换量21.25 cmol·kg-1,速效氮73.43 mg·kg-1,速效磷7.23 mg·kg-1。将土壤自然风干后过2 mm筛,再将正十六烷、环十二烷和芘均匀混入土壤中,配制成混合烃污染土壤,用于微生物修复试验。经气相色谱分析,土壤中正十六烷、环十二烷和芘的实际浓度分别为4 744.54、947.22、48.81 mg·kg-1。
1.1.3 培养基的配制牛肉膏蛋白胨培养基的配制参照《微生物学实验(第4版)》[14]。
无机盐培养液(g·L-1):(NH4)2SO4 1.5,NaNO3 1.5,K2HPO4 1,KCl 0.5,MgSO4·7H2O 0.5,FeSO4·7H2O 0.01,pH 7.0。
富集用无机盐培养基:向上述无机盐培养液中加入0.5%的石油,pH 7.0。
烷烃降解菌筛选培养基的配制参考Kuba等[15]的方法;芳烃降解菌筛选培养基参考Alley等[16]的方法。
1.2 试验方法 1.2.1 降解菌的筛选石油降解菌的筛选:称取5 g石油污染土壤,加至100 mL富集用无机盐培养基中,置于恒温振荡培养箱中,30 ℃、150 r·min-1振荡培养7 d,取出5 mL培养液转入新鲜培养基中;相同条件下转接富集培养3次,将培养液梯度稀释后,取0.1 mL涂布于牛肉膏蛋白胨固体培养基平板上,待平板上长出菌落后,挑取不同颜色、不同形态的单菌落,重新接于富集用无机盐培养基中培养;再次进行分离,直至分离出纯菌。
直链烷烃/环烷烃降解菌的筛选:将筛选出的石油降解菌株各挑取一环,分别接种于100 mL直链烷烃/环烷烃降解菌筛选培养基中,置于恒温振荡培养箱中培养(30 ℃,150 r·min-1),待培养液明显浑浊后,取1 mL接种于100 mL新鲜的直链烷烃/环烷烃降解菌筛选培养基中。如此重复三次后,如仍能使培养液变浑浊,则为直链烷烃/环烷烃降解菌[17]。
芳烃降解菌的筛选:采用平板升华法[18]。
1.2.2 单株菌对TPH降解能力分析将筛选出的各菌株进行活化,挑取一环接种于100 mL牛肉膏蛋白胨液体培养基中,30 ℃、150 r·min-1振荡培养24 h,取6 mL培养液至富集用无机盐培养基中,30 ℃、150 r·min-1培养5 d,用二氯甲烷萃取培养基中残留污染物3次,合并提取液。将漏斗中塞入脱脂棉,上面放置5~6勺已烘干的无水硫酸钠(400 ℃,2~3 h),将提取液进行过滤脱水,采用旋转蒸发仪蒸干溶剂后,再用石油醚溶解定容至25 mL,采用紫外分光光度法测定225 nm处吸光值,带入标准曲线得到残油含量,计算石油去除率。以同样条件下不接菌的处理作为对照。
1.2.3 混合菌液与单菌液降解能力对比分析将筛选出的具有直链烷烃、环烷烃和芳烃降解能力的菌株活化后,挑取一环接种于100 mL牛肉膏蛋白胨液体培养基中,30 ℃、150 r·min-1振荡培养24 h,将各菌液按同比例混合制成混合菌液,分别取混合菌液和单菌液6 mL加至富集用无机盐培养基中,30 ℃、150 r·min-1振荡培养5 d,萃取培养液中残留石油,计算石油去除率,对比混合菌液与单菌液降解能力。以同样条件下不接菌的处理为对照。
1.2.4 混合菌群的构建及其降解能力分析按1.2.3中的方法培养所筛选出的具有直链烷烃、环烷烃和芳烃降解能力的菌株,采用正交试验法,将各菌株按不同比例混合,取6 mL培养液加至富集用无机盐培养基中,30 ℃、150 r·min-1培养5 d,萃取培养液中残留石油,计算石油去除率,分析混合菌群的最佳配比。
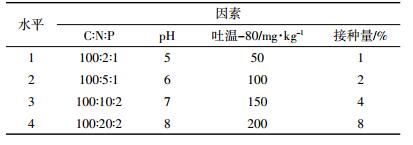
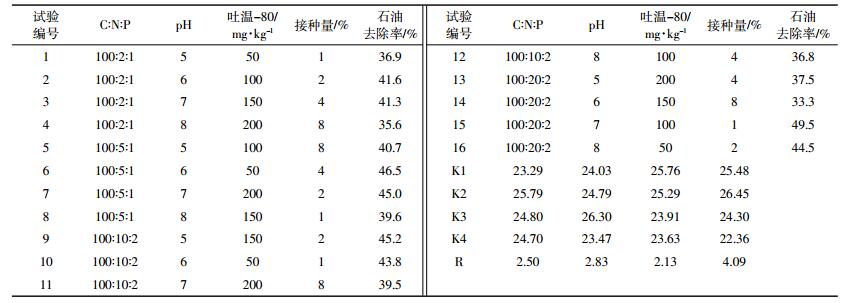
1.2.5 石油降解菌群影响因素分析选择C:N:P、pH、表面活性剂以及微生物接种量4个因素,按表 1设计正交试验,分析微生物菌群最佳降解条件。称取10 g人工配制的石油污染土壤(石油浓度为5%),加入到装有30 mL灭菌水的三角瓶中,调节不同因素水平,30 ℃、150 r·min-1培养5 d,检测残余石油量,计算其去除率。
称取混合烃污染土壤2 kg,将选出的菌株进行培养,按1.2.4中确定的比例混合、离心,再按1.2.5中确定的比例投加至污染土壤中,根据1.2.5的条件,调节土壤C:N:P比例、pH和表面活性剂的量,其中N由NaNO3和(NH4)2SO4提供(1:1),P由K2HPO4提供。用蒸馏水均匀喷洒土壤并不断翻动搅拌,使土壤的湿度达到16%~19%(W:W)。将土壤分层铺放在土壤塑料盒内,每10 d进行采样分析,试验共进行100 d。试验在恒温(25±1)℃下进行,并定期向土壤箱中喷洒蒸馏水以保持土壤湿度。在相同条件下与未投加微生物和条件优化的土壤进行对照。
1.3 分析方法 1.3.1 土壤中正十六烷、环十二烷以及芘含量的测定将土样风干后研磨,过40目筛,称取10 g于250 mL磨口三角瓶中,加入30 mL二氯甲烷和丙酮混合溶剂(1:1),170 r·min-1振荡提取30 min后再超声萃取10 min,将提取液转入50 mL离心管中,8000 r·min-1低温离心2 min,收集上清液,其沉淀物再用30 mL二氯甲烷和丙酮混合溶剂以同样的方法提取三次,合并提取液。将提取液用旋转蒸发仪浓缩至干,用正己烷溶解并定容至5 mL,用0.22 μm有机滤膜过滤后,采用气相色谱法测定正十六烷、环十二烷和芘的含量。
工作条件和升温程序:初始温度80 ℃,保持1 min,以20 ℃·min-1升至300 ℃,保持5 min;进样口温度为250 ℃,检测器温度为300 ℃;所用检测器为氢火焰离子检测器;色谱柱为非极性毛细管柱(TR-1 MS,30 m×0.25 mm×0.25 μm);载气为99.99%高纯氮,流量1.0 mL·min-1;不分流进样,进样量为1 μL。
1.3.2 菌株鉴定将筛选出的高效菌株交由测序公司进行16S rRNA序列测定,将序列信息输入NCBI(www.ncbi.nlm.nih.gov)数据库进行Blast分析,根据同源性比对结果初步鉴定到属。
1.3.3 微生物群落变化微生物群落采用PCR-DGGE法进行分析[19]。
土壤基因组DNA采用快速提取试剂盒提取(MP Biomedicals,LCC.,Ohio,美国)。16S rRNA经PCR(MJ Research Inc.,Waltham,MA,美国)扩增后用于后续的DGGE分析。所用引物为GC-341F(5′-CGCCCGCCGCGCGCGGCGGGCGGGGCGGGGGCACGGGGGGC-CTACGGGAGGCAGCAG-3′)及907R(5′-CCGTCA-ATTCCTTTRAGTTT-3′)。
采用Dcode基因突变检测系统(Bio-Rad)对PCR反应产物进行分析。DGGE电泳所用胶浓度为6%聚丙烯酰胺,变性剂梯度范围40%~60%,60 ℃条件下,70 V运行16 h,电泳完成后用0.01% Genefinder染色45 min。用凝胶成像系统捕获凝胶数字图象,并用图形分析软件Quantity One 4.4.0进行图像分析,各泳道图谱的相似性通过计算Dice系数进行比较,聚类树状图用非加权配对算术平均法(UPGMA)生成。泳道光密度曲线上的峰面积代表条带密度,每个条带密度所占比例用来比较各种群的丰度。采用香侬指数(H′)[20]来表示微生物多样性:
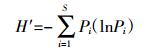
式中:Pi为DGGE图谱中第i条带的峰密度与所有条带(S)总峰密度的比值。
1.3.4 脱氢酶活性脱氢酶活性根据TTC还原法[21]测定。
1.3.5 可培养微生物数量变化可培养微生物数量分析采用平板菌落计数法[22]。
2 结果与讨论 2.1 石油降解菌群的构建 2.1.1 石油降解菌的筛选经过富集、分离和纯化,从石油污染土壤中共分离得到10株降解菌B1~B10。采用选择性培养基进一步筛选可知,10株菌均能在直链烷烃降解菌筛选培养基中生长,表明其均能以直链烷烃为唯一碳源生长;菌B1、B2、B3、B6、B7和B9能以环烷烃为唯一碳源生长;菌B2和B3能在芳烃降解菌筛选培养基上生长并产生透明圈,表明其能利用多环芳烃。
2.1.2 单株菌对TPH的降解能力单株菌对TPH的降解能力如图 1所示。菌株B2和B3对TPH的降解能力尤为突出,5 d后对TPH的去除率分别达75%和72%;其他菌株降解能力大小依次为B9、B1、B7、B6、B5、B10、B8和B4。综合分析表明,菌株B1、B2、B3、B6、B7和B9对TPH具有较高的降解能力,而且B2和B3对直链烷烃、环烷烃和芳烃均具有降解能力,B1、B6、B7和B9对直链烷烃和环烷烃具有降解能力,故选择菌株B1、B2、B3、B6、B7和B9构建微生物混合菌群。
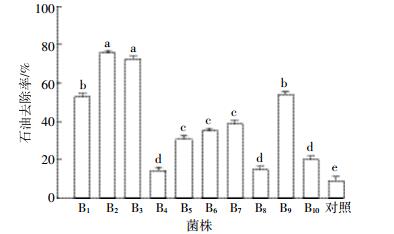
|
| 图 1 单株菌对TPH的降解能力 Figure 1 Removal rate of TPH by different degrading strains |
采用16S rRNA基因序列对比,结果如表 2所示。对六株高效菌株B1、B2、B3、B6、B7和B9进行鉴定,结果表明,该六株菌归为5个不同的属,其中:B1与嗜氢菌属(Hydrogenophaga sp.)多个菌种基因序列相似性达99%以上;B2与苯基杆菌属(Phenylobacterium sp.)多种基因序列相似性达98%以上;B3和B9与节杆菌属(Arthrobacter sp.)多个菌种基因序列相似性达99%以上;B6与鞘脂菌属(Sphingobium sp.)多种基因序列相似性达到99%以上;B7与芽胞杆菌属(Bacillus sp.)多个菌种基因序列相似性达99%以上。因此,初步鉴定B1、B2、B6和B7分别属于嗜氢菌属(Hydrogenophaga sp.)、苯基杆菌属(Phenylobacterium sp.)、鞘脂菌属(Sphingobium sp.)和芽胞杆菌属(Bacillus sp.);B3和B9属于节杆菌属(Arthrobacter sp.)。目前,已在石油烃类污染土壤中检测到多种嗜氢菌属、苯基杆菌属、鞘脂菌属、芽胞杆菌属和节杆菌属的微生物,已发现的菌株不仅可以降解烷烃、多环芳烃,还可以降解混合烃[23-26]。
由B1、B2、B3、B6、B7和B9构成的混合菌株与各单菌株对TPH的去除能力如图 2所示。5 d后,六种单菌体系中,B2对石油的去除率最高,为65.7%,B6对石油的去除率最低,为25.1%。接种复合菌液,石油的去除率为77.9%,比单菌体系的最高去除率提高了12.2%。石油组成成分复杂,其中包含脂肪烃和多环芳烃等多种成分,靠单一的微生物菌种很难实现其全面降解。该结果表明,将具有不同去除能力的菌种进行混合,作为石油烃的降解微生物,其对石油的降解性能明显高于单菌。
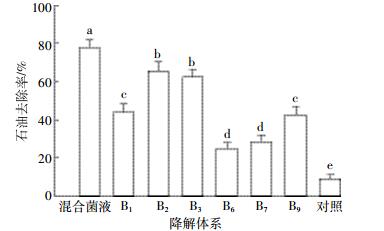
|
| 图 2 混合菌株与单菌株对TPH的去除能力 Figure 2 Removal rate of TPH by mixed bacteria and single degrading strains |
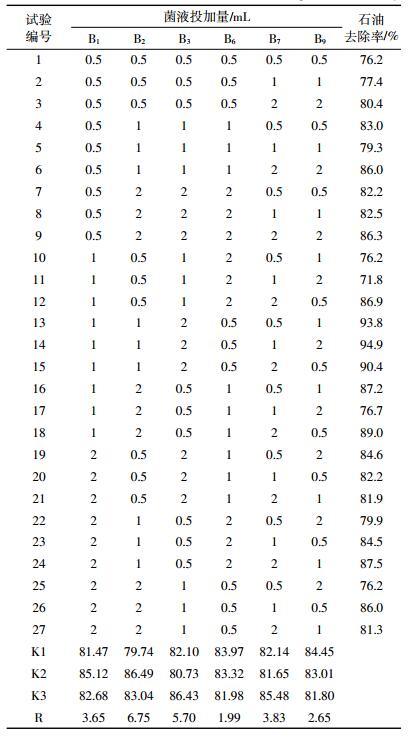
根据六因素三水平正交试验L27(36)对所选六株菌投加比例进行优化。不同投加比例下各菌群对TPH的降解能力如表 3所示。从极差分析结果可知,所选出的六株菌中,B2对混合菌群降解TPH的影响最大,然后依次为B3、B7、B1、B9和B6。菌株B2和B3对环烷烃、直链烷烃和芳烃均具有降解能力,而且菌株B2和B3对TPH的降解能力明显高于其他菌株。这可能是B2和B3菌株对混合菌群降解TPH影响最大的主要原因。
直观分析表明,14号试验中石油去除率最高,为94.9%。将六株菌三个水平处理下的石油平均去除率用图 3表示,发现B1:B2:B3:B6:B7:B9=1:1:2:0.5:2:0.5时石油去除率应为最高(对应于15号试验),而15号试验中TPH去除率为90.4%,低于14号试验。因此,再次以14号和15号配比进行降解条件的验证试验,结果表明,5 d后,14号试验中TPH去除率为95.6%,15号试验中TPH去除率为91.5%。综合分析,最佳的菌株投加比例应为B1:B2:B3:B6:B7:B9=1:1:2:0.5:1:2。不同投加比例的菌群对石油降解能力存在差异的原因可能是菌群中各菌株在生长过程中存在既相互影响又相互制约的关系,当各菌株的数量达到一定比例时才能发挥最大的优势[27]。
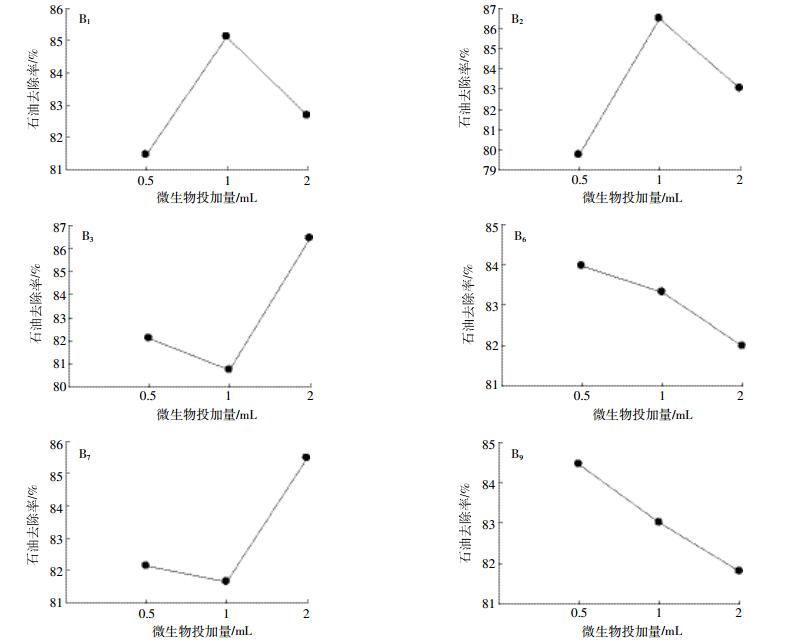
|
| 图 3 不同水平处理下石油烃平均去除率 Figure 3 Average removal rate of petroleum hydrocarbons under different treatment levels |
根据四因素四水平正交试验L16(44)对C:N:P、pH、表面活性剂以及微生物接种量等进行优化,结果如表 4所示。从极差分析结果可知,影响石油降解的四个因素中,接种量对石油降解效果影响最大,其后依次为pH、C:N:P和表面活性剂添加量。
直观分析表明,15号试验中石油去除率最高,达到49.5%。将各因素不同水平处理下石油烃的平均去除率用图 4表示,发现当C:N:P比例为100:5:1、pH为7、吐温-80添加量为50 mg·kg-1、微生物降解菌群接种量为2%时,石油去除率应为最高,而这一配比并未出现在正交表中。因此,以该配比进行降解条件的验证试验,结果石油去除率达58.3%,高于正交试验中的石油去除率,说明该配比为最优组合。综合考虑土壤中污染物、有机碳、速效氮和速效磷的浓度,通过调整使最终C:N:P比例为10:5:1,即NaNO3和(NH4)2SO4(1:1)用量为6.01 g·kg-1,K2HPO4的用量为1.21 g·kg-1。

|
| 图 4 各因素不同水平处理下石油烃平均去除率 Figure 4 Average removal rate of petroleum hydrocarbons under different treatment levels of each factor |
土壤中微生物群落变化如图 5所示。根据聚类分析(图 5b),0 d各自聚为一类,10 d和40 d聚为一类,而70 d和100 d聚为一类。根据多样性分析(图 5a),在试验进行的前10 d,土样中微生物多样性明显升高,可能的原因是微生物混合菌与氮磷营养物的添加使土壤降解菌多样性迅速上升。70 d后多样性有所降低,但100 d后,土样中微生物多样性仍高于初始值,说明所优化的土壤环境适宜微生物的生长,使其保持良好的多样性。

|
| 图 5 土壤微生物群落变化(a)及聚类分析(b) Figure 5 Microbial community shifts (a) and cluster analysis (b) in soil |
微生物活性和可培养微生物数量随时间变化情况如图 6所示。试验过程中,微生物活性和数量均呈先上升后下降的趋势,与微生物多样性变化趋势一致。在试验进行的前10 d,微生物活性和数量显著升高(P < 0.01),之后则有所降低。试验初期向土壤中添加了营养物质,使土壤中营养物质充足,且C:N:P比例符合微生物生长的需要,故微生物活性和数量迅速上升,而后期随着营养物质的消耗和一些有毒中间产物的产生,微生物活性和数量降低[28]。
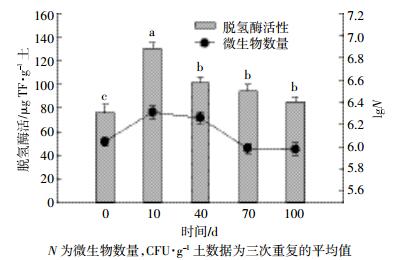
|
| 图 6 微生物活性及可培养微生物数量变化 Figure 6 Changes of culturable bacterial numbers and dehydrogenase activity |
土壤中正十六烷、环十二烷和芘的含量随时间变化情况如图 7所示。40 d后,土壤中环十二烷含量由初始的947.22 mg·kg-1降至140.89 mg·kg-1,去除率达85.1%,对照组去除率仅为10.6%(图 7a)。经过100 d的修复,土壤中的正十六烷含量由初始的4 744.54 mg·kg-1降至1 585.37 mg·kg-1,去除率达66.6%,而对照组去除率仅为9.7%(图 7b)。100 d后,土壤中芘的含量由48.81 mg·kg-1降至28.91 mg·kg-1,去除率达71.8%,对照组去除率为11.4%(图 7c)。
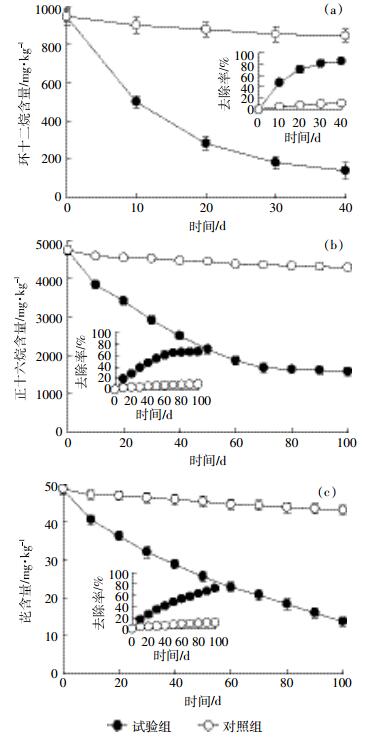
|
| 图 7 土壤中环十二烷(a)、正十六烷(b)和芘(c)含量变化 Figure 7 Changes of the contents of cyclododecane (a), n-hexadecane (b) and pyrene (c) in soil |
该菌群对土壤中三类烃的去除均具有明显的促进作用。石油组成成分复杂,靠单一的微生物菌种很难实现其完全降解。本研究构建了包含环烷烃、直链烷烃和多环芳烃降解能力的降解菌群,而由于不同的微生物具有不同的代谢途径[29],该菌群综合了各菌株在降解不同烃类组分的过程中所体现出的优势,实现了石油烃类污染物的高效降解。
在试验进行的前10 d,各类污染物降解速率均最快,后期则逐渐降低,与土壤微生物多样性、数量以及活性变化趋势一致。试验初期,微生物混合菌与营养物的投加解决了土壤中营养缺乏与微生物活性低下的问题,故降解菌数量、活性与污染物去除率均迅速上升,而随着微生物多样性、数量和活性的降低,污染物去除率也随之下降。据此,可根据微生物活性和污染物降解速率的变化趋势,寻找污染物降解节点,适时调节微生物群落和活性,使污染物保持较高的降解速率。另外,试验初期,土壤中积累的中间产物较少,故污染物减少速率较大,而随着中间产物的积累,不但要进行环十二烷、正十六烷和芘的转化,还要进行中间产物的进一步氧化,使得污染物减少速率降低[30]。该研究结果可为石油烃类污染物的生物降解过程调控提供一定的理论依据。
3 结论从大庆油田石油污染土壤中筛选得到6株分别具有直链烷烃、环烷烃和芳烃降解能力的菌株,通过对接种比例、接种量的优化,构建了微生物降解菌群,并对其环境条件进行了优化,所优化的土壤环境适宜微生物的生长。该菌群对去除土壤中由环十二烷、正十六烷和芘所构成的混合烃类污染物具有明显的促进作用,各类污染物的降解速率均呈现先高后低的趋势,与微生物多样性、数量以及活性变化趋势相似。该研究结果有望为石油烃类污染物的生物降解过程调控提供一定的理论基础。
| [1] | Lladó S, Covino S, Solanas A, et al. Comparative assessment of bioremediation approaches to highly recalcitrant PAH degradation in a real industrial polluted soil[J]. Journal of Hazardous Materials, 2013, 248/249(6): 407–414. |
| [2] | Moliterni E, Rodriguez L, Fernández F J, et al. Feasibility of different bioremediation strategies for treatment of clayey and silty soils recently polluted with diesel hydrocarbons[J]. Water, Air & Soil Pollution, 2012, 223(5): 2473–2482. |
| [3] | 李凤梅, 郭书海, 张灿灿, 等. 多环芳烃降解菌的筛选及其在焦化场地污染土壤修复中的应用[J]. 环境污染与防治, 2016, 38(4): 1–5. LI Feng-mei, GUO Shu-hai, ZHANG Can-can, et al. Isolation of PAHs degrading bacteria and its application to mediation of pulluted soil in coking site[J]. Environmental Pollution & Control, 2016, 38(4): 1–5. |
| [4] | Boopathy R. Factors limiting bioremediation technologies[J]. Bioresource Technology, 2000, 74(1): 63–67. DOI:10.1016/S0960-8524(99)00144-3 |
| [5] | Grace Liu P W, Chang T C, Whang L M, et al. Bioremediation of petroleum hydrocarbon contaminated soil:Effects of strategies and microbial community shift[J]. International Biodeterioration & Biodegradation, 2011, 65(8): 1119–1127. |
| [6] | Reddy M V, Devi M P, Chandrasekhar K, et al. Aerobic remediation of petroleum sludge through soil supplementation:Microbial community analysis[J]. Journal of Hazardous Materials, 2011, 197(24): 80–87. |
| [7] | Chen J, Yang Q Y, Huang T P, et al. Enhanced bioremediation of soil contaminated with viscous oil through microbial consortium construction and ultraviolet mutation[J]. World Journal of Microbiology and Biotechnology, 2011, 27(6): 1381–1389. DOI:10.1007/s11274-010-0589-y |
| [8] | Chen J L, Au K C, Wong Y S, et al. Using orthogonal design to determine optimal conditions for biodegradation of phenanthrene in mangrove sediment slurry[J]. Journal of Hazardous Materials, 2010, 176(1): 666–671. |
| [9] | 乔俊, 陈威, 张承东. 添加不同营养助剂对石油污染土壤生物修复的影响[J]. 环境化学, 2010, 29(1): 6–11. QIAO Jun, CHEN Wei, ZHANG Cheng-dong. Bioremediation of petroleum contaminated soil by various nutrient amendments[J]. Environmental Chemistry, 2010, 29(1): 6–11. |
| [10] | 钟毅, 李广贺, 张旭, 等. 污染土壤石油生物降解与调控效应研究[J]. 地学前缘, 2006, 13(1): 128–133. ZHONG Yi, LI Guang-he, ZHANG Xu, et al. A study of the bioremediation effects on the petroleum contaminated soil[J]. Earth Science Frontiers, 2006, 13(1): 128–133. |
| [11] | McCray J E, Bai G, Maier R M, et al. Biosurfactant-enhanced solubilization of NAPL mixtures[J]. Journal of Contaminant Hydrology, 2001, 48(1): 45–68. |
| [12] | Bueno-Montes M, Springael D, Ortega-Calvo J J. Effect of a nonionic surfactant on biodegradation of slowly desorbing PAHs in contaminated soils[J]. Environmental Science & Technology, 2011, 45(7): 3019–3026. |
| [13] | 叶淑红, 丁鸣, 马达, 等. 微生物修复辽东湾油污染湿地研究[J]. 环境科学, 2005, 26(5): 143–146. YE Shu-hong, DING Ming, MA Da, et al. Research of Microbio-remediation of oil-contaminated wetland in Liaodong Bay[J]. Environmental Science, 2005, 26(5): 143–146. |
| [14] | 沈萍, 陈向东. 微生物学实验[M]. 四版. 北京: 高等教育出版社, 2010. SHEN Ping, CHEN Xiang-dong. Experiment of microbiology[M]. 4th Edition. Beijing: Higher Education Press, 2010. |
| [15] | Kuba T, Loosdrecht M C M V, Heijnen J J. Phosphorus and nitrogen removal with minimal COD requirement by integration of denitrifying dephosphatation and nitrification in a two-sludge system[J]. Water Research, 1996, 30(7): 1702–1710. DOI:10.1016/0043-1354(96)00050-4 |
| [16] | Alley J F, Brown L R. Use of sublimation to prepare solid microbial media with water-insoluble substrates[J]. Applied & Environmental Microbiology, 2000, 66(1): 439–442. |
| [17] | 李大平, 李福德. LHG1菌株的分离和降解环己烷的研究[J]. 应用与环境生物学报, 1999, 5(S1): 136–138. LI Da-ping, LI Fu-de. Isolation of strain LHG1 and its biodegradation of cyclohexane[J]. Chinese Journal of Applied and Environmental Biology, 1999, 5(S1): 136–138. |
| [18] | 高野萌, 杨雪莲, 李凤梅, 等. 高环多环芳烃降解菌的筛选及其降解特性[J]. 生态学杂志, 2016, 35(6): 1539–1546. GAO Ye-meng, YANG Xue-lian, LI Feng-mei, et al. Isolation of heavy PAH-degrading bacteria and their characteristics of degradation[J]. Chinese of Journal of Ecology, 2016, 35(6): 1539–1546. |
| [19] | Muyzer G, De Waal E C, Uitterlinden A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695–700. |
| [20] | Lear G, Harbottle M J, van der Gast C J, et al. The effect of electrokinetics on soil microbial communities[J]. Soil Biology and Biochemistry, 2004, 36(11): 1751–1760. DOI:10.1016/j.soilbio.2004.04.032 |
| [21] | Megharaj M, Singleton I, McClure N, et al. Influence of petroleum hydrocarbon contamination on microalgae and microbial activities in a long-term contaminated soil[J]. Archives of Environmental Contamination and Toxicology, 2000, 38(4): 439–445. DOI:10.1007/s002449910058 |
| [22] | Randall J D, Hemmingsen B B. Evaluation of mineral agar plates for the enumeration of hydrocarbon-degrading bacteria[J]. Journal of Microbiological Methods, 1994, 20(2): 103–113. DOI:10.1016/0167-7012(94)90013-2 |
| [23] | Borah D, Yadav R N S. Biodegradation of complex hydrocarbon by a novel Bacillus cereus strain[J]. Journal of Environmental Science & Technology, 2014, 7(7): 176–184. |
| [24] | Li F, Zhu L Z, Wang L W, et al. Gene expression of an Arthrobacter in surfactant-enhanced biodegradation of a hydrophobic organic compound[J]. Environmental Science & Technology, 2015, 49(6): 3698–3704. |
| [25] | Alkindi S, Rmm A. Effect of biostimulation using sewage sludge, soybean meal, and wheat straw on oil degradation and bacterial community composition in a contaminated desert soil[J]. Frontiers in Microbiology, 2016, 7(2836): 240. |
| [26] | Martin F, Torelli S, Paslier D L, et al. Betaproteobacteria dominance and diversity shifts in the bacterial community of a PAH-contaminated soil exposed to phenanthrene[J]. Environmental Pollution, 2012, 162: 345–353. DOI:10.1016/j.envpol.2011.11.032 |
| [27] | 李宝明, 姜瑞波. 营养和环境条件对微生物菌群降解石油的影响[J]. 中国土壤与肥料, 2008(3): 78–82. LI Bao-ming, JIANG Rui-bo. Effects of nutrition and environment on oil biodegradation by bacterial community[J]. Soil and Fertilizer Sciences in China, 2008(3): 78–82. |
| [28] | Chu H Y, Lin X G, Fujii T, et al. Soil microbial biomass, dehydrogenase activity, bacterial community structure in response to long-term fertilizer management[J]. Soil Biology and Biochemistry, 2007, 39(11): 2971–2976. DOI:10.1016/j.soilbio.2007.05.031 |
| [29] | 于慧敏, 马玉超. 工业微生物代谢途径调控的基因敲除策略[J]. 生物工程学报, 2010, 26(9): 1199–1208. YU Hui-min, MA Yu-chao. Gene knockout strategies for metabolic pathway regulation in industrial microbes[J]. Chinese Journal of Biotechnology, 2010, 26(9): 1199–1208. |
| [30] | Guo S H, Fan R J, Li T T, et al. Synergistic effects of bioremediation and electrokinetics in the remediation of petroleum-contaminated soil[J]. Chemosphere, 2014, 109: 226–233. DOI:10.1016/j.chemosphere.2014.02.007 |
 2017, Vol. 36
2017, Vol. 36