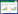文章信息
- 全志星, 田佳源, 张思宇, 皇甫超河, 刘红梅, 杨殿林, 常泓, 王慧
- QUAN Zhi-xing, TIAN Jia-yuan, ZHANG Si-yu, HUANGFU Chao-he, LIU Hong-mei, YANG Dian-lin, CHANG Hong, WANG Hui
- 不同入侵地区黄顶菊DNA表观遗传多样性变化特征
- Studies on epigenetic diversity variation characteristics of Flaveria bidentis genome DNA of four different geographical distributions
- 农业环境科学学报, 2017, 36(4): 625-634
- Journal of Agro-Environment Science, 2017, 36(4): 625-634
- http://dx.doi.org/10.11654/jaes.2016-1245
文章历史
- 收稿日期: 2016-09-26
2. 农业部环境保护科研监测所, 天津 300191
2. Agro-Enviromental Protection Institute, Ministry of Agriculture, Tianjin 300191, China
黄顶菊 (Flaveria bidentis) 又称二齿黄菊, 属于菊科堆心菊族黄菊属, 一年生草本植物, 原产于南美洲, 是近十年来发现的一种新型外来入侵植物[1]。2001年在天津市和河北省衡水湖先后被发现, 后快速扩散。2008年河北省黄顶菊入侵现象尤为严重, 入侵面积高达2万hm2, 占领该省的70多个县城[2]; 2006-2008年天津市黄顶菊入侵面积由200 hm2扩散到了277.6 hm2[3]。黄顶菊具有极强耐盐碱和耐干旱的特性, 依靠其强大的繁殖能力、易于扩散等特点扰乱生态系统原有的食物链和能量秩序, 已被列入《中华人民共和国进境植物检疫性有害生物名录》[4]。
表观遗传学是指基于DNA或组蛋白上的共价修饰而非基因序列改变导致的基因表达水平的变化, 这种变化具有遗传性且遗传是可逆的[5]。研究表明不同地区生长的植物种群间, 存在较大的表观基因组差异[6]。植物利用基因组的表观修饰, 改变相同基因型个体的基因表达式样, 通过表型可塑性来响应环境条件的变化。
DNA甲基化是最重要的表观遗传修饰方式之一, 也是目前机制研究最为透彻的表观遗传过程[7-9]。Angers等[10]发现, 环境条件能够诱导DNA甲基化变异, 且环境诱导的DNA甲基化和表观变异具有持续性和可遗传性。对于植物而言, 气候条件、土壤理化性质等可以诱导其表观遗传变异。潘丽娜[11]发现拟南芥通过表观遗传修饰能更好地适应环境中各种胁迫 (高盐、干旱、高温)。韩雅楠等[12]发现高盐渍土壤使碱蓬和蒙古黄芪的CCGG位点发生大量的去甲基化现象。基因组DNA甲基化的改变很有可能是调控入侵植物生境适应能力的重要机制之一, 然而目前对黄顶菊基因组DNA甲基化的分布模式、变异情况等还缺乏全面的认识。入侵植物黄顶菊DNA表观遗传多样性是否受地理分布区域及入侵生境条件的影响, 尚不清楚。Reyna-Lopez等[13]在1997年首次报道MSAP技术, MSAP技术是在AFLP基础上衍生的基于PCR扩增多态性的DNA甲基化检测方法。该技术的优点在于无需知道被测DNA序列信息, 通过两组酶对CCGG酶切位点敏感程度不同, 就可以检测出样品中大量的甲基化位点, 因而被广泛应用于植物DNA甲基化水平的研究。目前对黄顶菊基因组序列信息没有全面的了解, 故通过甲基化MSAP技术, 研究不同地理分布区域黄顶菊种群间的表观遗传多样性, 可为探索黄顶菊生态适应性获得的表观遗传机制提供理论基础。
1 材料和方法 1.1 植物样品采集2015年9月5-7日分别从四个地区——河北省邯郸市永年县 (HDY)、河北省沧州市献县 (CZX)、河北省衡水市衡水湖 (HSH) 和天津市静海县 (TJJ), 采集处于营养盛期黄顶菊的第3对完全展开叶片, 采集的个体之间距离不低于50 m, 每个地区采集15个个体。将采集叶片用锡箔纸包裹放入液氮中速冻, 随后转入-70 ℃冰箱贮存。在黄顶菊叶片采集处就地采集四个典型入侵地区的土壤各15份, 并用塑封袋封装, 随后转入-70 ℃冰箱冰冻保存。通过实验测定土壤的理化指标, 重复5次并剔除差异较大的数据, 取其平均值。黄顶菊采集地基本概况见表 1。
黄顶菊叶片基因组DNA采用改良CTAB法提取, 参照Wang等[14]方法提取DNA并做进一步改进:在液氮中充分碾磨的植物样品加入65 ℃预热的CTAB裂解液[2% CTAB (W/V), 0.2 mmol·L-1 Tris-CL (pH 8.0), 0.05 mmol·L-1 EDTA (pH 8.0), 1.4 mmol·L-1 NaCl, 2% β-巯基乙醇 (V/V) 1 mL, 1% PVP-40 (W/V)], 等体积氯仿抽提上清液后, 用3 mmol·L-1 KAc和异丙醇沉淀DNA, 最后经无水乙醇漂洗, 加入1 mmol·L-1 NaCl溶解DNA并放入4 ℃冰箱保存。
1.3 MSAP体系建立与优化MSAP分析方法参照梁宏伟等[15]方法并对酶切、连接、预扩增、选扩增进行一定的优化调整。酶切连接反应体系 (20 μL) 组成成分:10×T4 DNA ligase buffer 2.5 μL, EcoRⅠadaptor (5 μmol·L-1) 1 μL, HpaⅡ/MSPⅠadaptor (50 μmol·L-1) 1 μL, BSA (1 mg·L-1) 1 μL, HpaⅡ (或MSPⅠ) 1 U, EcoRⅠ0.5 U, T4 DNA ligase 0.5 U, 模板DNA 500 ng, 加ddH2O补至20 μL。将上述溶液混匀后置入PCR仪, 反应条件为:37 ℃酶切6 h, 接着16 ℃连接8 h, 最后65 ℃ 10 min, 4 ℃冰箱保存备用。
预扩增反应体系 (25 μL) 组成成分:酶切连接产物 (稀释10倍) 2.0 μL, dNTPs (2.5 μmol·L-1) 1.0 μL, 10×PCR buffer 2.5 μL, 引物pre-E-A (5 μmol·L-1)1 μL, 引物pre-H/M-T (5 μmol·L-1)1 μL, Taq DNA polymerase 0.5 U, 加ddH2O补至25 μL。将上述溶液混匀后置入PCR仪, 反应条件为94 ℃预变性5 min, 接着进行30个循环, 每个循环包括94 ℃ 30 s, 56 ℃ 60 s, 72 ℃ 60 s, 最后72 ℃ 10 min, 4 ℃冰箱保存备用。
选择扩增体系的组成成分除引物3'末端添加2个选择性碱基外, 其余与预扩增反应体系相同。反应条件为:94 ℃预变性5 min, 以94 ℃ 30 s、65 ℃ 60 s、72 ℃ 60 s为一个循环, 之后每个循环复性温度递减0.7 ℃共扩增13个循环, 接着进行23个循环, 每个循环包括94 ℃ 45 s、55 ℃ 45 s、72 ℃ 90 s, 最后72 ℃ 10 min, 4 ℃冰箱保存备用。接头和引物序列见表 2。
配置5%聚丙烯酰胺凝胶电泳[16] (尿素33.6 g, 5×TBE 16 mL, 40%丙烯酰胺贮液10 mL, TEMED 75 μL, 10% APS 320 μL, 补水至50 mL), 对选择性扩增产物进行进一步分离。制作好的凝胶板组装放入含有1xTBE的电泳槽中, 预电泳1 h使胶面温度达到55 ℃时上样, 55 W恒功率下电泳2 h。电泳结束后, 剥离两块玻璃板, 将长玻璃板上的电泳条带进行固定、银染和显色, 清水冲洗后自然晾干, 扫描并进行条带计数和分析。
1.5 数据统计与分析利用Quantity One软件标记出5%聚丙烯酰胺凝胶电泳图谱中100~500 bp区间扩增出来的条带, 其中有DNA条带记为1, 无DNA条带记为0, 转化成MSAP的表型数据0/1矩阵。利用POPGene软件分析样品的多样性指数。通过NTsys软件将0/1矩阵转化成相应的MSP (甲基化敏感位点) 和MISP (甲基化不敏感位点) 聚类图谱。用SPSS软件进行不同地区土壤环境因子与黄顶菊甲基化水平的相关性分析。
2 结果与分析 2.1 黄顶菊MSAP体系的建立MSAP技术对基因组DNA质量要求很高, 模板DNA的纯度直接影响后续酶切、扩增以及MSAP谱带的稳定性。为满足MSAP实验要求, 采用改良的CTAB法提取黄顶菊叶片基因组DNA, 用蛋白核酸定量测定仪检测DNA浓度, 要求模板DNA OD260/280在1.7~1.9范围内, 用1%琼脂糖凝胶电泳检测, 要求基因组DNA条带清晰、无降解、无杂质, 即可用于甲基化MSAP检测。基因组DNA凝胶电泳图见图 1。MSAP体系的建立包括:酶切连接反应、预扩增反应和选择性扩增反应。
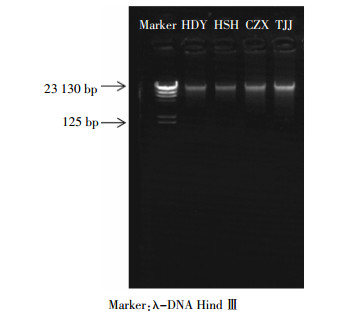
|
| 图 1 黄顶菊基因组DNA提取 Figure 1 Extraction of the Flaveria bidentis genomic DNA |
随机选取10个黄顶菊单株样本做模板进行独立重复实验, 通过对144对引物组合进行筛选, 确定13对扩增效果较好的引物组合。利用13对引物组合扩增四个入侵地区共40个黄顶菊单株, 40个黄顶菊单株部分引物的MSAP图谱如图 2所示。本研究只统计片段长度在100~500 bp范围内的片段。部分泳道的量化分析如图 3所示。以第1泳道为参照, 其余泳道扩增相同分子量的条带相似比, 第7泳道与第1泳道扩增条带的相似度最高, 为77%, 而第25泳道与其相似度最低, 为14.1%。
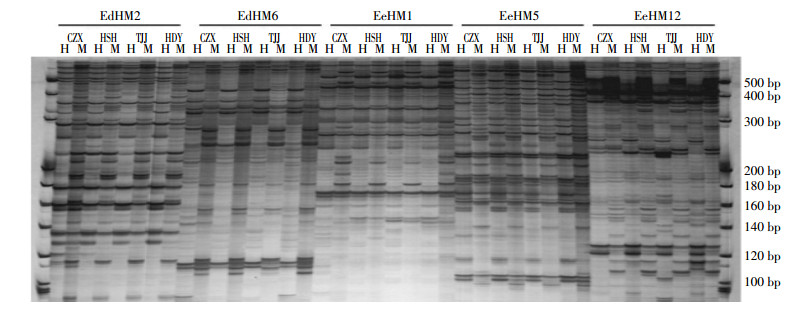
|
| 图 2 40个黄顶菊单株MSAP检测结果 Figure 2 MSAP profile of 40 Flaveria bidentis plants |
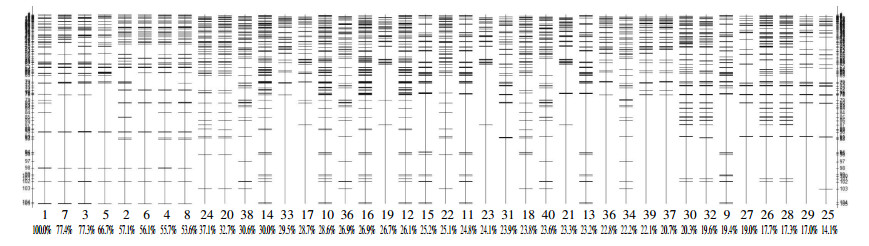
|
| 图 3 部分泳道量化分析图 Figure 3 Quantitative analysis diagram of part lane |
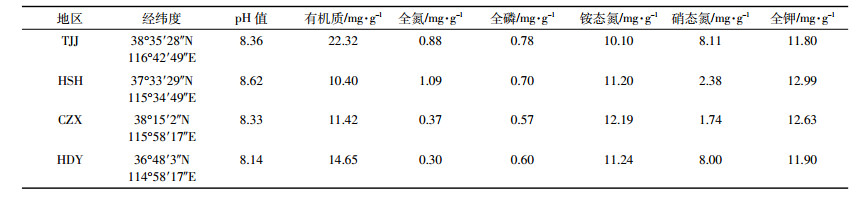
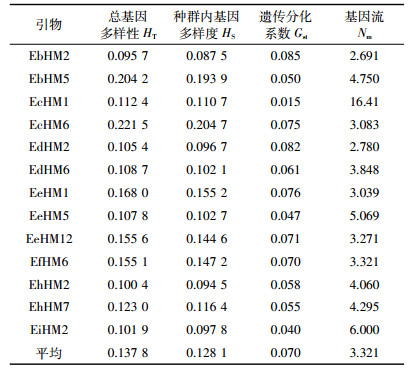
统计不同引物组合扩增的条带数, 结果如表 3所示, 13对引物共扩增出993条MSAP条带, 平均每对引物扩增76条。利用POPGene软件对样本遗传多样性的各项指标进行分析 (表 4), 13对引物观察等位基因数na平均值为1.885 2, 有效等位基因数ne平均值为1.4799, 杂合度h平均值为0.2910, 香农多态性指数I平均值为0.4425。多样性指数越高则多态性位点百分比也越高, 13对引物获得的993个位点中, 多态性位点为879个, 多态性百分比达到88.52%, 表明本研究筛选获得的13对引物组合非常适用于黄顶菊表观遗传多样性的研究。引物组合EdHM6的观察等位基因数、有效等位基因数、杂合度、香农多态性指数均高于平均值, 表明该引物对表观遗传多样性贡献率最大。EbHM5多态百分比最低 (仅72.73%), 其多样性指数偏低, 则该引物对表观遗传多样性贡献率最小。由表 5可知, 四个入侵地区黄顶菊种群多态性位点百分比在84.03%~92.31%范围内。TJJ黄顶菊种群多态性位点百分比最高, 为92.31%。香农多态性指数I在0.3981~0.4921范围内, 其中CZX黄顶菊种群香农多样性指数最高。Nei基因多样度H在0.2264~0.2645范围内, 其中CZX黄顶菊种群Nei基因多样度最高。根据Nei指数计算获得不同入侵地黄顶菊种群总基因多样度HT均值为0.1378, 种群内基因多样度HS均值为0.1281, 遗传分化系数Gst为0.07 (表 6)。四个不同入侵地黄顶菊种群间的遗传变异占种群总遗传变异的7%, 表明遗传变异主要是以相同入侵地区黄顶菊种群内变异为主。基因流Nm均值为3.321 > 1, 说明四个入侵地的黄顶菊种群间存在广泛的基因交流, 侧面印证不同入侵地区黄顶菊种群间遗传变异不显著。在种群遗传变异不显著情况下, 猜测入侵环境变化诱导黄顶菊表观遗传发生变化, 从而使得不同入侵地黄顶菊种群间甲基化模式改变。

|

|
|
HpaⅡ和MSPⅠ能识别相同的酶切位点5'-CCGG-3', 这是真核生物中常见的甲基化位点。但两者对5'-CCGG-3'位点内外侧胞嘧啶的敏感程度不同。因此HpaⅡ和MSPⅠ两种酶组合切割同一份DNA能产生不同的甲基化模式。MSAP甲基化模式共有4种类型。类型Ⅰ: HpaⅡ和MSPⅠ都能切开, 此类型DNA条带未甲基化; 类型Ⅱ: HpaⅡ能切开, MSPⅠ不能切开, 此类型DNA条带半甲基化; 类型Ⅲ: HpaⅡ不能切开, MSPⅠ能切开, 此类型DNA条带全甲基化; 类型Ⅳ: HpaⅡ和MSPⅠ都不能切开。
由表 7可知:四个入侵地区中, HDY获得的类型Ⅰ条带数最多, 平均785.3条; HSH获得的类型Ⅱ条带数最多, 平均103.2条; CZX获得的类型Ⅳ条带数最多, 平均111条; HSH和HDY获得的类型Ⅲ条带数最多, 平均66.2条。由图 4可知HSH与TJJ甲基化状态变化趋势相近, CZX与HDY甲基化状态变化趋势相近, HSH、TJJ、CZX、HDY半甲基化和全甲基化状态变化趋势有显著差异。

|
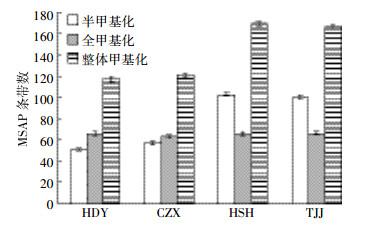
|
| 图 4 四个不同入侵地黄顶菊甲基化状态比较 Figure 4 Comparation of Flaveria bidentis methylation status of four different invasive areas |
利用Quantity One软件获得MSP (甲基化敏感位点) 和MISP (甲基化不敏感位点) 两类表观遗传数据, 进一步通过NTSYS软件绘制MSP和MISP各自的聚类图谱 (图 5和图 6)。聚类分析结果表明:四个不同入侵地区黄顶菊按照各自地理位置聚类在一起。黄顶菊种群个体间遗传距离越小, 彼此亲缘关系越近。其中TJJ黄顶菊与HDY黄顶菊最先聚类在一起, 说明TJJ与HDY这两个入侵地区黄顶菊种群间亲缘关系最为相近。TJJ与HDY黄顶菊种群作同一分支随后与HSH黄顶菊聚类在一起。TJJ、HDY、HSH作为一大分支最后与CZX黄顶菊聚类在一起。说明CZX黄顶菊种群与其他入侵地黄顶菊种群在亲缘关系上存在一定距离。四个不同入侵地黄顶菊MSP与MISP的聚类先后顺序大致相同, 说明在黄顶菊整个的种群遗传过程中, 四个不同入侵地区黄顶菊种群间没有较大的遗传分化。但不同入侵地黄顶菊种群间甲基化模式存在显著差异, 推测黄顶菊通过自身甲基化修饰来改变自身甲基化模式, 以更好地适应不同环境。
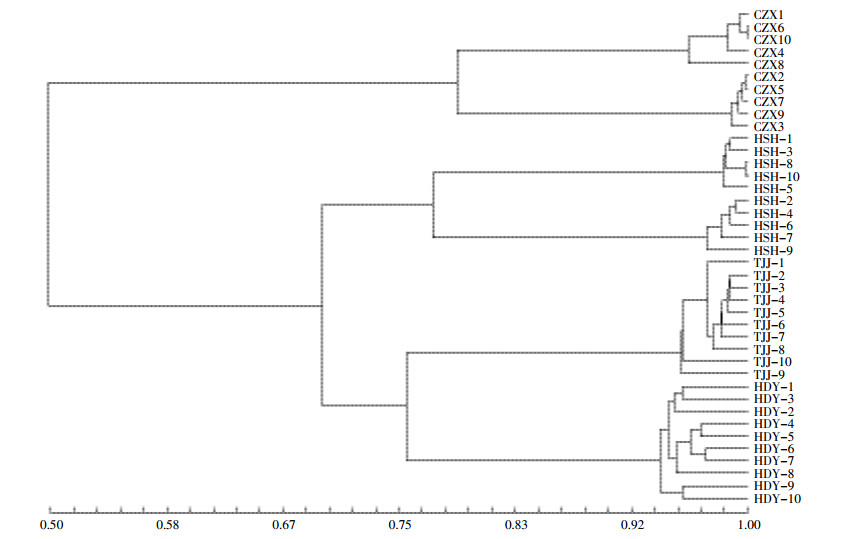
|
| 图 5 四个不同入侵地40个黄顶菊个体甲基化敏感(MSP)位点的聚类分析 Figure 5 Clustering analysis of 40 Flaveria bidentis methylation sensitive (MSP) loci in four different invasive areas |

|
| 图 6 四个不同入侵地区40个黄顶菊个体甲基化不敏感(MISP)位点的聚类分析 Figure 6 Clustering analysis of 40 Flaveria bidentis methylation non-sensitive (MISP) loci in four different invasive areas |
相关性分析是考察两个变量之间线性关系的一种统计分析方法, 可精确反应当一个变量发生变化时另一个变量的变化情况。利用SPSS软件分析不同入侵地区土壤环境因子与黄顶菊甲基化水平的相关性 (表 8)。土壤理化因子与黄顶菊整体甲基化 (或半甲基化) 相关系数从大到小排序:全氮 > 全磷 > pH > 铵态氮 > 有机质 > 全钾 > 硝态氮。土壤理化因子与黄顶菊全甲基化相关系数从大到小为:铵态氮 > 全磷 > 硝态氮 > 全氮 > 有机质 > 全钾 > pH。一般来说, 相关系数取绝对值后, 0~0.09视为没有相关性, 0.1~0.3为弱相关, 0.3~0.5为中等相关, 0.5~1.0为强相关。从表 8可以看到黄顶菊甲基化状态与土壤因子相关性强弱关系:黄顶菊整体甲基化 (或半甲基化) 与土壤全氮浓度、土壤全磷浓度、土壤pH最为相关; 黄顶菊全甲基化则与土壤铵态氮浓度、硝态氮浓度、全磷浓度最为相关, 其中与土壤铵态氮、硝态氮浓度为负相关关系。相关性仅仅看系数的大小是不够的, 还需要做显著性差异检验, 经t检验发现黄顶菊甲基化状态与土壤主要理化因子不存在显著相关性 (P > 0.01)。推测地区环境对黄顶菊表观遗传多样性的影响是受多重因素共同作用的结果, 而非某个单一因子的作用。
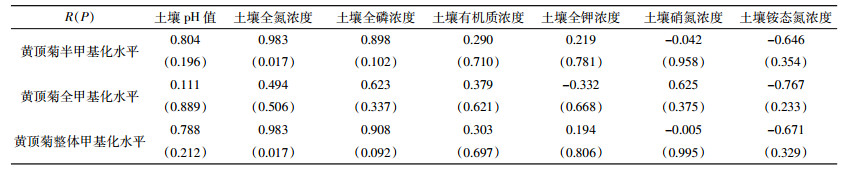
|
以往研究入侵植物与环境的相互作用以及物种的适应性进化都特别强调遗传变异的作用, 但事实上, 植物面对环境条件的变化也可以通过表型可塑性方式做出相应的变化来维持其适合度, 使其更好地适应环境。基因组表观遗传变异是环境适应性和表型可塑性发生的重要基础。Grativol等[17]发现, 遗传背景差异小的情况下, 环境变化能够引起植物有机体DNA甲基化的变异, 且这些DNA甲基化变异影响表型形成过程。基因组DNA甲基化的改变很有可能是调控入侵植物生境适应能力的重要机制之一。近年来, 黄顶菊入侵的生态学机制已经引起人们的重视, 但已有研究多集中在不同环境条件下黄顶菊的种子萌发特性[18-19]、入侵途径、传播方式、生物学特性[20]及生理生化指标等变化规律, 对黄顶菊基因组DNA甲基化的分布模式、变异情况等还缺乏全面的认识。因此, 本文通过研究四个典型入侵地生长的黄顶菊的甲基化水平及甲基化水平与环境理化因子相关性, 探究黄顶菊DNA表观遗传多样性是否受地理分布区域及入侵生境条件的影响。
从四个不同入侵地黄顶菊Nei氏遗传多样性指数H来看, TJJ、HSH、HDY、CZX之间数值相近, 没有明显的种群间遗传分化。但四个不同入侵地区黄顶菊种群间甲基化模式差异较大, 其中CZX与HDY两地植株的各自半甲基化位点和整体甲基化位点相近, 且两者均表现为半甲基化位点高于各自全甲基化位点。TJJ与CZX、HSH与CZX、HSH与HDY、TJJ与HDY两两相互比较发现:前者与后者半甲基化的比值略大于2, 整体甲基化的比值接近2。四个不同入侵地区黄顶菊种群间无明显的遗传变异情况下, 不同地区黄顶菊甲基化模式却存在显著差异, 推测是由于采样地生境不同所致, TJJ与HSH为水生境, 而CZX、HDY为陆生境。陈冬青等[21]对于不同环境下黄顶菊浸提液对多年生黑麦草萌发与生长影响的研究发现, 黄顶菊自身化感潜力是水边生境大于陆边生境。采样地生境的不同可能是导致黄顶菊自身甲基化模式差异的原因。
李红岩[22]对河北省黄顶菊遗传多样性和遗传分化的研究中, 采用AFLP来分析河北省7个种群、4个生境下的黄顶菊遗传多样性, 也证实了黄顶菊物种水平具有丰富遗传多样性, 对于黄顶菊入侵初期, 即使有很高的基因交流仍不能阻止黄顶菊种群遗传分化。在遗传漂流、地理隔离、选择压力下黄顶菊种群分化, 种群水平遗传多样性差异显著。在非适宜的环境, 如光照不足、重金属胁迫下, 遗传多样性会持续在一个低水平; 在良好的生长环境条件下, 则维持在高水平。对本文采样的四个典型黄顶菊入侵地进行对比发现, 采样地在地理位置上临近, 气候环境偏差不显著, 但存在一定生境差异, 且土壤理化因子也不同, 故推测入侵环境土壤因子理化差异也可能对黄顶菊甲基化模式产生一定的影响。四个入侵地土壤pH、铵态氮浓度、全钾浓度数值相近, 而土壤有机质、全氮浓度、硝态氮浓度数值却有显著差异, 四个地区土壤全磷浓度差异不明显。
通过分析黄顶菊甲基化模式与土壤因子相关性发现, 整体甲基化、半甲基化一致, 均与土壤全氮浓度、土壤全磷浓度、土壤pH最为相关, 说明全氮、全磷、土壤pH对黄顶菊整体甲基化和半甲基化模式影响大。全甲基化则与土壤铵态氮浓度、硝态氮浓度、全磷浓度最为相关, 其中与铵态氮、硝态氮浓度为负相关关系。铵态氮、硝态氮、全磷对全甲基化模式影响最大。通过t检验发现这种相关性不显著 (P > 0.01), 推测地区环境对黄顶菊表观遗传多样性的影响是多重因素共同作用的结果, 而非某个单一因子的作用。在种群间遗传分化不显著情况下, 土壤因子差异对黄顶菊甲基化模式有一定诱导作用, 使得黄顶菊对于不同入侵环境会通过甲基化修饰来改变自身甲基化模式, 用于适应新环境。
4 结论四个入侵地区的黄顶菊种群间遗传变异并不显著, 但表观遗传多样性丰富且其甲基化水平差异显著。不同环境中土壤理化因子对黄顶菊甲基化水平具有一定的影响, 尽管这种影响并不显著。推测黄顶菊通过甲基化修饰方式改变自身甲基化水平, 使得黄顶菊表观遗传发生了变化, 从而更好地适应新环境。对不同入侵地区黄顶菊表观遗传变化特征的初步探究有别于以往在遗传、生理、生化水平上的研究。这不仅从新的切入方向, 即表观遗传水平上对黄顶菊进行研究, 也为相同或相似种属的菊科植物表观遗传研究提供了参考。
| [1] | 张天瑞, 皇甫超河, 杨殿林, 等. 外来植物黄顶菊的入侵机制及生态调控技术研究进展[J]. 草业学报, 2011, 20(3): 268–278. ZHANG Tian-rui, HUANGFU Chao-he, YANG Dian-lin, et al. Invasion mechanism and ecological management of exotic plant Flaveria bidentis[J]. Chinese Journal of Aeronautics, 2011, 20(3): 268–278. DOI:10.11686/cyxb20110333 |
| [2] | 郭成亮, 胡文多, 朱敏峰, 等. 有害杂草黄顶菊在河北衡水的入侵途径调查[J]. 植物检疫, 2007, 2(3): 187–188. GUO Cheng-liang, HU Wen-duo, ZHU Min-feng, et al. Harmful weeds Flaveriabidentis in Hebei Hengshui invading route survey[J]. Plant Quarantine, 2007, 2(3): 187–188. |
| [3] | 芦站根, 崔兴国, 蒋文静. 衡水湖黄顶菊的入侵情况的初步调查研究[J]. 衡水学院学报, 2006, 8(1): 69–71. LU Zhan-gen, CUI Xing-guo, JIANG Wen-jing. The primary investigation and studies on the alien invasion of Flaveria bidentis Kuntze inHengshui Lake[J]. Hengshui College Journal, 2006, 8(1): 69–71. |
| [4] | 李香菊, 王贵启, 张朝贤, 等. 外来植物黄顶菊的分布、特征特性及化学防除[J]. 杂草科学, 2006(4): 58–61. LI Xiang-ju, WANG Gui-qi, ZHANG Chao-xian, et al. The distribution of alien plants Flaveria bidentis characteristics and chemical control[J]. Weed Science, 2006(4): 58–61. |
| [5] | Bossdorf O, Auge H, Lafuma L, et al. Phenotypic and genetic differentiation between native and introduced plant populations[J]. Oecologia, 2005, 14(3): 1–11. |
| [6] | Richards C L, Pigliucci M, et al. Invasion of diverse habitats by few Japanese knotweed genotypes is correlated with epigenetic differentiation[J]. Ecol Lett, 2012, 15(4): 1016–1025. |
| [7] | Richardson D M, Pysek P. Plant invasions:Merging the concepts of species invasiveness and community invasibility[J]. Prog Phys Geog, 2006, 30(2): 409–431. |
| [8] | Schmitz R J, Schultz M D, et al. Patterns of population epigenomic diversity[J]. Nature, 2013, 495(7440): 193–198. DOI:10.1038/nature11968 |
| [9] | Ruizgarcia L, Cervera M T, Martinezzapater J M, et al. DNA methylation increases throughout Arabidopsis development[J]. Planta, 2005, 222(2): 301–306. DOI:10.1007/s00425-005-1524-6 |
| [10] | Angers B, Castonguay E, Massicotte R. Environmentally induced phenotypes and DNA methylation:How to deal with unpredictable conditions until the next generation and after[J]. Mol Ecol, 2010, 19(7): 1283–1295. DOI:10.1111/mec.2010.19.issue-7 |
| [11] | 潘丽娜. 表观遗传修饰调控非生物胁迫应答提高植物抗逆性[J]. 遗传, 2013, 35(6): 745–751. PAN Li-na. Epigenetic regulation of abiotic stress response in plants to improve the stress tolerance[J]. Genetic, 2013, 35(6): 745–751. |
| [12] | 韩雅楠, 赵秀娟, 蔡禄. MSAP技术在植物抗逆性方面的应用[J]. 生物技术通报, 2010, 11(6): 71–79. HAN Ya-nan, ZHAO Xiu-juan, CAI Lu. Application of methylation-sensitive amplified polymorphism technology in plant stress resistance[J]. Biological Technical Bulletin, 2010, 11(6): 71–79. |
| [13] | Reyna-Lopez G E, Simpson J, Ruiz-Herrera J. Differences in DNA methylation pattern are detectable during the dimorphic transition of fungi by amplification of restriction polymorphism[J]. Molecular Genetics and Genomics, 1997, 253(6): 703–710. DOI:10.1007/s004380050374 |
| [14] | Wang B R, Li W G, Wang J B. Genetic diversity of Alternanthera philoxeroides in China[J]. Aquat Bot, 2005, 81(3): 277–283. DOI:10.1016/j.aquabot.2005.01.004 |
| [15] | 梁宏伟, 王长忠, 李忠, 等. 聚丙烯酰胺凝胶快速、高效银染方法的建立[J]. 遗传, 2008, 30(10): 1379–1382. LIANG Hong-wei, WANG Chang-zhong, LI Zhong, et al. Improvement of the silver-stained technique of polyacrylamide gel electrophoresis[J]. Hereditas, 2008, 30(10): 1379–1382. DOI:10.3321/j.issn:0253-9772.2008.10.021 |
| [16] | 陈瑞娟, 何蕾, 孙梨宗, 等. 铜胁迫对拟南芥幼苗生长和基因组DNA甲基化的影响[J]. 生态学杂志, 2014, 19(4): 2650–2657. CHEN Rui-juan, HE Lei, SUN Li-zong, et al. Effects of copper stress on the growth and genomic DNA methylation of Arabidopsis thaliana seedlings[J]. Chinese Journal of Ecology, 2014, 19(4): 2650–2657. |
| [17] | Grativol C, Hemerly A S, Ferreira P C G. Genetic and epigenetic regulation of stress responses in natural plant populations[J]. Biochim Biophys Acta, 2012, 1819(2): 176–185. DOI:10.1016/j.bbagrm.2011.08.010 |
| [18] | 苏立军, 王建平. 黄顶菊种子萌发特性研究[J]. 河北农业科学, 2008, 12(4): 39–40. SU Li-jun, WANG Jian-ping. Study of germination characteristics of Flaveria bidentis(L.) Kuntze[J]. Hebei Agricultural Science, 2008, 12(4): 39–40. |
| [19] | 徐兴友, 郭艾英, 胡京蕊, 等. 环境因子对黄顶菊种子萌发的影响[J]. 生态学报, 2009, 12(4): 1947–1953. XU Xing-you, GUO Ai-ying, HU Jing-rui, et al. Influence of environmental factors on seed germination of Flavera bidentis(L.) Kuntza[J]. Acta Ecologica Sinica, 2009, 12(4): 1947–1953. |
| [20] | 彭军, 马艳, 李香菊, 等. 黄顶菊化感作用研究进展[J]. 杂草科学, 2011, 31(1): 17–22. PENG Jun, MA Yan, LI Xiang-ju, et al. Flaveria bidentis allelopathy research progress[J]. Weed Science, 2011, 31(1): 17–22. |
| [21] | 陈冬青, 皇甫超河, 王楠楠, 等. 不同生长环境下黄顶菊浸提液对多年生黑麦草萌发与生长的影响[J]. 中国生态农业学报, 2012, 20(5): 586–591. CHEN Dong-qing, HUANGFU Chao-he, WANG Nan-nan, et al. Effect of extracts of Flaveria bidentis in different growth habitats on Loliumperenne germination and seedling growth[J]. Journal of Chinese Ecological Agriculture, 2012, 20(5): 586–591. |
| [22] | 李红岩. 河北省黄顶菊种群遗传多样性和遗传分化[J]. 中国农学通报, 2009, 25(10): 29–35. LI Hong-yan. Genetic diversity and differentiation among population of Flaveria bidentis in Hebei Province[J]. Chinese Agricultural Science Bulletin, 2009, 25(10): 29–35. |
 2017, Vol. 36
2017, Vol. 36