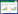文章信息
- 宋婕, 王鹤潼, 崔伟娜, 孙梨宗, 曹霞, 何蕾, 姜丽思, 成智博, 惠秀娟, 台培东, 杨悦锁, 刘宛
- SONG Jie, WANG He-tong, CUI Wei-na, SUN Li-zong, CAO Xia, HE Lei, JIANG Li-si, CHENG Zhi-bo, HUI Xiu-juan, TAI Pei-dong, YANG Yue-suo, LIU Wan
- Cd胁迫诱导拟南芥幼苗DNA损伤分析
- Analysis of Cd-induced DNA damage in Arabidopsis seedlings
- 农业环境科学学报, 2017, 36(4): 635-642
- Journal of Agro-Environment Science, 2017, 36(4): 635-642
- http://dx.doi.org/10.11654/jaes.2016-1467
文章历史
- 收稿日期: 2016-11-21
2. 中国科学院沈阳应用生态研究所污染生态与环境工程重点实验室, 沈阳 110016;
3. 辽宁何氏医学院, 沈阳 110163;
4. 上海应用技术学院, 上海 201418;
5. 沈阳农业大学, 沈阳 110866;
6. 通辽市农业科学研究院蔬菜所, 内蒙古 通辽 028000;
7. 沈阳大学区域污染环境生态修复教育部重点实验室, 沈阳 110044
2. Key Laboratory of Pollution Ecology and Environmental Engineering, Institute of Applied Ecology, Chinese Academy of Sciences, Shenyang 110016, China;
3. Liaoning He University, Shenyang 110163, China;
4. Shanghai Institute of Technology, Shanghai 201418, China;
5. Shenyang Agricultural University, Shenyang 110866, China;
6. Institute of Vegetable Science, Tongliao Academy of Agricultural Sciences, Tongliao 028000, China;
7. Laboratory of Ecological Restoration of Polluted Environment and Resource Technology, Shenyang University, Shenyang 110044, China
2014年, 环境保护部和国土资源部联合发布《全国土壤污染状况调查公报》显示, 全国受重金属污染的耕地面积超过2000万hm2, 其中我国耕地土壤镉 (Cd) 污染点位超标率达7%[1]。Cd具有极强的毒性及蓄积性, 土壤中的Cd会在农作物中富集, 通过生物放大和积累作用, 对动物和人的肾、肝、肺、骨、生殖和免疫系统产生一系列损伤[2]。近年来, Cd对于生物的毒性, 特别是遗传毒性[3-5]已在全球范围内引起广泛关注。
目前, 关于Cd的生态遗传毒理研究已有很多报道, 马引力等[6]和张旭红等[7]报道了Cd胁迫可对小麦和蚕豆造成不同类型的DNA损伤; Pierron等[8]发现, 长期Cd胁迫会造成欧洲鳝鱼的基因组甲基化水平升高, 引起表观遗传损伤; 本实验室近年来研究发现, Cd胁迫会导致拟南芥幼苗基因组甲基化图谱改变, 引起表观遗传损伤[9-11]。虽有证据表明Cd胁迫会造成动、植物遗传损伤, 然而对Cd胁迫诱导植物DNA损伤修复机制的研究比较少。
研究表明, Cd对基因组稳定性的损伤多为非直接的, 而是通过引起氧化损伤以及对DNA修复系统酶系的抑制[12-13], 使大量未修复的DNA在细胞中积累并随细胞周期大量复制, 引起基因突变率和基因组不稳定性升高[14], 最终导致细胞凋亡。植物针对不同类型的DNA损伤有多种修复途径, 如修复单核苷酸损伤的碱基切除修复和核苷酸切除修复途径、错配修复以及对双链断裂进行修复的非同源末端连接 (Non-homologous end joining, NHEJ) 和同源重组 (Homologous recombination, HR)[15-16]。Jia等[17]报道了乙基甲磺酸诱导拟南芥jhs1幼苗中DNA损伤明显, DNA修复相关的BRCA1、RAD51、GR1、KU70、MRE11基因表达明显上调。但是, 目前关于Cd胁迫对拟南芥HR和NHEJ系统基因表达的影响, 国内外尚未见报道。
本文使用RAPD方法对Cd胁迫5 d的拟南芥幼苗DNA损伤情况进行了检测。同时采用ELISA方法, 探究了不同浓度Cd处理对拟南芥幼苗全基因组DNA甲基化的影响, 并分析了多个DNA损伤修复以及细胞增殖相关基因的表达, 首次在胁迫损伤、损伤后修复及细胞周期响应三个层面研究了Cd胁迫造成的拟南芥幼苗DNA损伤及其修复机制, 并寻找对Cd胁迫敏感的生物标记物。
1 材料方法 1.1 供试材料的培养与处理实验选用拟南芥 (Arabidopsis thaliana, 哥伦比亚生态型) 为材料。种子经10%次氯酸钠及70%乙醇溶液消毒、灭菌, 水中4 ℃春化24~48 h后, 用含CdCl2 (以Cd2+计) 0、0.125、0.25、1.0、2.5 mg·L-1的0.5×M&S培养基 (Caisson, 美国, 0.5%蔗糖) 于21 ℃培养5 d, 光暗周期为12 h/12 h, 光强为3000 lx。每个处理重复3次。
1.2 核酸的提取将相同处理长势良好的整株幼苗进行混合后提取核酸[18-19], 使用北京康维世纪生物科技有限公司的新型植物基因组DNA提取试剂盒 (CW0531) 提取幼苗全基因组DNA; 使用EZ-10 DNAaway RNA Mini-prep Kit (生工, 上海) 提取全基因组RNA。所得DNA及RNA用Eppendorf BioPhotometer Plus核酸蛋白测定仪对其纯度及含量进行检测, 用1%琼脂糖凝胶电泳及Takara DL2000 DNA Marker进行含量校准。
1.3 RAPD分析RAPD分析参照Liu等[9,20]的方法进行, 对PCR条件略加优化, 引物选用Primer 3: 5'-CTGCGCTGGA-3'; Primer 5: 5'-CTGGGGCTGA-3';Primer 9: 5'-AAAGTGC GGC-3'; Primer 11: 5'-AGACCCAGAG-3'。
随机引物扩增使用的PCR体系如下: 80 ng DNA模板, 0.5 μmol·L-1引物, 0.2 mmol·L-1 dNTP Mixture, 1×PCR Buffer以及1 U LA Taq DNA聚合酶 (宝生物, 大连), 并用ddH2O补足体系体积至25 μL。
PCR反应程序为: 94 ℃预变性5 min, 94 ℃ 60 s, 38 ℃ 60 s, 72 ℃ 90 s, 循环35次, 72 ℃延伸10 min。PCR产物用5%聚丙烯酰氨凝胶电泳 (50%尿素) 进行分离, 经银染后用Bio-Rad凝胶成像系统成像, 并使用Image Lab (Bio-Rad) 软件对条带进行分析。处理组与对照组比较, 增加或缺失条带记为1个多态性条带, 条带亮度增加或减少一半以上, 记为0.5个多态性条带。同一个多态性条带在3次重复中出现2次及以上记为有效多态性条带。
1.4 全基因组甲基化率的测定参照Keller等[21]的方法, 使用5-mC DNA ELISA Kit (ZYMO, 美国) 建立标准曲线, 并对各组幼苗全基因组DNA进行全基因组甲基化率分析。
1.5 Real-time PCR分析使用PrimeScript ® 1st Strand cDNA Synthesis Kit (宝生物, 大连) 对所提取的RNA进行反转录, 每个处理组取3 μg RNA合成相应的cDNA, 用于后续的Real-time PCR反应, 合成的cDNA于-20 ℃保存。
Real-time PCR反应使用SYBR ® Premix Ex TaqTM Ⅱ (Tli RNaseH Plus) (宝生物, 大连), 在Bio-rad iQTM5多重实时荧光定量PCR仪上进行, 扩增所使用的引物如表 1所示。PCR扩增条件为:95 ℃预变性30 s, 95 ℃变性5 s, 60 ℃退火1 min, 74 ℃延伸10 s, 反应进行40个循环, 每个反应结束后采集荧光。所有样品及空白对照均做3个重复, 并建立实时扩增曲线及溶解曲线。
数据结果采用SPSS 19.0软件和Microsoft Excel 2013进行统计分析, 组间比较采用单因素方差分析, 结果表示为平均值±标准差。
2 结果与分析 2.1 Cd胁迫对拟南芥幼苗根系生长的影响如表 2所示, 对照组与处理组幼苗叶片数均为2。0.125 mg·L-1 Cd对拟南芥幼苗根系生长有显著的促进作用, 根长诱导率为16.85% (P < 0.05)。0.25 mg·L-1 Cd胁迫对幼苗根系的影响表现为促进生长, 但与对照组并没有显著差异。高浓度处理组 (1.0、2.5 mg·L-1 Cd) 对根系的生长表现出显著的抑制效应, 根系抑制率分别为52.56% (P < 0.05) 和65.17% (P < 0.01)。
|
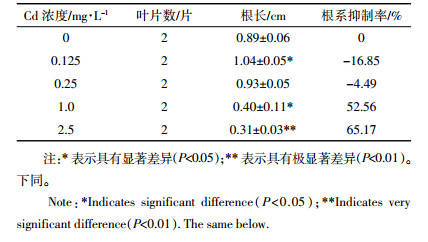
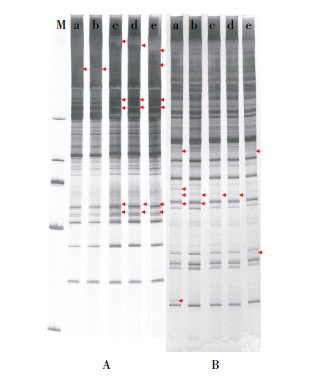
图 1为Cd胁迫5 d的拟南芥幼苗基因组DNA使用随机引物Primer 3、11扩增后的RAPD图谱, 在200~2000 bp范围内, 每条泳道均有10条以上条带, 适于进行RAPD分析。对4条随机引物扩增图谱中多态性条带数量进行分析, 结果如图 2所示。Cd胁迫处理5 d后, 与对照组相比, 0.125 mg·L-1 Cd胁迫幼苗中可检测出3条多态性条带。随着Cd胁迫浓度的增加, 处理组中检测到的多态性条带数量有所增加, 0.25、1.0、2.5 mg·L-1 Cd处理组多态性条带数量分别为10、12、17。
|
| 图 1 Cd胁迫5 d后拟南芥幼苗RAPD图谱 Figure 1 RAPD fingerprints of Arabidopsis seedlings exposed to 0~2.5 mg·L-1 Cd for 5 d M为DNA marker(Takara DL2000),片段从大到小分别为2 kb、1 kb、750 bp、500 bp、250 bp;A为Primer 3,B为Primer 11的RAPD图谱;a~e分别表示Cd胁迫浓度为0、0.125、0.25、1.0、2.5 mg·L-1,下同 M stands for DNA marker (Takara DL2000); a~e for RAPD fingerprints using Primer 3 (A) and Primer 11 (B) of Arabidopsis seedlings exposed to 0~2.5 mg· L-1 Cd for 5 d, The same below |
|
| 图 2 Cd胁迫5 d后拟南芥幼苗DNA随机引物扩增多态性 Figure 2 Polymorphism variations detected by RAPD from Arabidopsis seedlings exposed to 0~2.5 mg·L-1 Cd for 5 d |
如图 3所示, 随着Cd处理浓度的增加, 拟南芥幼苗全基因组甲基化率有所增加, 两者呈现出良好的剂量效应关系。Cd胁迫5 d后, 对照组全基因组甲基化率为20.19%;0.125 mg·L-1 Cd处理组的全基因组甲基化率略高于对照组, 但并无显著差异; 0.25 mg·L-1处理组全基因组甲基化率为25.94%, 与对照组有显著差异 (P < 0.05); 1.0、2.5 mg·L-1 Cd处理后拟南芥幼苗全基因组甲基化率分别为30.25%、32.30%, 与对照组有极显著差异 (P < 0.01)。
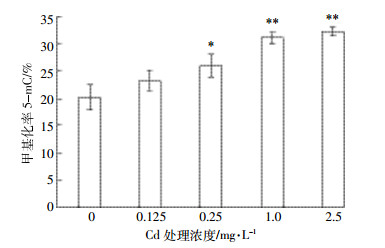
|
| 图 3 Cd胁迫5 d后拟南芥幼苗全基因组甲基化率 Figure 3 Global methylation levels of Cd-induced Arabidopsis seedlings for 5 d |
本实验采用实时荧光定量PCR方法, 以UBQ10为看家基因, 检测拟南芥幼苗基因在不同浓度Cd胁迫5 d后的相对表达情况 (以对照组各基因的表达量为100%)。如图 4A所示, 在0.125 mg·L-1 Cd胁迫下, PCNA1、PCNA2基因的表达量均显著增加1.5倍左右; 在Cd胁迫浓度为0.25 mg·L-1时, PCNA1基因表达量仍然显著增加, 而PCNA2基因与对照组相比并无显著差异。1.0 mg·L-1 Cd胁迫下, PCNA1、PCNA2基因的表达与对照组相比均无显著性差异。Cd胁迫浓度增至2.5 mg·L-1时, 两个细胞周期相关基因的表达均受到明显抑制, 表达量仅为对照组的52.7%和66.6%。
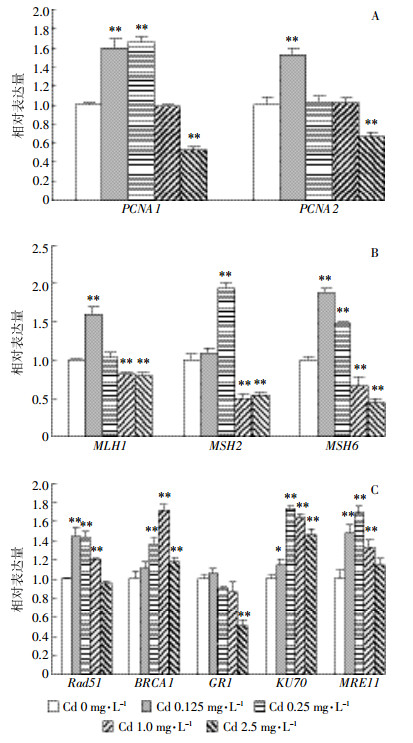
|
| 图 4 Cd胁迫5 d后拟南芥幼苗细胞周期和DNA损伤修复标志基因的表达情况 Figure 4 Expression levels of marker genes from Cd-induced Arabidopsis seedlings for 5 d |
拟南芥幼苗错配修复基因MLH1、MSH2、MSH6在Cd胁迫5 d后表达情况如图 4B所示。Cd胁迫浓度为0.125 mg·L-1时, MLH1、MSH6的表达均增加至原来的1.5倍以上, MSH2的表达与对照组相比无显著差异; 0.25 mg·L-1 Cd胁迫下, MLH1的表达与对照组并无显著差异, MSH2、MSH6基因的表达与对照组相比仍然显著增加; 1.0、2.5 mg·L-1 Cd胁迫下, 3个错配修复基因的表达均受到明显抑制。总体上, 随Cd胁迫浓度的增加, 错配修复基因MLH1、MSH2、MSH6的表达均呈明显的倒U型。
Cd胁迫5 d对拟南芥幼苗DNA损伤修复标志基因表达的影响如图 4C所示。5个基因的表达随Cd胁迫浓度的增加均呈现倒U型剂量效应关系。RAD51、MRE11、KU70、BRCA1的表达最大值分别出现在0.125、0.25、0.25、1.0 mg·L-1, 最大表达量分别为对照组的1.44、1.69、1.72、1.71倍。Cd胁迫浓度增至2.5 mg·L-1时, 这4个基因仍然未表现出显著的表达抑制, 且BRCA1和KU70基因的表达量仍显著高于对照组。GR1虽也呈现出相似的变化趋势, 但在0.25、1.0 mg·L-1 Cd胁迫下表达量的变化与对照组相比并无显著差异, 在2.5 mg·L-1 Cd胁迫下表现出明显的表达抑制, 表达量降至对照组的51.4%。
3 讨论 3.1 Cd胁迫诱导拟南芥幼苗DNA损伤0.125~2.5 mg·L-1 Cd处理组均检测到多态性条带 (图 2), 且随着胁迫程度的增加, RAPD多态性条带明显增加, DNA损伤加重, 二者呈剂量效应关系。这与Wang等[10]的结果虽略有不同, 但趋势一致。Cd胁迫15 d时, Wang等在0.25 mg·L-1及以下浓度Cd处理组未检测到多态性, 其可能原因一是本研究选用了扩增能力极强的LA Taq DNA聚合酶, 使得RAPD图谱中, 同等处理条件下条带更多且长片段居多, 敏感性得以增强, 更易检测出DNA损伤。二是胁迫时间不同造成的差异:在低浓度Cd胁迫下, 胁迫5 d后的幼苗中DNA损伤可能由于时间较短不能及时修复, 而胁迫15 d后, 由于错配修复系统增强, 错配等轻损伤已经得到修复, 导致5 d Cd胁迫损伤大于15 d; 在高浓度Cd胁迫下, 损伤涉及染色体损伤与分裂异常重组, 较难恢复, 而且修复系统同样会受到抑制, 还可激活跨损伤DNA合成机制[28], 导致15 d Cd胁迫拟南芥幼苗的DNA损伤显著高于5 d Cd胁迫。
3.2 Cd胁迫诱导拟南芥幼苗全基因组超甲基化植物全基因组甲基化水平是反映植物胁迫响应、基因组不稳定性以及遗传损伤的重要指标[29-30]。近年来研究发现, 胁迫引起的全基因组甲基化改变既可以呈现超甲基化又可以表现为低甲基化[13]。在本研究中, 随着Cd胁迫浓度的增加, 拟南芥幼苗全基因组甲基化率增加 (超甲基化), 且两者呈明显剂量效应关系。目前研究认为, 全基因组超甲基化一般为低剂量毒害导致的生物胁迫响应。Pierron等[8]和Jiang等[31]报道了低剂量Cd能诱导欧洲鳝鱼、人胚胎肺纤维组织母细胞全基因组甲基化水平升高。而全基因组低甲基化一般是遗传严重损伤的表现, 难于恢复或逆转, 并可激活跨损伤复制[32], 将损伤传递, 最终导致细胞死亡或癌变。如Pogribny等[33]研究发现, 辐射诱导的小鼠基因组低甲基化与DNA损伤修复有关; Silva等[34]研究表明, 全基因组低甲基化与结肠直肠癌有关。然而, 无论基因组呈现超甲基化或是低甲基化, 都会增加基因组的不稳定性, 因为胁迫引起的改变本身比改变方向更为重要[35]。在本研究中, Cd胁迫幼苗虽都呈现超甲基化, 但其剂量效应变化趋势却与RAPD结果一致, 说明随Cd胁迫的增强, 遗传损伤与基因组不稳定性增加 (图 2、图 3)。类似的结果在Cd胁迫诱导的萝卜[36]中也有报道。
本实验选用简便、高效的ELISA试剂盒对全基因组甲基化率进行测定, 与传统的HPLC法、MSAP法以及本实验室之前发展的MSAP-PCR方法[37]相比, 更加快速、简单, 无需设计、筛选引物等。然而, 缺点在于无法检测出具体超甲基化及去甲基化的细节, 只可检测整体水平基因组甲基化情况, 比较适合甲基化的快速检测。
3.3 Cd胁迫诱导拟南芥幼苗DNA修复机制及细胞周期响应增殖细胞核抗原 (Proliferating cell nuclear antigen, PCNA), 是存在于所有真核生物细胞核中的一种蛋白复合物, 是诱导细胞周期由G1期向S期转变的标志基因之一[38], 在DNA合成与修复中也被作为常用的增殖标志物, 检测PCNA是研究细胞增殖活性与细胞周期的可靠方法[39]。在拟南芥中, PCNA的同源基因为PCNA1和PCNA2, 二者均参与DNA修复及细胞周期调控过程。本研究中, 在Cd胁迫浓度为0.125 mg·L-1时, 这2个基因的表达均明显增加 (图 4A), 随后开始下降, 到2.5 mg·L-1时表达受到明显抑制, 说明低浓度Cd胁迫促进拟南芥生长, 随着胁迫浓度增加开始出现生长抑制, 与幼苗根系的生长一致 (表 2)。类似的结果在Cd胁迫60 h[40]的拟南芥幼苗中也有报道。
本研究通过分析DNA损伤修复相关基因的表达情况, 从损伤后修复角度探索了Cd胁迫下拟南芥幼苗DNA损伤机制。本研究发现:(1) 低浓度Cd胁迫可以引起拟南芥幼苗的DNA错配损伤, 由于该损伤可被MMR系统修复, 因此MMR基因MLH1、MSH2、MSH6在0.125、0.25 mg·L-1 Cd胁迫下表达显著增加。虽然MMR系统在识别错配后会激活DNA损伤检验点阻滞细胞周期, 但由于胁迫浓度低、损伤小、易修复, 导致细胞周期阻滞程度较低, 而胁迫响应诱导了PCNA1、PCNA2基因的表达增加 (S期DNA合成增加), 反而代偿了错配损伤的细胞周期阻滞, 而且进一步导致了细胞增殖加快, 促进了拟南芥幼苗生长。(2) 随着Cd胁迫增加, 诱导拟南芥幼苗产生染色体损伤和有丝分裂异常重组, 表现在同源重组和非同源末端连接相关基因的表达增加; 另外, 因PCNA2基因表达降低, 细胞周期开始出现阻滞, 细胞分裂减弱, 生长受到抑制 (表 2)。(3) 当Cd胁迫浓度达到1.0 mg·L-1时, MMR基因表达受到明显抑制, 而BRCA1基因表达量达到最大值, 说明此时错配修复系统受到抑制, 参与DNA损伤修复的是HR与NHEJ途径。在最大浓度2.5 mg·L-1 Cd胁迫下, 所有修复与细胞周期相关基因表达均明显下降 (图 4), 说明此时DNA损伤严重, 可能修复基因本身或相关调控基因已经受到损伤, 所有修复途径均被抑制。这与本文中DNA损伤以及甲基化损伤在此浓度下明显增加的结果一致。
3.4 DNA损伤修复基因表达作为对Cd胁迫敏感的生物标记物生物标记物可准确高效地指示出生物体受到环境胁迫的情况, 寻找敏感的生物标记物已成为当今污染诊断和胁迫毒理研究中的热点之一[5,11]。本研究结果显示, 拟南芥幼苗在Cd胁迫5 d后, 随着胁迫浓度的增大, 各损伤修复基因表达变化量以及达到峰值的先后顺序为:MSH6、MLH1、RAD51、MLH2、MRE11、KU70、BRCA1、GR1。在对Cd胁迫敏感度上, DNA损伤修复系统总体表现为MMR > HR > NHEJ。其中, MSH6和MLH1基因在0.125 mg·L-1 Cd胁迫下表达差异最显著, 均在1.5倍以上。MSH2虽然有着最显著的表达差异, 但其峰值出现在0.25 mg·L-1, 在敏感性上弱于MSH6、MLH1基因。这可能是因为MSH2与MSH6共同形成MutLα复合体, 而MSH6基因有研究证明是Cd的胁迫靶位点[41], 从而导致MSH6的敏感性高于MSH2。综合本研究检测的8个DNA损伤修复基因在对Cd胁迫敏感性与响应显著性上的表现, MSH6、MLH1基因可作为拟南芥中对Cd胁迫敏感的生物标记物。
4 结论(1) Cd (0.125~2.5 mg·L-1) 胁迫5 d后, 胁迫浓度越大, 拟南芥幼苗受到的DNA损伤和表观遗传损伤越严重。
(2) 在对Cd胁迫敏感度上, 拟南芥DNA损伤修复系统总体表现为MMR > HR > NHEJ。轻度Cd胁迫主要引起DNA错配损伤, 同时促进生长。随着Cd胁迫浓度的增大, 会引起DNA断裂与染色体损伤, 从而造成细胞周期阻滞与生长抑制。
(3) 本文检测的细胞周期及DNA损伤修复基因的表达均与Cd胁迫浓度的增加呈明显的倒U型剂量效应关系。根据各损伤修复基因表达变化量以及达到峰值的先后顺序, 拟南芥幼苗MSH6、MLH1基因的表达对Cd胁迫最为敏感, 可作为检测Cd胁迫对植物遗传毒性效应的敏感生物标记物。
| [1] | 庞荣丽, 王瑞萍, 谢汉忠, 等. 农业土壤中镉污染现状及污染途径分析[J]. 天津农业科学, 2016, 22(12): 87–91. PANG Rong-li, WANG Rui-ping, XIE Han-zhong, et al. Analysis of cadmium pollution in agricultural soils and analysis of its way of pollution[J]. Tianjin Agricultural Sciences, 2016, 22(12): 87–91. DOI:10.3969/j.issn.1006-6500.2016.12.023 |
| [2] | 刁书永, 张立志, 袁慧. 镉中毒机理研究进展[J]. 动物医学进展, 2005, 26(5): 49–51. DIAO Shu-yong, ZHANG Li-zhi, YUAN Hui. Research Progress on the mechanism of cadmium poisoning[J]. Progress in Veterinary Medicine, 2005, 26(5): 49–51. |
| [3] | Adams S V, Barrick B, Christopher E P, et al. Genetic variation in metallothionein and metal-regulatory transcription factor 1 in relation to urinary cadmium, copper, and zinc[J]. Toxicol Appl Pharmacol, 2015, 289(3): 381–388. DOI:10.1016/j.taap.2015.10.024 |
| [4] | Virani S, Rentschler K M, Nishijo M, et al. DNA methylation is differentially associated with environmental cadmium exposure based on sex and smoking status[J]. Chemosphere, 2016, 145: 284–290. DOI:10.1016/j.chemosphere.2015.10.123 |
| [5] | 何蕾, 王鹤潼, 宋婕, 等. 拟南芥MLH1基因启动子甲基化作为镉胁迫生物标记物潜力评估[J]. 生态学杂志, 2016, 35(5): 1386–1393. HE Lei, WANG He-tong, SONG Jie, et al. Methylation of MLH1 promoter in Arabidopsis thaliana as a biomarker of cadmium stress[J]. Chinese Journal of Ecology, 2016, 35(5): 1386–1393. |
| [6] | 马引利, 佘小平. 铝、镉对小麦幼苗生长的影响及其DNA损伤效应研究[J]. 西北植物学报, 2006, 26(4): 729–735. MA Yin-li, SHE Xiao-ping. Effects and DNA damages of Cd2+ and Al3+ in wheat seedlings[J]. Botanica Boreali-Occidentalia Sinica, 2006, 26(4): 729–735. |
| [7] | 张旭红, 林爱军, 苏玉红, 等. 镉引起蚕豆 (Vicia faba) 叶片DNA损伤和细胞凋亡研究[J]. 环境科学, 2006, 27(4): 787–793. ZHANG Xu-hong, LIN Ai-jun, SU Yu-hong, et al. DNA damages and apoptosis induced by Cd in the leaves of horsebean Vicia faba[J]. Environmental Science, 2006, 27(4): 787–793. |
| [8] | Pierron F, Baillon L, Sow M, et al. Effect of low-dose cadmium exposure on DNA methylation in the endangered European eel[J]. Environmental Science Technology, 2014, 48(1): 797–803. DOI:10.1021/es4048347 |
| [9] | Liu W, Li P J, Qi X M, et al. DNA changes in barley (Hordeum vulgare) seedlings induced by cadmium pollution using RAPD analysis[J]. Chemosphere, 2005, 61(2): 158–167. DOI:10.1016/j.chemosphere.2005.02.078 |
| [10] | Wang H, He L, Song J, et al. Cadmium-induced genomic instability in arabidopsis:Molecular toxicological biomarkers for early diagnosis of cadmium stress[J]. Chemosphere, 2016, 150: 258–265. DOI:10.1016/j.chemosphere.2016.02.042 |
| [11] | Hsu T, Huang K M, Tsai H T, et al. Cadmium (Cd)-induced oxidative stress down-regulates the gene expression of DNA mismatch recognition proteins MutS homolog 2(MSH2) and MSH6 in zebrafish (Danio rerio) embryos[J]. Aquat Toxicol, 2013, 126: 9–16. DOI:10.1016/j.aquatox.2012.09.020 |
| [12] | 朱美霖, 魏富刚, 崔斌, 等. 土壤Cd胁迫对三七生长和根系DNA损伤及抗氧化酶活性的影响[J]. 植物资源与环境学报, 2014, 23(1): 58–64. ZHU Mei-lin, WEI Fu-gang, CUI Bin, et al. Effect of soil Cd stress on growth, root system DNA damage and antioxidant enzyme activity of Panax notoginseng[J]. Journal of Plant Resources and Environment, 2014, 23(1): 58–64. |
| [13] | Filipic M. Mechanisms of cadmium induced genomic instability[J]. Mutation Research, 2012, 733(1/2): 69–77. |
| [14] | 范文红, 詹启敏. BRCA1和基因组稳定性[J]. 癌症, 2003, 22(3): 331–335. FAN Wen-hong, ZHAN Qi-min. BRCA1 and genomic stability[J]. Chinese Journal of Cancer, 2003, 22(3): 331–335. |
| [15] | 严振鑫, 徐冬一. DNA双链断裂的非同源末端连接修复[J]. 生命科学, 2014, 26(11): 1157–1165. YAN Zhen-xin, XU Dong-yi. Role of nonhomologous end joining pathway in the repair of DNA double-strand breaks[J]. Chinese Bulletin of Life Sciences, 2014, 26(11): 1157–1165. |
| [16] | Roth N, Klimesch J, Dukowic-Schulze S, et al. The requirement for recombination factors differs considerably between different pathways of homologous double-strand break repair in somatic plant cells[J]. Plant Journal, 2012, 72(5): 781–790. DOI:10.1111/tpj.2012.72.issue-5 |
| [17] | Jia N, Liu X, Gao H. A DNA2 homolog is required for DNA damage repair, cell cycle regulation, and meristem maintenance in plants[J]. Plant Physiology, 2016, 171(1): 318–333. DOI:10.1104/pp.16.00312 |
| [18] | Yuan D, Lai J, Xu P, et al. Atmms21 regulates DNA damage response and homologous recombination repair in arabidopsis[J]. DNA Repair, 2014, 21: 140–147. DOI:10.1016/j.dnarep.2014.04.006 |
| [19] | Kandasamy M K, McKinney E C, Deal R B, et al. Arabidopsis actin-related protein ARP5 in multicellular development and DNA repair[J]. Developmental Biology, 2009, 335(1): 22–32. DOI:10.1016/j.ydbio.2009.08.006 |
| [20] | Liu W, Yang Y S, Li P J, et al. Risk assessment of cadmium-contaminated soil on plant DNA damage using RAPD and physiological indices[J]. Journal of Hazardous Materials, 2009, 161(2/3): 878–883. |
| [21] | Keller M, Kralisch S, Rohde K, et al. Global DNA methylation levels in human adipose tissue are related to fat distribution and glucose homeostasis[J]. Diabetologia, 2014, 57(11): 2374–2383. DOI:10.1007/s00125-014-3356-z |
| [22] | Liu W, Yang Y S, Francis D, et al. Cadmium stress alters gene expression of DNA mismatch repair related genes in Arabidopsis seedlings[J]. Chemosphere, 2008, 73(7): 1138–1144. DOI:10.1016/j.chemosphere.2008.07.020 |
| [23] | Wang J, Huang S, Xing L, et al. Role of hMLH1 in sterigmatocystin-induced G2 phase arrest in human esophageal epithelial Het-1A cells in vitro[J]. Toxicology Letter, 2013, 217(3): 226–234. DOI:10.1016/j.toxlet.2012.12.020 |
| [24] | Edelbrock M A, Kaliyaperumal S, Williams K J. Structural, molecular and cellular functions of MSH2 and MSH6 during DNA mismatch repair, damage signaling and other noncanonical activities[J]. Mutation Research, 2013, 743/744: 53–66. DOI:10.1016/j.mrfmmm.2012.12.008 |
| [25] | Xue C, Liang K, Liu Z, et al. Similarities and differences between arabidopsis PCNA1 and PCNA2 in complementing the yeast DNA damage tolerance defect[J]. DNA Repair, 2015, 28: 28–36. DOI:10.1016/j.dnarep.2015.02.003 |
| [26] | Lafarge S, Montane M H. Characterization of arabidopsis thaliana ortholog of the human breast cancer susceptibility gene 1:AtBRCA1, strongly induced by gamma rays[J]. Nucleic Acids Research, 2003, 31(4): 1148–1155. DOI:10.1093/nar/gkg202 |
| [27] | Rein K, Stracker T H. The MRE11 complex:An important source of stress relief[J]. Experimental Cell Research, 2014, 329(1): 162–169. DOI:10.1016/j.yexcr.2014.10.010 |
| [28] | 秦周帅, 张传林, 萧伟. 受PCNA翻译后修饰调控的DNA损伤耐受机制[J]. 生命科学, 2014, 26(11): 1143–1156. QIN Zhou-shuai, ZHANG Chuan-lin, XIAO Wei. DNA damage tolerance controlled by PCNA post-translational modifcations[J]. Chinese Bulletin of Life Sciences, 2014, 26(11): 1143–1156. |
| [29] | Ou X, Zhang Y, Xu C, et al. Transgenerational inheritance of modified DNA methylation patterns and enhanced tolerance induced by heavy metal stress in rice (Oryza sativa L.)[J]. PLoS One, 2012, 7(9): e41143. DOI:10.1371/journal.pone.0041143 |
| [30] | Li Z, Liu Z, Chen R, et al. DNA damage and genetic methylation changes caused by Cd in Arabidopsis thaliana seedlings[J]. Environ Toxicol Chem, 2015, 34(9): 2095–2103. DOI:10.1002/etc.3033 |
| [31] | Jiang G, Xu L, Song S, et al. Effects of long-term low-dose cadmium exposure on genomic DNA methylation in human embryo lung fibroblast cells[J]. Toxicology, 2008, 244(1): 49–55. DOI:10.1016/j.tox.2007.10.028 |
| [32] | Zang H, Goodenough A K, Choi J Y, et al. DNA adduct bypass polymerization by sulfolobus solfataricus DNA polymerase Dpo4:Analysis and crystal structures of multiple base pair substitution and frameshift products with the adduct 1, N2-ethenoguanine[J]. Journal of Biological Chemistry, 2005, 280(33): 29750–29764. DOI:10.1074/jbc.M504756200 |
| [33] | Pogribny I, Raiche J, Slovack M, et al. Dose-dependence, sex-and tissue-specificity, and persistence of radiation-induced genomic DNA methylation changes[J]. Biochem Biophys Res Commun, 2004, 320(4): 1253–1261. DOI:10.1016/j.bbrc.2004.06.081 |
| [34] | Silva T D, Vidigal V M, Felipe A V, et al. DNA methylation as an epigenetic biomarker in colorectal cancer[J]. Oncology Letters, 2013, 6(6): 1687–1692. |
| [35] | Aypar U, Morgan W F, Baulch J E. Radiation-induced epigenetic alterations after low and high let irradiations[J]. Mutation Research, 2011, 707(1/2): 24–33. |
| [36] | 杨金兰, 柳李旺, 龚义勤, 等. 镉胁迫下萝卜基因组DNA甲基化敏感扩增多态性分析[J]. 植物生理与分子生物学学报, 2007, 33(3): 219–226. YANG Jin-lan, LIU Li-wang, GONG Yi-qin, et al. MSAP analysis of genome in radish under cadmium stress[J]. Journal of Plant Physiology and Molecular Biology, 2007, 33(3): 219–226. |
| [37] | 王鹤潼, 何蕾, 宋杰, 等. 改进MSAP-PCR技术应用于Cd胁迫下拟南芥DNA甲基化分析[J]. 农业环境科学学报, 2015, 34(8): 1618–1624. WANG He-tong, HE Lei, SONG Jie, et al. Assay of DNA methylation in Arabidopsis under Cd stress using improved MSAP-PCR technique[J]. Journal of Agro-Environment Science, 2015, 34(8): 1618–1624. DOI:10.11654/jaes.2015.08.027 |
| [38] | Pena L B, Barcia R A, Azpilicueta C E, et al. Oxidative post translational modifications of proteins related to cell cycle are involved in cadmium toxicity in wheat seedlings[J]. Plant Science, 2012, 196: 1–7. DOI:10.1016/j.plantsci.2012.07.008 |
| [39] | Amoroso A, Concia L, Maggio C, et al. Oxidative DNA damage bypass in Arabidopsis thaliana requires DNA polymerase λ and proliferating cell nuclear antigen 2[J]. Plant Cell, 2011, 23(2): 806–822. DOI:10.1105/tpc.110.081455 |
| [40] | Liu W, Zhou Q, Li P, et al. DNA mismatch repair related gene expression as potential biomarkers to assess cadmium exposure in Arabidopsis seedlings[J]. Journal of Hazardous Materials, 2009, 167(1/2/3): 1007–1013. |
| [41] | Giaginis C, Gatzidou E, Theocharis S. DNA repair systems as targets of cadmium toxicity[J]. Toxicology and Applied Pharmacology, 2006, 213(3): 282–290. DOI:10.1016/j.taap.2006.03.008 |
 2017, Vol. 36
2017, Vol. 36