文章信息
- 魏琳琳, 杨殿林, 侯萌瑶, 倪土, 李刚, 修伟明, 王慧, 赵建宁
- WEI Lin-lin, YANG Dian-lin, HOU Meng-yao, NI Tu, LI Gang, XIU Wei-ming, WANG Hui, ZHAO Jian-ning
- 氮高效转基因水稻OsNRT2.3b对土壤氨氧化细菌群落多样性的影响
- Effect of N-efficient transgenic rice on diversity and community structure of soil ammonia-oxidizing bacteria
- 农业环境科学学报, 2017, 36(6): 1149-1159
- Journal of Agro-Environment Science, 2017, 36(6): 1149-1159
- http://dx.doi.org/10.11654/jaes.2017-0116
文章历史
- 收稿日期: 2017-01-20
2. 东北农业大学资源与环境学院, 哈尔滨 150030
2. College of Resource and Environment, Northeast Agricultural University, Harbin 150030, China
从1996年到2015年,全球转基因作物累计种植面积达到空前的20亿hm2,转基因作物已经成为现代农业史上推广最为迅速的农作物[1],但转基因作物在给人们带来巨大经济效益的同时,其所带来的生态安全性问题也日益引起公众的广泛关注,尤其是对土壤微生物群落多样性的影响[2]。有研究表明,转基因棉花的种植能使土壤细菌和真菌的数量明显增加,使其群落组成发生变化[3-4],也有文献报道转基因作物的种植未对土壤微生物群落结构产生显著影响[5-6]。然而,要更为深入评价转基因植物对土壤微生物的影响,在研究分析整体微生物群落的过程中,还应研究转基因植物对土壤指示性微生物的影响[7]。
氨氧化细菌作为微生物生态学研究的指示性微生物,同时也是执行硝化作用第一步(将氨氧化为亚硝酸盐即硝化速率限制性步骤)的关键微生物[8-9],在土壤氮素循环中占有重要地位[10],但其群落组成容易受气候条件、土壤利用方式和植被类型等的影响[11-13],因此受到相关领域科学家的广泛关注[14-15]。
氮高效转基因水稻OsNRT 2.3b是利用转基因技术将水稻高亲和硝酸盐转运蛋白OsNRT 2.3b基因导入受体而获得的超表达材料。Fan等[16]和唐仲[17]研究发现,与常规稻相比,OsNRT 2.3b超表达株系中积累的氮素总量提高了21%,铵态氮吸收速率提高了12%,氮素利用效率提高了40%,单株产量提高了30%。由于土壤氮循环长期处于一个动态平衡的状态[18],氮高效转基因水稻的种植势必会从土壤中吸收更多的氮素,改变土壤氮素动态特征[11],进而可能影响土壤氨氧化细菌群落结构。
本研究采用PCR-DGGE技术,以氮高效转基因水稻OsNRT2.3b的两个不同株系N-04和N-08为对象,研究其土壤氨氧化细菌16S rDNA基因群落结构及多样性组成,为科学评价氮高效转基因水稻对土壤微生物的影响提供理论依据。
1 材料与方法 1.1 试验地概况与试验设计试验在农业部环境保护科研监测所网室内进行,种植小区四周及底部为混凝土结构,内部长、宽、高均为1 m,小区内土壤为天津市津南区未种植过作物的潮土,全磷含量1.19 g·kg-1,全氮含量0.96 g·kg-1,有机质含量24.55 g·kg-1,pH 8.21。
试验采用完全随机区组设计,设施氮和不施氮两种处理,5次重复。氮源(20 g·m-2)[19-20]为尿素[CO(NH2)2],其中50%用作基肥,50%作追肥,追肥在水稻分蘖后期施用。分别以磷酸二氢钾(P2O5:15 g·m-2)和硫酸钾(K2O:18 g·m-2)作为磷肥和钾肥,全部用作基肥。
1.2 供试材料试验所用水稻为氮高效转基因水稻OsNRT 2.3b的两个不同株系N-04和N-08及非转基因亲本日本晴Nipp,均由南京农业大学资源与环境科学学院植物营养分子生物学实验室提供。水稻种子于2015年5月8日播种于培养盘中,每穴5粒,于6月25日移苗,挑选长势一致的水稻苗,每个小区内移栽水稻20株。
1.3 土壤样品采集分别于水稻分蘖期(7月27日)、拔节期(9月8日)、抽穗扬花期(10月10日)和成熟期(11月12日)采集土样。采集土样时,去除表面杂草和枯枝落叶,用直径3.5 cm的土钻在距水稻主茎2 cm处取20 cm深的土样,每小区3个取样点。将各个小区的样品分别混合,置于-20 ℃冰箱,用于土壤氨氧化细菌群落多样性分析。
1.4 测定方法 1.4.1 土壤微生物总DNA提取本研究采用Mo Bio公司的Powerlyzer powersoil DNA isolation kit(Mo Bio laboratories,Solana Beach,CA,USA)试剂盒,取0.5 g鲜土置于Glass Bead Tube中,按操作说明逐步进行提取,将提取到土壤的DNA用1.5%的琼脂糖凝胶检测样品质量,并于-20 ℃保存。
1.4.2 PCR扩增采用巢式PCR(Nested PCR)方法扩增氨氧化细菌16S rDNA基因序列[21],引物及反应条件见表 1。第一轮PCR反应产物大小为465 bp,PCR反应体系为50 μL(两种引物各0.5 μL,Premix Ex Taq 25 μL,稀释2倍的土壤DNA模板5 μL,用灭菌水补足至50 μL);第二轮PCR反应产物大小为250 bp,PCR反应体系为50 μL(两种引物各0.5 μL,Premix Ex Taq 25 μL,第一轮PCR产物5 μL,用灭菌水补足至50 μL)。
PCR产物采用Bio-Rad公司的DcodeTM通用突变检测系统(Bio-Rad,USA),按照操作说明进行检测。主要步骤如下:浓度为8%的聚丙烯酰胺,变性梯度为40%~60%(100%变性剂含有7 mol·L-1尿素和40%(V/V)去离子甲酰胺),60 ℃预热,将30 μL PCR产物(与loading buffer预混好)加入胶孔,先在60 ℃、60 V恒定电压下预跑30 min,然后在60 ℃、150 V电泳6 h。电泳完毕后用SYBR Green Ⅰ(1:10 000)染色30 min,再用Gel Dox XR凝胶成像系统(Bio-Rad)进行观察与拍照,选取主要条带割胶回收。
1.4.4 条带纯化克隆及测序比对回收后的条带用不带GC夹子的338f和518r引物进行扩增,PCR产物经过电泳分析确定为单一条带后,采用Wizad R SV Gel and PCR Clean-Up system试剂(Progema,USA)纯化,并与载体pGEM-T Easy Vector(Progema,USA)连接转化(4 ℃培养8 h),挑取培养后的白色菌落接种到LB液体培养基中,37 ℃摇床培养8 h,阳性克隆送出测序。测序结果在NCBI上经Blast比对分析,获得相近典型菌株序列。
1.5 数据分析采用SAS 9.1.3(Tukey′s test)对试验数据进行分析,Quantity One 4.6.2软件进行数字化处理并进行聚类分析。土壤氨氧化细菌16S rDNA基因多样性采用香农-威纳指数(Shannon-Wiener index,H)、均匀度(Evenness index,En)和丰富度(Richness,S)来评价,其计算公式如下:
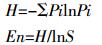
式中:H代表香农-威纳指数;Pi代表第i条带占总强度的比值;En代表均匀度指数;S代表丰富度指数。
2 结果与分析 2.1 土壤氨氧化细菌16S rDNA基因DGGE图谱分析DGGE结果表明,各生长期N-04、N-08和Nipp在施氮和不施氮处理条件下DGGE指纹图谱多为共有条带,只有少部分属于差异条带。在施氮条件下(图 1),N-04仅在拔节期与Nipp有2条差异条带(1B-2和1B-4);N-08在分蘖期、抽穗扬花期和成熟期与Nipp有4条差异条带(1A-3和1A-5;1C-4;1D-5)。

|
| 图 1施氮处理不同生长时期土壤样品DGGE指纹图谱 Figure 1DGGE profile in soil grown with different rice varieties under nitrogenous fertilizer application |
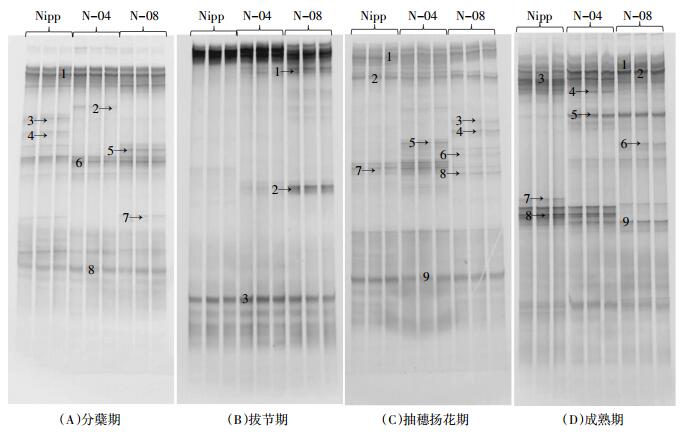
不施氮处理条件下(图 2),N-04在分蘖期、抽穗扬花期和成熟期与Nipp各有1条差异条带(2A-2;2C-5;2D-4);N-08在4个生长期与Nipp共有8条差异条带(2A-5和2A-7;2B-1;2C-3、2C-4、2C-6和2C-8;2D-6)。
|
| 图 2不施氮处理不同生长时期土壤样品DGGE指纹图谱 Figure 2DGGE profile in soil grown with different rice varieties without nitrogenous fertilizer application |
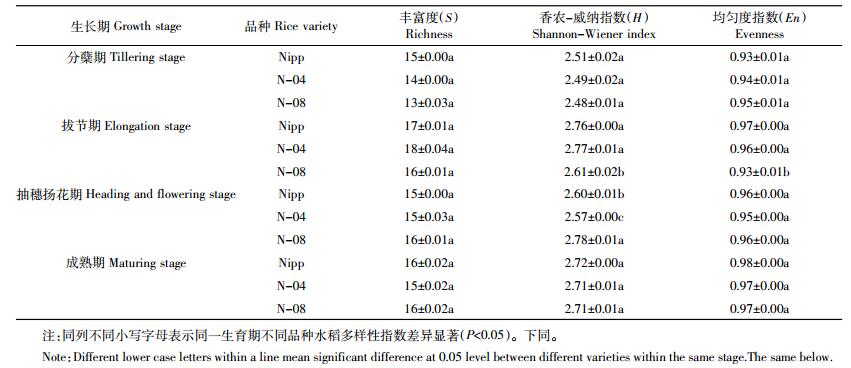
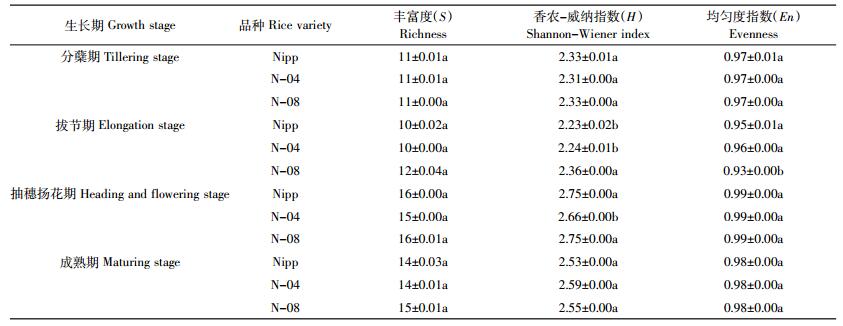
根据DGGE指纹图谱中每条条带的灰度比率,对种植N-04、N-08和Nipp的土壤氨氧化细菌16S rDNA基因丰富度指数(S)、香农-威纳指数(H)和均匀度指数(En)进行分析。结果发现,在施氮和不施氮条件下,N-04和N-08的土壤丰富度指数与Nipp在各生长期内均未出现显著差异(表 2和表 3)。
|
|
施氮条件下,N-08的土壤氨氧化细菌香农-威纳指数在拔节期时显著低于Nipp,而在抽穗扬花期显著高于Nipp,N-04的在分蘖期、拔节期和成熟期均未出现显著差异,仅在抽穗扬花期显著低于Nipp(表 2);不施氮条件下,N-08的土壤氨氧化细菌香农-威纳指数在分蘖期、抽穗扬花期和成熟期均未出现显著差异,仅在拔节期显著高于Nipp,N-04除抽穗扬花期显著低于Nipp外,其余各时期与Nipp均无显著差异(表 3)。
在施氮和不施氮条件下,N-04各生长期土壤氨氧化细菌均匀度指数与Nipp均无显著差异(表 2);N-08仅在拔节期显著低于Nipp,其余生育期未与Nipp产生显著差异(表 3)。
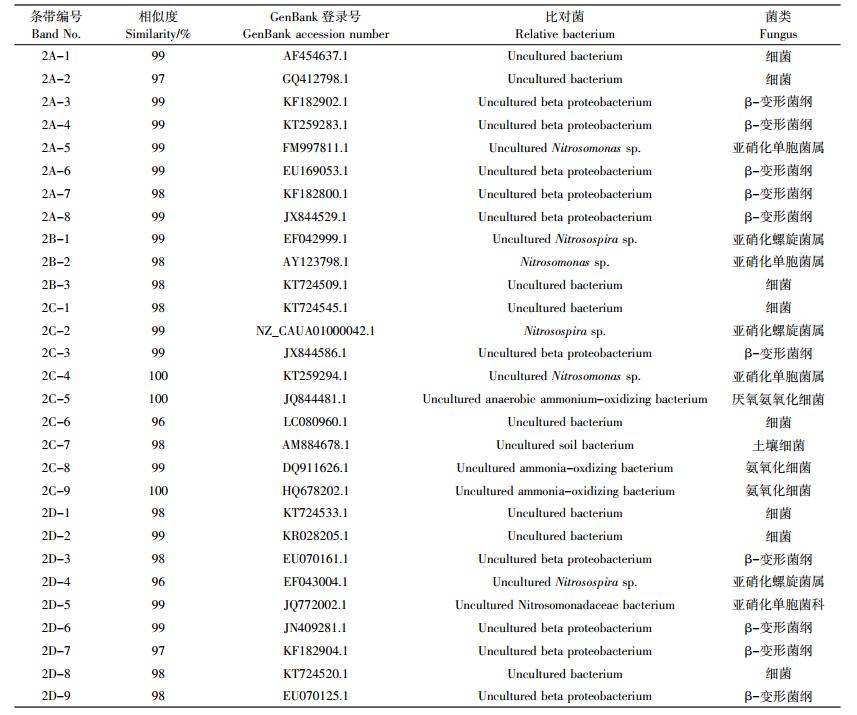
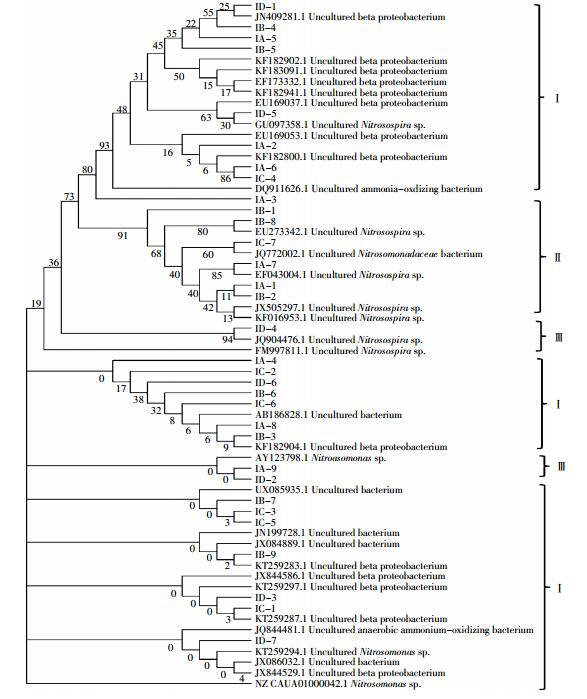
2.3 土壤氨氧化细菌16S rDNA基因测序结果及系统发育分析根据土壤氨氧化细菌DGGE指纹图谱(图 1和图 2)及条带灰度比率值大小,在施氮条件下选取32条条带,不施氮条件下选取29条条带,进行克隆测序,并经NCBI序列比对分析。结果发现各选取条带与已知序列相似度均在96%~100%之间(表 4和表 5),将测序获得的基因序列与Genbank其他相似序列对比,绘制系统发育树并进行系统发育分析(图 3和图 4)。

|
|
|
| Ⅰ:不可培养β-变形菌纲(Uncultured beta proteobacterium);Ⅱ:亚硝化螺旋菌属(Nitrosospira sp.);Ⅲ:亚硝化单胞菌属(Nitrosomonas sp.) 图 3施氮条件下土壤氨氧化细菌16S rRNA基因系统发育树 Figure 3Neighbour-journing tree depicting the phylogenetic relationships among the 16S rRNA sequences under nitrogenous fertilizer application |

|
| Ⅰ:不可培养β-变形菌纲(Uncultured beta proteobacterium);Ⅱ:亚硝化螺旋菌属(Nitrosospira sp.);Ⅲ:亚硝化单胞菌属(Nitrosomonas sp.);Ⅳ:不可培养的细菌(Uncultured bacterium) 图 4不施氮条件下土壤氨氧化细菌16S rDNA基因系统发育树 Figure 4Neighbour-journing tree depicting the phylogenetic relationships among the 16S rRNA sequences without nitrogenous fertilizer application |
从系统发育树可以看出,施氮处理条件下32个阳性克隆和不施氮处理条件下29个阳性克隆主要属于不可培养β-变形菌纲(Uncultured beta proteobacterium)、亚硝化螺旋菌属(Nitrosospira sp.)和亚硝化单胞菌属(Nitrosomonas sp.),其中N-08的优势属为亚硝化螺旋菌属(Nitrosospira sp.),N-04和Nipp的优势属均为不可培养β-变形菌纲(Uncultured beta proteobacterium)。
根据土壤氨氧化细菌DGGE指纹图谱及条带比对结果发现,在施氮处理条件下,各生长期内N-08属于β-变形菌门的亚硝化单胞菌属(Nitrosomonas sp.)的条带共有6条,属于β-变形菌门的亚硝化螺旋菌属(Nitrosospira sp.)的有3条,N-04属于上述菌属的条带共有6条,而Nipp有5条(表 4)。不施氮条件下,N-08属于β-变形菌门的亚硝化单胞菌属(Nitrosomonas sp.)和亚硝化螺旋菌属(Nitrosospira sp.)的条带共有6条,N-04则有5条,Nipp仅有1条属于亚硝化螺旋菌属(Nitrosospira sp.)的条带,未发现属于亚硝化螺旋菌属(Nitrosospira sp.)的条带,且该条带属于三种水稻品种共有条带(表 5)。由此可以看出,N-08和N-04属于亚硝化螺旋菌属(Nitrosospira sp.)和亚硝化单胞菌属(Nitrosomonas sp.)的条带数量显著高于Nipp。
3 讨论目前关于转基因水稻生态环境安全性评价的研究主要集中在基因漂移、靶标及非靶标生物的影响以及农业生态环境等方面[22]。转基因生物释放后是否对土壤微生物产生影响,近年已成为研究热点[23-25]。陈丽华等[26]研究发现抗真菌转基因水稻秸秆降解对土壤细菌数量的影响不显著,且转基因土壤样品与非转基因土壤样品中土壤细菌的丰富度、多样性指数、均匀度指数均不存在显著差异。但陈晓雯等[27]却发现转Cry1Ac基因和转Cry1Ab基因水稻在生长旺盛时期,土壤中细菌数量显著高于非转基因亲本水稻,不过这种影响的持续时间较短。陈丽华等[28]也证实了广谱抗真菌蛋白转基因水稻秸秆降解对土壤可培养真菌数和真菌群落结构有影响,但所产生的影响是短暂的、不持续的。
本研究发现,N-08的土壤氨氧化细菌香农-威纳指数在施氮条件下的抽穗扬花期和不施氮条件下的拔节期均显著高于Nipp,其余时期无显著差异。这与Jin等[29]的研究结果一致,其也发现转基因大豆的土壤氨氧化细菌香农-威纳指数仅在收获期显著增高。本研究还发现N-04与Nipp的土壤氨氧化细菌香农-威纳指数在施氮和不施氮处理条件下均无显著差异(除抽穗扬花期外),说明氮高效转基因水稻本身对土壤氨氧化细菌的影响也是短暂的,但不同施肥处理、不同生长期之间土壤氨氧化细菌香农-威纳指数差异较大。这与金凌波等[30]对转基因大豆土壤微生物群落水平的研究结果相似。Heuer等[31]对转T4溶菌酶基因土豆根际微生物群落结构的研究,也发现其主要与季节、种植地点和年份有关。
本文对土壤氨氧化细菌均匀度指数的研究发现,在两种供氮处理条件下,N-08与Nipp的土壤氨氧化细菌均匀度指数在分蘖期、抽穗扬花期和成熟期均无显著差异,仅在拔节期显著低于Nipp。邵婧鑫[32]、徐广惠[33]也证明了这一现象,其发现转基因大豆的土壤氨氧化细菌均匀度指数仅在生长旺盛时期显著低于非转基因品种。董莲华等[7]的研究也发现,转Bt+CpTI基因抗虫棉的土壤氨氧化细菌均匀度指数在花铃期显著低于非转基因材料,且其余时期均未发现明显差异,与本研究结果也一致,说明氮高效转基因水稻对土壤氨氧化细菌均匀度指数的影响是短暂的。
亚硝化单胞菌属(Nitrosomonas sp.)在硝化作用第一阶段亚硝化作用中起主导作用,可以促进NH4+氧化成NO2-,具有限制二氧化碳固定的能力[34-35],亚硝化螺旋菌属(Nitrosospira sp.)可作为硝化细菌,将亚硝酸盐氧化为硝酸盐[36-37]。本研究中施氮和不施氮处理下,氮高效转基因水稻(N-04和N-08)土壤中属于亚硝化螺旋菌属(Nitrosospira sp.)和亚硝化单胞菌属(Nitrosomonas sp.)的氨氧化细菌显著多于Nipp。因此,N-04和N-08的种植存在促进土壤中铵态氮氧化为硝态氮的可能。本研究对各生长期土壤硝态氮和铵态氮含量也进行了分析(研究结果未在文中列出),结果发现N-08和N-04在生长旺盛时期土壤硝态氮含量确实显著高于Nipp,而铵态氮含量显著低于Nipp,也进一步证明了氮高效转基因水稻的种植有利于土壤铵态氮向硝态氮的转化。
4 结论(1)各生长期内氮高效转基因水稻(N-04和N-08)和非转基因水稻(Nipp)在施氮和不施氮处理条件下DGGE指纹图谱多为共有条带。
(2)N-04和N-08的土壤氨氧化细菌香农-威纳指数和均匀度指数仅在个别时期与Nipp产生显著差异,说明氮高效转基因水稻本身对土壤氨氧化细菌群落多样性的影响是短暂的。
(3)氮高效转基因水稻(N-04和N-08)土壤中属于亚硝化螺旋菌属(Nitrosospira sp.)和亚硝化单胞菌属(Nitrosomonas sp.)的氨氧化细菌显著多于Nipp,说明氮高效转基因水稻的种植有利于土壤铵态氮向硝态氮的转化。
| [1] | JamesC. 2015年全球生物技术/转基因作物商业化发展态势[J]. 中国生物工程杂志, 2016, 36(4): 1–11. James C. Global biotech/GM crops commercialization development situation in 2015[J]. China Biotechnology, 2016, 36(4): 1–11. |
| [2] | 赖欣, 张永生, 赵帅, 等. 转基因大豆对土壤氨氧化细菌的影响[J]. 华北农学报, 2011, 26(1): 210–214. LAI Xin, ZHANG Yong-sheng, ZHAO Shuai, et al. Effect of transgenic soybean on amount and diversity of ammonia-oxidizing bacteria in rhizospheric soil[J]. Acta Agriculturae Boreali-Sinica, 2011, 26(1): 210–214. DOI:10.7668/hbnxb.2011.01.043 |
| [3] | Han C, Zhong W H, Shen W S, et al. Transgenic Bt rice has adverse impacts on CH4 flux and rhizospheric methanogenic archaeal and methanotrophic bacterial communities[J]. Plant and Soil, 2013, 369(112): 297–316. |
| [4] | Wang H X, Chen X, Tang J J. Influence of the straw decomposition of Bt transgenic rice on soil culturable microbial flora[J]. Acta Ecologica Sinica, 2004, 24(1): 89–94. |
| [5] | Song Y N, Su J, Chen R, et al. Diversity of microbial community in a paddy soil with cry1Ac/cpti transgenic rice[J]. Soil Science Society of China, 2014, 24(3): 349–358. |
| [6] | Zhu W J, Lu H H, Hill J, et al. 13C pulse-chase labeling comparative assessment of the active methanogenic archaeal community composition in the transgenic and nontransgenic parental rice rhizospheres[J]. Fems Microbiology Ecology, 2014, 87(3): 746–756. DOI:10.1111/fem.2014.87.issue-3 |
| [7] | 董莲华, 孟盈, 王晶. 转Bt+CpTI基因棉花对根际土壤细菌及氨氧化细菌数量的影响[J]. 微生物学报, 2014, 54(3): 309–318. DONG Lian-hua, MENG Ying, WANG Jing. Effects of transgenic Bt+CpTI cotton on rhizosphere bacteria and ammonia oxidizing bacteria population[J]. Acta Microbiologica Sinica, 2014, 54(3): 309–318. |
| [8] | Oved T, Shaviv A, Goldrath T, et al. Inflence of effluent irrigation on community composition and function of ammonia-oxidizing bacteria in soil[J]. Applied and Environmental Microbiology, 2001, 67(8): 3426–3433. DOI:10.1128/AEM.67.8.3426-3433.2001 |
| [9] | 董莲华, 杨金水, 袁红莉. 氨氧化细菌的分子生态学研究进展[J]. 应用生态学报, 2008, 19(6): 1381–1388. DONG Lian-hua, YANG Jin-shui, YUAN Hong-li. Research advances in moleclar ecology of ammonia oxidizing bacteria[J]. Chinese Journal of Applied Ecology, 2008, 19(6): 1381–1388. |
| [10] | Kowalchuk G A, Stephen J R. Ammonia-oxidizing bacteria:A model for molecular microbial ecology[J]. Annual Review Microbiol, 2001, 55(1): 485–529. DOI:10.1146/annurev.micro.55.1.485 |
| [11] | 张慧, 袁红朝, 朱亦君, 等. 不同利用方式对红壤坡地微生物多样性和硝化势的影响[J]. 生态学杂志, 2011, 30(6): 1169–1176. ZHANG Hui, YUAN Hong-chao, ZHU Yi-jun, et al. Microbial diversity and nitrification potential of sloping land red soil under different land use patterns[J]. Chinese Journal of Ecology, 2011, 30(6): 1169–1176. |
| [12] | Zhang L M, Wang M, Prosser J I, et al. Altitude ammonia oxidizing bacteria and archaea in soils of Mount Everest[J]. FEMS Microbiology Ecology, 2009, 70(4): 208–217. |
| [13] | 文都日乐, 李刚, 杨殿林, 等. 内蒙古呼伦贝尔草原土壤氨氧化细菌多样性及群落结构[J]. 应用生态学报, 2011, 22(4): 929–935. WENDU Ri-le, LI Gang, YANG Dian-lin, et al. Diversity and community structure of soil ammonia-oxidizing bacteria in Hulunbeier Grassland, Inner Mongolia[J]. Chinese Journal of Applied Ecology, 2011, 22(4): 929–935. |
| [14] | Mccaig A E, Philips C J, Stephen J R, et al. Nitrogen cycling and community structure of proteobacterial β-subgroup ammonia-oxidizing bacteria within polluted marine fish farm sediments[J]. Applied & Environmental Microbiology, 1999, 65(1): 213–220. |
| [15] | Nicolaisen M H, Ramsing N B. Denaturing gradient gel electrophoresis(DGGE) approaches to study the diversity of ammonia-oxidizing bacteria[J]. Journal of Microbiological Methods, 2002, 50(2): 189–203. DOI:10.1016/S0167-7012(02)00026-X |
| [16] | Fan X R, Tang Z, Tan Y W, et al. Over expression of a pH-sensitive nitrate transporter in rice increases crop yield[J]. Proceedings of the National Academy of Sciences, 2016, 113(26): 7118–7123. DOI:10.1073/pnas.1525184113 |
| [17] | 唐仲. 水稻高亲和硝酸盐转运蛋白基因OsNRT 2. 3a/b生物学功能分析[D]. 南京: 南京农业大学, 2012. TANG Zhong. Function analyses of high-affinity nitrate transporter gene OsNRT 2.3a/b in rice[D]. Nanjing:Nanjing Agricultural University, 2012. |
| [18] | 张春娜. 中国陆地土壤氮库研究[D]. 重庆: 西南农业大学, 2004. ZHANG Chun-na. Study on terrestrial soil nitrogen pool in China[D]. Chongqing:Southwest Agricultural University, 2004. |
| [19] | 晏娟, 尹斌, 张绍林, 等. 不同施氮量对水稻氮素吸收与分配的影响[J]. 植物营养与肥料学报, 2008, 14(5): 835–839. YAN Juan, YIN Bin, ZHANG Shao-lin, et al. Effect of nitrogen application rate on nitrogen uptake and distribution in rice[J]. Plant Nutrition and Fertilizer Science, 2008, 14(5): 835–839. |
| [20] | 叶永印, 张时龙. 氮肥施用技术对水稻产量及构成因素的影响[J]. 安徽农业科学, 2002, 30(3): 366–368. YE Yong-yin, ZHANG Shi-long. Effect of nitrogen application skill on the yield and yield components of rice[J]. Journal of Anhui Agricultural Sciences, 2002, 30(3): 366–368. |
| [21] | Zhang B, Sun B, Ji M, et al. Quantification and comparison of ammonia-oxidizing bacterial communities in MBRs treating various types of waste water[J]. Bioresource Technology, 2010, 101(9): 3054–3059. DOI:10.1016/j.biortech.2009.12.048 |
| [22] | Lu B R, Xia H. Environmental biosafety of transgenic plants:Research and assessment of transgene escape and its potential ecological impacts[J]. Chinese Bulletin of Life Sciences, 2011, 23(2): 186–194. |
| [23] | 王敏, 关潇. 不同转基因作物对土壤微生物的影响[J]. 山西农业科学, 2015, 43(2): 229–232, 243. WANG Min, GUAN Xiao. The effects of different genetically modified crops on soil microorganisms[J]. Journal of Shanxi Agricultural Sciences, 2015, 43(2): 229–232, 243. |
| [24] | 张艳军, 谢明, 彭德良. 转基因作物对土壤微生物的影响[J]. 应用生态学报, 2013, 24(9): 2685–2690. ZHANG Yan-jun, XIE Ming, PENG De-liang. Effects of transgenic crops on soil microorganisms:A review[J]. Chinese Journal of Applied Ecology, 2013, 24(9): 2685–2690. |
| [25] | Sohn S I, Oh Y J, Kim B Y, et al. Effect of genetically modified rice producing resveratrol on the soil microbial communities[J]. J Korean Soc Appl Biol Chem, 2015, 58(6): 795–805. DOI:10.1007/s13765-015-0106-y |
| [26] | 陈丽华, 吕新, 林碧娇, 等. 抗真菌转基因水稻秸秆降解对土壤细菌数量及多样性的影响[J]. 生态安全学报, 2012, 21(2): 125–129. CHEN Li-hua, LÜ Xin, LIN Bi-jiao, et al. Effects of straw decomposition of antifungal transgenic rice on the amount and diversity of soil bacteria[J]. Journal of Biosafety, 2012, 21(2): 125–129. |
| [27] | 陈晓雯, 林胜, 尤民生, 等. 转基因水稻对土壤微生物群落结构及功能的影响[J]. 生物安全学报, 2011, 20(2): 151–159. CHEN Xiao-wen, LIN Sheng, YOU Min-sheng, et al. Effects of transgenic rice on the structure and function of soil microbial communities[J]. Journal of Biosafety, 2011, 20(2): 151–159. |
| [28] | 陈丽华, 吕新, 林碧娇, 等. 广谱抗真菌蛋白转基因水稻秸秆模拟还田对土壤真菌群落结构的影响[J]. 中国生态农业学报, 2015, 23(1): 87–94. CHEN Li-hua, LÜ Xin, LIN Bi-jiao, et al. Effects of simulated straw return of transgenic rice expressing broad-spectrum antifungal proteins on soil fungal community structure[J]. Chinese Journal of Eco-Agriculture, 2015, 23(1): 87–94. |
| [29] | Jin Y, Qu J J, Ren G M, et al. Effects of transgenic DREB soybean Dongnong 50 on the diversity of soil ammonia-oxidizing bacteria[J]. Plant Protection, 2013, 14(7): 988–992. |
| [30] | 金凌波, 周峰, 姚涓, 等. 磷高效转基因大豆对根际微生物群落的影响[J]. 生态学报, 2012, 32(7): 2082–2090. JIN Ling-bo, ZHOU Feng, YAO Juan, et al. Effects of P-efficient transgenic soybean on rhizosphere microbial community[J]. Acta Ecologica Sinica, 2012, 32(7): 2082–2090. |
| [31] | Heuer H, Kroppenstedt R M, Lottmann J, et al. Effects of T4 Lysozyme release from transgenic potato roots on bacterial rhizosphere communities are negligible relative to natural factors[J]. Applied and Environmental Microbiology, 2002, 68(3): 1325–1335. DOI:10.1128/AEM.68.3.1325-1335.2002 |
| [32] | 邵婧鑫. SRTS对盐碱土氮素转化及AOB多样性的影响[D]. 哈尔滨: 东北农业大学, 2012. SHAO Jing-xin. Effects of SRTS on the nitrogen transition and the diversity of AOB in saline-alkaline soil[D]. Harbin:Northeast Agricultural University, 2012. |
| [33] | 徐广惠. RRS对根际土壤AOB与AOA群落多样性的影响[D]. 哈尔滨: 东北农业大学, 2010. XU Guang-hui. Effects of RRS on the diversity of AOB and AOA in rhizospheric soils[D]. Harbin:Northeast Agricultural University, 2010. |
| [34] | Marsh K L, Sims G K, Mulvaney R L, et al. Availability of urea to autotrophic ammonia-oxidizing bacteria as related to the fate of 14C-and 15N-labeled urea added to soil[J]. Biol Fert Soil, 2005, 42(8): 137–145. |
| [35] | Purkhold U, Wagner M, Timmermann G, et al. 16S rRNA and amoA-based phylogeny of 12 novel betaproteobacterial ammonia-oxidizing isolates:Extension of the dataset and proposal of a new lineage within the nitrosomonads[J]. Microbiol, 2003, 53(5): 1485–1494. |
| [36] | Angel R, Asaf L, Ronen Z, et al. Nitrogen transformations and diversity of ammonia-oxidizing bacteria in a desert ephemeral stream receiving untreated wastewater[J]. Microb Ecol, 2010, 59(1): 46–58. DOI:10.1007/s00248-009-9555-3 |
| [37] | Meincke M, Bock E, Kastrau D, et al. Nitrite oxidoreductase from Nitrobacter hamburgensis:Redox centers and their catalytic role[J]. Arch Microbiol, 2004, 158(2): 127–31. |
 2017, Vol. 36
2017, Vol. 36