2. 中国农业科学院农业环境与可持续发展研究所农业清洁流域创新团队, 北京 100081
2. Agricultural Clear Watershed Group, Institute of Environment and Sustainable Development in Agriculture, CAAS, Beijing 100081, China
抗生素被广泛应用于人类医疗、动物疾病防治以及畜牧养殖等领域[1-2],据统计2013年中国有53 800 t抗生素排放到环境中[3]。环境中大量未被利用的抗生素促进了抗生素抗性菌(Antibiotic-resistant bacteria,ARB)和抗性基因(Antibiotic resistance genes,ARGs)的产生,进一步造成环境中抗性致病菌数量增加从而危害人类健康[4-5]。如“超级细菌”鲍氏不动杆菌,从1970年的抗生素敏感菌(Antibiotic susceptible bacteria,ASB)通过基因水平转移(Horizontal gene transfer,HGT)进化成了当前具有多重抗性的内感染致病菌,构建了包含45种ARGs的基因岛[6]。2011年德国爆发的“毒黄瓜”事件,蔓延到欧洲9个国家,致使超过3000人受感染,33人死亡;引起本次疫情的O104:H4血清型肠出血性大肠杆菌是一种新型高传染性有毒菌株,该菌株携带氨基糖苷类、大环内酯类、磺胺类等抗生素的耐药基因,导致抗生素治疗无效[7]。因此世界卫生组织已把抗生素抗性列为本世纪危害人类健康的重大问题。即使在没有抗生素存在的条件下ARGs也可以维持很多代[8],并且可以通过HGT促进其在环境中的扩散[9]。
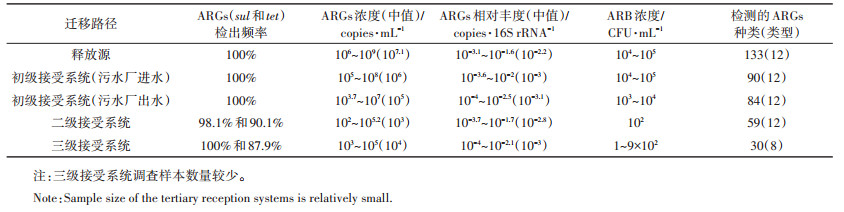
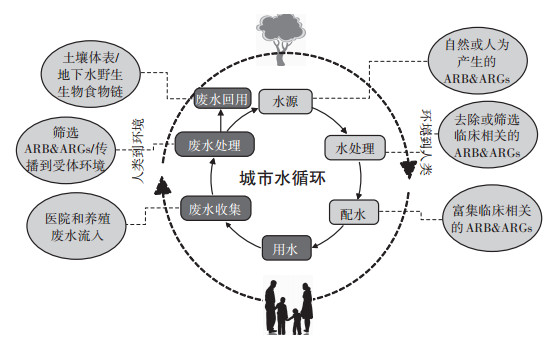
水环境是细菌的主要栖息地与繁殖介质,在ARB与ARGs传播方面扮演着重要的角色。抗生素生产厂废水、医院废水和养殖废水是环境中ARB的主要初始来源[10],部分经过污水处理厂处理后排放到地表水中,或回用进入农田环境中。Gao等[10]根据ARB和ARGs的迁移转化将水环境划分为四个系统,即释放源(医院、畜牧养殖废水)、初级接受系统(污水处理厂)、二级接受系统(地下和地表水体)和三级接受系统(河口、沿海和海洋),在各级水环境中的分布情况如表 1所示。城市水循环主要有两部分组成,一是废水的收集、输送与处理;二是地表水或地下水处理后供人类使用[11]。由城市水循环系统(图 1)可以看出,污水处理厂在城市水循环中发挥着重要作用。它既是各种污水的汇集地,又是污染地表水的源头,而地表水又是饮用水的重要来源[12]。因此污水处理厂却被认为是环境中ARB和ARGs的重要来源[13]。尽管污水处理厂出水中ARB和ARGs浓度相比进水已有大幅降低,但仍比自然水体和土壤高很多[2, 12]。综上,开展污水厂出水中ARB和ARGs的深度去除方法研究是十分必要的。本文综述了近年来去除ARB和ARGs的深度处理工艺研究,并阐述了相应的去除机理,在此基础上,对今后的研究重点和方向提出建议,以期为我国污水深度处理工艺的选择提供借鉴。
|
|
表 1 不同水环境中ARB和ARGs分布情况[10] Table 1 Distribution of ARB and ARGs in different aquatic environments[10] |
|
图 1 城市水循环的示意图以及可能对抗生素抗性选择、传播和控制至关重要的地点或过程[12] Figure 1 Schematic representation of the urban water cycle and of sites or processes potentially critical for antibiotic resistance selection, spread, and control[12] |
由于氯消毒成本较低,并能有效去除浮游细菌,因此被广泛应用于污水和饮用水处理[14]。氯消毒中的有效成分为自由氯(Free available chlorine,FAC),由HOCl和OCl-组成,HOCl的氧化能力要比OCl-强,HOCl的占比随pH的升高而降低。研究表明,当反应体系的pH从7.0升到8.0后,FAC对胞外ARGs的去除速率明显降低[15];同时,pH对胞内ARGs去除的影响不大。这是因为进入细胞内的FAC可能会受到细胞质pH条件(一般为7.2~7.8)的影响,致使其组成发生改变[15]。此外,FAC对不同嘌呤和嘧啶的氧化效率是不同的。它可以快速氧化鸟嘌呤(kCl2=2.1×104 mol-1· s-1)和胸腺嘧啶(kCl2=4.3×103 mol-1·s-1),但是与胞嘧啶(kCl2=66 mol-1·s-1)和腺嘌呤(kCl2=6.4 mol-1·s-1)的反应效率较低[16-17]。
Sullivan等[18]对6种含有tetW的ARB进行氯消毒实验,结果表明当氯浓度 > 0.5 mg·L-1,接触10 min后,6种ARB的去除(对数值log)均大于5;继而在黑暗条件下放置24 h,ARB的去除率可达到100%,但是却不能有效地去除ARGs。也有研究发现氯消毒虽然可以有效去除ARB,但却不能有效控制消毒后ARB的再生[19]。Yuan等[20]调查了氯消毒对污水厂二级出水中9种ARB的去除情况,发现在用Cl2剂量为15 mg·min-1·L-1时,即可有效去除大部分ARB,但是对红霉素抗性基因(ereA、ereB、ermA和ermB)和四环素抗性基因(tetA、tetB、tetM和tetO)的总去除率仅为60.0%和20.0%,即使Cl2剂量增加到300 mg·min-1·L-1,ARGs的去除率也没有明显变化。可见,虽然氯消毒可以有效杀灭ARB,但是却不能有效去除ARGs[21]。排放到环境中的ARGs可以通过水平转化机制被下游细菌吸收,从而使其获得抗生素抗性,继续对环境造成危害[22]。
不同抗生素的抗性菌对氯具有不同的耐受能力[23-24]。Yuan等[20]的研究表明,磺胺嘧啶抗性菌和红霉素抗性菌相比于其他抗性菌对氯有更强的耐受能力。这可能是由于某些ARGs对抗生素和氯有相似的抗性机理,如细菌体内用于排除抗生素的外排泵也可以把氯排到细菌体外,从而减小氯对细菌的伤害[20]。不同种属的细菌对氯的耐受能力也存在很大的差异,所以氯消毒会改变水体中微生物的群落结构。Jia等[25]发现嗜甲基菌属(Methylophilus)、多核杆菌属(Polynucleobacter)等能被有效去除,而假单胞菌属和食酸菌属(Acidovorax)却对氯有更强的耐受能力,从而使其成为优势种属;同时,处理后四环素类抗性基因和磺胺类抗性基因相对丰度降低,mex系列基因和BacA相对丰度增加,而两者主要存在于优势种属中,并由此导致总ARGs的相对丰度增加。Lin等[26]的研究也发现氯消毒会使部分ARGs[dfrA1、tetPB-03、tetPA、ampC- 04、tetA-02和erm(36)]呈现不同程度的富集,但是在其研究中总ARGs的相对丰度是降低的。Yoon等[15]的研究发现氯对抗性基因e-ampR的去除速率要比对e-kanR快,这可能是由于e-ampR中A-T碱基对数量更多的缘故(449 bp vs. 430 bp)。Lin等[27]的研究表明氯消毒可以降低ARGs的接合转移能力,当氯的浓度大于0.3 mg·L-1(接触时间0~48 h)时,细菌之间的ARGs接合转移频率可以降低到4.40×10-5甚至是检测限以下[27],而Guo等[28]的研究结果相反,在氯浓度为0.5~4 mg·L-1(接触时间10 min)时细菌间ARGs的接合转移被显著增强。这可能是由于氯消毒处理的差异造成的;此外Guo等的反应体系中产生的氯氨可能对ARGs接合转移起到了促进作用。
2 紫外照射紫外杀菌主要是通过直接和间接两种机制,直接机制是指紫外光(Ultraviolet,UV)可以穿过细胞壁、细胞膜和细胞质直接被核酸吸收,导致相邻的胞嘧啶或胸腺嘧啶形成二聚体;间接机制指细菌胞内或胞外的光敏物质吸收紫外光产生活性氧(Reactive oxygen species,ROS),活性氧氧化细胞膜、蛋白质、核酸和其他细胞物质以杀灭细菌[29]。短波紫外线(100~280 nm)主要通过直接机制来杀灭细菌,核酸物质对紫外光的最强吸收峰为λ=254 nm,此波长的杀菌效果最好[30]。Yoon等[15]采用UV254对一株抗性大肠杆菌进行紫外照射实验,研究发现细菌可培养数量和ARGs浓度都快速降低,但是紫外对细胞膜的破坏却是微乎其微的。紫外照射下,嘧啶碱基比嘌呤碱基更容易形成破坏核酸活性的产物[16]。McKinney等[31]的研究也发现紫外照射后可以扩增的ARGs的比例与ARGs中相邻胸腺嘧啶的数量呈很强的负相关。也有研究得出在紫外照射过程中ARGs变化与可移动遗传元件(Mobile genetic elements,MGEs)的变化具有很强的相关性[32]。
Zheng等[33]研究了紫外照射对污水厂二级出水中ARB和ARGs的去除情况,研究发现当紫外照射剂量为40 mJ·cm-2时,四环素类抗性基因的去除率仅为52.0%~73.5%;当照射剂量升高到160 mJ·cm-2时,去除率提高为79.7%~92.0%,对磺胺类抗性基因去除率为71.1%~78.1%;当紫外剂量达到80 mJ·cm-2时,即无法检测到可培养的细菌。McKinney等[31]的研究也发现破坏ARGs比灭活ARB需要更多的紫外剂量。这可能是由于紫外光被RNA和蛋白质遮挡从而减小ARGs被照射的几率[15]。Guo等[34]的研究也发现紫外照射可以有效去除污水厂二级出水中ARB,当照射剂量为5 mJ·cm-2时,可去除1.0~2.4(log值)的ARB,同时造成了部分抗生素的抗性菌的富集。但是也有学者发现紫外照射对抗性大肠杆菌和普通大肠杆菌的去除效果并没有明显差异[35-36]。Hu等[32]研究发现污水厂二级出水中气单胞菌属和盐单胞菌等优势菌群在紫外照射后几乎消失;而假单胞菌和芽孢杆菌等转而成为优势菌属,这可能与这些菌中含有抵抗紫外照射的基因和质粒有关。因此造成实际污水中ARB富集的原因可能是某些种属ARB对紫外照射耐受力较强,在照射后成为优势种属[32]。Guo等[36]也对紫外照射后ARB的光复活现象进行了研究,在照射剂量为5 mJ·cm-2时,抗性大肠杆菌的复活率最高为1.0%,但当升高紫外剂量时,光复活基本可以忽略不计。也有研究人员对紫外照射后ARB中ARGs的水平转移能力进行了研究,发现紫外照射可以显著降低ARGs的水平转移能力[27-28]。
3 臭氧氧化臭氧(O3)的还原电势为2.07 V,是一种强氧化剂。臭氧可以选择性地和有机分子反应,最易和亲核性物质反应,如碳碳双键、苯环和含有硫、磷、氮和氧的官能团[37]。所以一般认为臭氧首先氧化细菌磷脂层和脂多糖中的碳碳不饱和键、细胞膜和细胞壁中肽聚糖、脂类和蛋白质上的氨基酸,溶解细胞壁和细胞膜,以杀死细菌;然后细胞的通透性增强,多余的臭氧进入细胞,穿过细胞质与核酸物质发生反应使DNA失活[16-17, 37]。臭氧在溶液中会发生部分分解,生成羟基自由基(·OH),pH越高分解程度越大,·OH(E=2.8 V)是一种氧化能力比臭氧更强的氧化剂,可以无选择性地氧化有机和无机物质[37]。但是也并不是臭氧的分解程度越大越好,有学者就发现臭氧对ARB的去除能力随着pH的降低而升高,因为虽然·OH的氧化能力更强,但由于其氧化的无选择性,使得其作用于ARB的量并不高[38-39]。同氯消毒一样,臭氧对鸟嘌呤和胸腺嘧啶的氧化速率远大于腺嘌呤和胞嘧啶[16]。有研究发现细菌DNA中G-C碱基对占比越大,其在臭氧消毒后的污水中的相对丰度就越高[40]。
Kordatou等[41]研究发现臭氧可以有效地去除污水厂二级出水中的抗性大肠杆菌,在初始臭氧浓度为0.3 mg·L-1,pH=8条件下,接触15 min就可以将初始浓度为5.00×102 CFU·mL-1的红霉素抗性大肠杆菌和1.50×102 CFU·mL-1的羟苯乙酯抗性大肠杆菌完全去除。Sousa等[42]在臭氧流量为150 cm3·min-1的条件下处理污水厂二级出水,接触30 min可以去除3.9(log值)的异养菌和2.0(log值)的真菌以及2.0(log值)的16S rRNA和intI1,几乎可以达到对抗生素抗性基因的完全去除。但是3 d后,可培养的细菌、16S rRNA、intI1和ARGs(除了qnrS)都恢复到了处理之前的水平。当接触时间延长到60 min时,就不会出现细菌复活和再生现象。但是Alexander等[40]的研究却发现臭氧对二级出水中ARGs(vanA、blaVIM、ampC和ermB)的去除率为18.7%~99.3%;对ARB的去除效果为60.2%~98.9%,所以经过臭氧氧化后仍有相当数量的ARGs和潜在致病ARB排放到环境中,此外还发现臭氧氧化改变了出水中的微生物群落结构,生物多样性减少了50.0%。经过臭氧处理后,假单胞菌的相对丰度增加43%,而肠球菌和葡萄球菌的相对丰度却大幅降低,造成这种结果的原因之一就是细菌基因组中G-C碱基对的比例不同,因为A-T碱基对更容易和臭氧发生反应[40]。也有研究发现臭氧氧化可以有效去除ARB,但是对ARGs的去除效果不佳,所以应用于传统消毒处理的臭氧CT值,并不适用于对抗生素抗性的去除[38]。
菌株对臭氧耐受能力是由几个因素(如菌落聚集形态、生物膜和色素的产生等)共同决定的,抗生素抗性可能并不是一个主要因素。也有学者发现,ARGs可能会对抗性菌造成代谢负担,从而使其相比于同种属的ASB对臭氧的抵抗能力更弱[43]。Lüddeke等[44]研究发现抗性大肠杆菌和葡萄球菌相比于对应的ASB对臭氧有更强的耐受力,然而抗性屎肠球菌和粪肠球菌对臭氧的耐受能力比其他种类没有抗性的肠球菌还弱。这可能是由于其他种类肠球菌主要存在于水环境中,已发展出应对氧化的机制;此外有些肠球菌,如铅黄肠球菌产生的色素也可能在应对ROS氧化方面发挥着重要作用。Heß等[45]的研究也发现大部分抗生素的抗性大肠杆菌对臭氧的耐受能力与ASB没有差异甚至有些还低于ASB。
4 光氧化光/H2O2氧化主要是靠H2O2吸收光能后产生具有强氧化能力的·OH,从而达到去除水体中ARB和ARGs的目的。Zhang等[46]的研究发现UV/H2O2可以有效去除污水厂二级出水中的ARGs,在[H2O2]0=0.01 mol·L-1,pH=7.0条件下,照射30 min可去除1.55~ 2.32(log值)。然而Ferro等[47]的研究却发现在[H2O2]0= 20 mol·L-1条件下,需要紫外照射240 min才能将抗性大肠杆菌(C0=105 CFU·mL-1)去除到检测限(5 CFU· mL-1)以下,且照射300 min后溶液中的抗性基因blaTEM 仍不能去除。研究结果的差异可能是初始H2O2浓度的不同。然而,过高的H2O2浓度会使生成的·OH重新结合,导致H2O2的再生[48];所以有研究发现随着H2O2浓度的增加,对ARB去除效率的提高并不明显[49]。
在光芬顿反应中,光可以促进Fe2+的再生,从而减少了Fe2+的用量,并且光还可以使H2O2直接发生光解产生·OH[50-51]。反应体系中Fe2+/H2O2的摩尔比、H2O2浓度、溶液pH以及光照剂量都会影响到光芬顿氧化对ARB和ARGs的去除。Fiorentino等[52]研究了不同Fe2+/H2O2摩尔比条件和光照剂量下太阳光芬顿氧化对抗生素抗性大肠杆菌的去除情况,研究发现在Fe2+/H2O2=5:10,QUV=15 kJ·L-1条件下ARB的去除速率最快。Ferro等[49]的研究表明,相比于污水的固有pH,pH=4.0时ARB的去除速率更快。这是因为在较低pH条件下铁离子以光敏性[Fe(OH)]2+的形式存在,不易生成沉淀[53]。Karaolia等[54]的研究表明,光芬顿氧化虽然可以实现目标ARB的完全去除,但是24 h后铜绿假单胞菌(Pseudomonas aeruginosa)即可恢复到处理前一半的水平;处理后水中总DNA的去除率达到97.0%,但是其中肠球菌特异标记基因、抗性基因sul1和ermB的相对丰度仍然很高。实际污水中的自然有机物质(NOM)也会对ARB的去除产生复杂影响,如反应初始阶段NOM对·OH的竞争消耗,导致对ARB的去除较慢;但是NOM不但能加快Fe2+/Fe3+的循环,更能在光的作用下直接产生ROS,所以某个时间点后当实际污水产生了足够的ROS,对ARB的去除就会急剧增加[55]。
光催化反应指太阳光或紫外光被催化剂(比如TiO2、ZnO等)吸收产生ROS的过程,产生的主要ROS有O2-·、·OH、1O2。在光催化反应中光的类型、催化剂的种类和用量都会影响到对ARB和ARGs的去除。Venieri等[56]研究了掺杂金属的TiO2在模拟太阳光照射下对抗性肺炎杆菌(Pneumobacillus)的去除情况,TiO2在模拟太阳光照射下对ARB的去除效果为3(log值),掺杂质量分数为0.1% Mn和0.1% Co的TiO2对ARB的去除分别为4(log值)和6(log值),这是因为掺杂Mn或Co后可以扩展TiO2可吸收光的波长范围,并且同时在TiO2中掺杂两种金属会产生协同效果。Das等[57]的研究发现在太阳光照射下掺杂Fe的ZnO纳米颗粒对抗性大肠杆菌的去除效果要比ZnO纳米颗粒和TiO2纳米颗粒好,进一步的电镜表征观察到细菌细胞膜在反应过程中受到了严重的破坏。为了避免实际水处理过程中颗粒状催化剂的回收问题,近年来有学者采用TiO2薄膜来去除ARB及ARGs,结果表明在UV254照射剂量为6 mJ·cm-2和12 mJ·cm-2时,TiO2薄膜对ARB的去除分别为4.5~5.0(log值)和5.5~5.8(log值)。同时,革兰氏阳性菌相比于革兰氏阴性菌对光催化氧化有更强的耐受力,这可能是由于两者细胞壁结构的不同[58]。当照射剂量为120 mJ·cm-2时,可去除5.8(log值)mecA和4.7(log值)ampC,而当仅采用UV254照射时,对应的去除量仅为2.9(log值)和1.6(log值)[31],由此可见TiO2薄膜在对ARGs的去除中发挥着重要的作用[58]。
5 其他人工湿地是人为设计和建造的模拟自然湿地的污水处理工艺,按照布水和水流方式的不同主要分为表流湿地、水平潜流湿地和垂直潜流湿地(上流式或下流式)[59]。人工湿地对细菌的去除主要是通过化学效应和物理效应的共同作用(机械过滤、沉淀、氧化和有机质的吸附)以及生物去除作用(生物膜截留、自然死亡以及原生动物和线虫的捕食)[60]。环境温度、湿地水力条件、湿地类型以及湿地填料都会影响到对ARB和ARGs的去除效果。
在季节影响方面,Fang等[61]研究了组合表流人工湿地对14种ARGs的去除情况,结果表明在冬季和夏季对总ARGs的去除率分别为77.8%和59.5%。同时,沉积物与水中的ARGs具有很强的相关性,由于夏季温度较高促进了ARB的繁殖以及水力扰动,使得夏季的去除率较低。Vivant等[62]在采用表面流人工湿地处理污水厂出水时也得到了相同的结论。在湿地类型方面,Chen等[63]研究发现6种不同人工湿地对总ARGs的去除率为63.9%~84.0%,其中垂直潜流人工湿地对ARGs的去除效果最好,水平潜流人工湿地(不种植植物)次之,表面流人工湿地去除效果最差;去除结果的差异可能与各湿地中的吸附过滤、生化过程和氧化还原条件有关。此外,垂直潜流中水流方向也会造成去除结果的差异,有研究发现虽然上流式和下流式垂直潜流人工湿地对四环素类ARGs和intI1的去除率没有明显差异,但是前者出水中抗性基因的相对丰度比后者高[64]。在填料方面,火山岩比沸石对ARGs的去除效果更好,这种差异可能与两种填料的表面平均孔径和pH有关[65]。Nolvak等[66]的研究表明人工湿地对ARGs的去除还与对NO2--N、NH4+-N和有机物的去除效率有关。此外,人工湿地对不同ARGs的去除效果存在差异。例如,相比于tetW和tetO,人工湿地对tetM的去除效果较差,这可能是由于在tet 基因中tetM具有最广的宿主范围;并且tetM一般与结合子和转座子相关联[67]。虽然人工湿地可以有效去除ARB和ARGs,但是同时也会造成抗生素和重金属在湿地土壤中的累积,进一步增加其中ARGs的相对丰度[64]。
混凝也是去除ARB和ARGs的一种有效方式。常用的混凝剂有氯化铁和聚合氯化铁,其中氯化铁主要是通过双电层压缩和电荷中和来去除抗性基因,聚合氯化铁主要是通过网捕作用和电荷中和来去除ARGs。两种混凝剂去除机理的不同可能是造成对不同ARG去除效率差异的原因[68]。Li等[68]的研究结果表明氯化铁和聚合氯化铁这两种混凝剂均可有效去除污水中的ARGs。进一步的研究表明,混凝剂的类型和用量都会影响对ARGs和intl1的去除效果,不同ARG实现完全去除所需要的混凝剂用量也是不同的[68]。
近年来还有一些应用膜生物反应器(MBR)去除ARB和ARGs的研究。如Du等[69]的结果表明,膜生物反应器对tetG、tetW、tetX、sul1和intI1的去除量分别为0.67~3.11、1.88~4.61、1.71~4.73、1.36~3.84(log值)和1.32~3.15(log值),且ARGs的去除与16S rDNA的去除具有很强的相关性,据此推断MBR对ARGs的去除主要是通过减少生物量来实现的[69]。Cheng等[70]研究了厌氧膜生物反应器对3种ARB及其携带ARG质粒的去除效果,研究发现相比于未污染的膜,受污染的膜对质粒的去除效果更好。但是3种不同污染程度的膜对质粒的去除效率差别不大,这说明体积排阻和生物量并不是影响质粒去除效率的主要因素,对质粒的去除主要是靠污染层的吸附和滤饼层诱导产生了更严重的浓差极化[70]。未污染的膜和严重污染的膜对ARB的去除都可以达到5(log值),但亚临界污染的膜对ARB的去除效果,却随着操作压力的增加而降低。这是因为随着膜的污染操作压力增加,膜的孔径会变大并且细菌也会变形,从而使得滤出的细菌数量增加,导致去除率的下降。但是当膜严重污染时,膜孔被严重堵塞,再增加压力也不能致使变形的细菌通过[70]。
研究表明人工湿地可以有效去除污水中的ARB和ARGs,但是随着人工湿地的运行,基质中累计的ARB、ARGs和污染物质也会越来越多,势必会影响湿地中微生物和植物的生长繁殖,从而导致去除效果的下降。所以,对于抗生素抗性和人工湿地生态系统之间的相互影响还需要更加深入地研究。此外,人工湿地对ARB和ARGs的去除机理还不明确,人工湿地的设计、运行和管理对ARB和ARGs去除效果的影响还需进一步探索。把混凝和膜技术等物理处理技术应用到对ARB和ARGs的去除上,为阻止抗生素抗性的传播提供了新的思路,但是目前对这方面的研究报道较少,还有待更广泛、更深入地研究。表 2归纳总结了各深度处理技术对ARB和ARGs的去除效果,并详细比较了其优缺点。
|
|
表 2 各深度处理技术优缺点及处理效果 Table 2 Advantages and disadvantages of advanced treatment processes and removal efficiency of ARB and ARGs |
目前有关污水深度处理中的ARB和ARGs的去除研究,一方面是高效低耗、且能稳定运行的技术研发,另一方面是各技术去除ARB和ARGs的机理以及ARGs的传播扩散机制等的深入研究。不同的报道中ARB和ARGs的去除结果相差较大,甚至得出相反的结论,这可能是各研究中环境变量(如细菌的种类和丰度、水体温度、pH和化学成分以及当地的气候条件)的差异造成的。因此,在应用上述深度处理方法时,也要考虑到环境变量对处理结果的影响。今后,在污水深度处理中ARB和ARGs的去除研究方向包括:
(1)目前各工艺对ARB和ARGs去除的大多数研究中都是仅采用单一的技术,而很少开展两种以上组合工艺对ARB和ARGs去除的研究。考虑到各工艺对ARB和ARGs的去除机制存在差异,所以组合工艺可能会产生协同效果,以降低整体运行成本。所以开展组合工艺对ARB和ARGs的去除效果及机理研究是很有必要的。
(2)可移动遗传元件种类众多,且是造成ARGs水平转移的重要因素之一,然而已有的研究报道却非常有限。仅有的涉及到ARGs与可移动遗传元件关系的研究中也只是考察了Ⅰ类整合子。因此,今后的研究范围也应扩展到各种工艺对其他可移动遗传元件活性的影响,包括对整合子捕获ARGs能力的影响以及对ARGs水平转移的影响。此外,重金属抗性基因、抗杀虫剂基因等常和ARGs位于同一可移动遗传元件上,所以其综合去除以及相互关系的探究也是十分必要的。
(3)在对ARB的去除研究中,大多数研究仅涉及到对ARB去除效果的分析,以及在研究对不同ARB的去除差异时仅进行了表观分析,如细胞外壁的结构,基因组的大小或不同类型碱基对的数量等。但是细菌在应对外界变化时也会作出复杂的反应,所以在研究去除技术对ARB的去除时,也应进行逆转录组学的分析,以对不同ARB之间的去除差异有更深入的认识。深度处理后抗生素抗性富集的原因还没有一个统一的结论,处理出水中ARB群落结构的改变对于下游环境的影响研究也较为缺乏,因此这些方面还需要更加深入地研究。
(4)实时荧光定量PCR法是ARGs定量检测的主要方法,但是不同研究中所用的不同ARGs扩增子的大小不完全一致,考虑到较大的扩增子被破坏的概率也大,而较小的扩增子受到破坏的概率小,那么这可能是造成各处理方法对不同ARGs去除效率不同的一个原因。此外,即使ARGs可以被扩增,它也可能已经丧失了其抗性功能和传播扩散的能力。基于此,实时荧光定量PCR不但会造成对不同ARGs检测的偏差,还可能会低估深度处理工艺对ARGs的去除效果。因此未来还需在ARGs检测方法上进一步研究,寻求更为准确有效的检测方法。
| [1] |
Levy S B, Marshall B. Antibacterial resistance worldwide:Causes, challenges and responses[J]. Nature Medicine, 2004, 10(12): S122-S129. |
| [2] |
窦春玲, 郭雪萍, 尹大强. 污水处理厂抗生素抗性基因分布和去除研究进展[J]. 环境化学, 2013, 32(10): 1885-1893. DOU Chun-ling, GUO Xue-ping, YIN Da-qiang. Distribution and removal of antibiotic resistance genes in wastewater treatment plants[J]. Environmental Chemistry, 2013, 32(10): 1885-1893. DOI:10.7524/j.issn.0254-6108.2013.10.010 |
| [3] |
李红娜, 阿旺次仁, 李斌绪, 等. 兽用抗生素研究的文献计量学分析[J]. 农业环境科学学报, 2017, 36(11): 2297-2306. LI Hong-na, A Wang-ci-ren, LI Bin-xu, et al. A bibliometric assessment of research into antibiotics in poultry and livestock breeding[J]. Journal of Agro-Environment Science, 2017, 36(11): 2297-2306. DOI:10.11654/jaes.2017-0659 |
| [4] |
Berendonk T U, Manaia C M, Merlin C, et al. Tackling antibiotic resistance:The environmental framework[J]. Nature Reviews Microbiology, 2015, 13(5): 310-317. DOI:10.1038/nrmicro3439 |
| [5] |
罗义, 周启星. 抗生素抗性基因(ARGs)——一种新型环境污染物[J]. 环境科学学报, 2008, 28(08): 1499-1505. LUO Yi, ZHOU Qi-xing. Antibiotic resistance genes (ARGs) as emerging pollutants[J]. Acta Scientiae Circumstantiae, 2008, 28(8): 1499-1505. DOI:10.3321/j.issn:0253-2468.2008.08.002 |
| [6] |
Fournier P E, Vallenet D, Barbe V, et al. Comparative genomics of multidrug resistance in Acinetobacter Baumannii[J]. PLoS Genetics, 2006, 2(1): e7. DOI:10.1371/journal.pgen.0020007 |
| [7] |
苏建强, 黄福义, 朱永官. 环境抗生素抗性基因研究进展[J]. 生物多样性, 2013, 21(4): 481-487. SU Jian-qiang, HUANG Fu-yi, ZHU Yong-guan. Antibiotic resistance genes in the environment[J]. Biodiversity Science, 2013, 21(4): 481-487. |
| [8] |
Davies J, Davies D. Origins and evolution of antibiotic resistance[J]. Microbiology and Molecular Biology Reviews, 2010, 74(3): 417-433. DOI:10.1128/MMBR.00016-10 |
| [9] |
张瑞泉, 应光国, 丁永祯, 等. 广东西枝江-东江流域抗生素抗性基因污染特征研究[J]. 农业环境科学学报, 2013, 32(12): 2471-2479. ZHANG Rui-quan, YING Guang-guo, DING Yong-zhen, et al. Pollution characteristics of antibiotic resistance genes in Xizhijiang -Dongjiang River Basin, Guangdong Province, China[J]. Journal of Agro-Environment Science, 2013, 32(12): 2471-2479. DOI:10.11654/jaes.2013.12.021 |
| [10] |
Gao H, Zhang L, Lu Z, et al. Complex migration of antibiotic resistance in natural aquatic environments[J]. Environmental Pollution, 2018, 232(Suppl C): 1-9. |
| [11] |
Manaia C M, Macedo G, Fatta-Kassinos D, et al. Antibiotic resistance in urban aquatic environments:Can it be controlled?[J]. Applied Microbiology and Biotechnology, 2016, 100(4): 1543-1557. DOI:10.1007/s00253-015-7202-0 |
| [12] |
LaPara T M, Burch T R, McNamara P J, et al. Tertiary-treated municipal wastewater is a significant point source of antibiotic resistance genes into Duluth-superior harbor[J]. Environmental Science & Technology, 2011, 45(22): 9543-9549. |
| [13] |
佟娟, 魏源送. 污水处理厂削减耐药菌与抗性基因的研究进展[J]. 环境科学学报, 2012, 32(11): 2650-2659. TONG Juan, WEI Yuan-song. State-of-the-art removal of antibiotic resistance bacteria (ARB) and antibiotic resistance gene (ARG) in wastewater treatmentplants (WWTPs)[J]. Acta Scientiae Circumstantiae, 2012, 32(11): 2650-2659. |
| [14] |
Deborde M, von Gunten U. Reactions of chlorine with inorganic and organic compounds during water treatment-kinetics and mechanisms:A critical review[J]. Water Research, 2008, 42(1): 13-51. |
| [15] |
Yoon Y, Chung H J, Wen Di D Y, et al. Inactivation efficiency of plasmid-encoded antibiotic resistance genes during water treatment with chlorine, UV, and UV/H2O2[J]. Water Research, 2017, 123: 783-793. DOI:10.1016/j.watres.2017.06.056 |
| [16] |
Dodd M C. Potential impacts of disinfection processes on elimination and deactivation of antibiotic resistance genes during water and wastewater treatment[J]. Journal of Environmental Monitoring, 2012, 14(7): 1754-1771. DOI:10.1039/c2em00006g |
| [17] |
Sharma V K, Johnson N, Cizmas L, et al. A review of the influence of treatment strategies on antibiotic resistant bacteria and antibiotic resistance genes[J]. Chemosphere, 2016, 150(Suppl C): 702-714. |
| [18] |
Sullivan B A, Vance C C, Gentry T J, et al. Effects of chlorination and ultraviolet light on environmental tetracycline-resistant bacteria and tet (w) in water[J]. Journal of Environmental Chemical Engineering, 2017, 5(1): 777-784. DOI:10.1016/j.jece.2016.12.052 |
| [19] |
Fiorentino A, Ferro G, Alferez M C, et al. Inactivation and regrowth of multidrug resistant bacteria in urban wastewater after disinfection by solar-driven and chlorination processes[J]. Journal of Photochemistry and Photobiology B:Biology, 2015, 148(Suppl C): 43-50. |
| [20] |
Yuan Q B, Guo M T, Yang J. Fate of antibiotic resistant bacteria and genes during wastewater chlorination:Implication for antibiotic resistance control[J]. PLoS ONE, 2015, 10(3): e0119403. DOI:10.1371/journal.pone.0119403 |
| [21] |
郑吉, 周振超, 陈芳, 等. 3种常规消毒方法对磺胺类抗性基因削减效果的比较[J]. 环境科学, 2017, 38(4): 1497-1505. ZHENG Ji, ZHOU Zhen-chao, CHEN Fang, et al. Reducing effect of three disinfection technologies for sulfonamides resistance genes[J]. Environmental Science, 2017, 38(4): 1497-1505. |
| [22] |
Naquin A, Shrestha A, Sherpa M, et al. Presence of antibiotic resistance genes in a sewage treatment plant in Thibodaux, Louisiana, USA[J]. Bioresource Technology, 2015, 188(Suppl C): 79-83. |
| [23] |
Huang J J, Hu H Y, Tang F, et al. Inactivation and reactivation of antibiotic-resistant bacteria by chlorination in secondary effluents of a municipal wastewater treatment plant[J]. Water Research, 2011, 45(9): 2775-2781. DOI:10.1016/j.watres.2011.02.026 |
| [24] |
Murray G E, Tobin R S, Junkins B, et al. Effect of chlorination on antibiotic resistance profiles of sewage-related bacteria[J]. Applied and Environmental Microbiology, 1984, 48(1): 73-77. |
| [25] |
Jia S, Shi P, Hu Q, et al. Bacterial community shift drives antibiotic resistance promotion during drinking water chlorination[J]. Environmental Science & Technology, 2015, 49(20): 12271-12279. |
| [26] |
Lin W, Zhang M, Zhang S, et al. Can chlorination co-select antibioticresistance genes?[J]. Chemosphere, 2016, 156(Suppl C): 412-419. |
| [27] |
Lin W, Li S, Zhang S, et al. Reduction in horizontal transfer of conjugative plasmid by UV irradiation and low-level chlorination[J]. Water Research, 2016, 91(Suppl C): 331-338. |
| [28] |
Guo M T, Yuan Q B, Yang J. Distinguishing effects of ultraviolet exposure and chlorination on the horizontal transfer of antibiotic resistance genes in municipal wastewater[J]. Environmental Science & Technology, 2015, 49(9): 5771-5778. |
| [29] |
Al-Jassim N, Mantilla-Calderon D, Wang T, et al. Inactivation and gene expression of a virulent wastewater Escherichia coli strain and the nonvirulent commensal Escherichia coli DSM1103 strain upon solar irradiation[J]. Environmental Science & Technology, 2017, 51(7): 3649-3659. |
| [30] |
Chang P H, Juhrend B, Olson T M, et al. Degradation of extracellular antibiotic resistance genes with UV254 treatment[J]. Environmental Science & Technology, 2017, 51(11): 6185-6192. |
| [31] |
McKinney C W, Pruden A. Ultraviolet disinfection of antibiotic resistant bacteria and their antibiotic resistance genes in water and wastewater[J]. Environmental Science & Technology, 2012, 46(24): 13393-13400. |
| [32] |
Hu Q, Zhang X X, Jia S, et al. Metagenomic insights into ultraviolet disinfection effects on antibiotic resistome in biologically treated wastewater[J]. Water Research, 2016, 101(Suppl C): 309-317. |
| [33] |
Zheng J, Su C, Zhou J, et al. Effects and mechanisms of ultraviolet, chlorination, and ozone disinfection on antibiotic resistance genes in secondary effluents of municipal wastewater treatment plants[J]. Chemical Engineering Journal, 2017, 317: 309-316. DOI:10.1016/j.cej.2017.02.076 |
| [34] |
Guo M T, Yuan Q B, Yang J. Microbial selectivity of UV treatment on antibiotic-resistant heterotrophic bacteria in secondary effluents of a municipal wastewater treatment plant[J]. Water Research, 2013, 47(16): 6388-6394. DOI:10.1016/j.watres.2013.08.012 |
| [35] |
Huang J J, Hu H Y, Wu Y H, et al. Effect of chlorination and ultraviolet disinfection on teta-mediated tetracycline resistance of Escherichia coli[J]. Chemosphere, 2013, 90(8): 2247-2253. DOI:10.1016/j.chemosphere.2012.10.008 |
| [36] |
Guo M, Huang J, Hu H, et al. UV inactivation and characteristics after photoreactivation of Escherichia coli with plasmid:Health safety concern about UV disinfection[J]. Water Research, 2012, 46(13): 4031-4036. DOI:10.1016/j.watres.2012.05.005 |
| [37] |
Michael-Kordatou I, Karaolia P, Fatta-Kassinos D. The role of operating parameters and oxidative damage mechanisms of advanced chemical oxidation processes in the combat against antibiotic-resistant bacteria and resistance genes present in urban wastewater[J]. Water Research, 2018, 129(Suppl C): 208-230. |
| [38] |
Pak G, Salcedo D E, Lee H, et al. Comparison of antibiotic resistance removal efficiencies using ozone disinfection under different pH and suspended solids and humic substance concentrations[J]. Environmental Science & Technology, 2016, 50(14): 7590-7600. |
| [39] |
Zuma F, Lin J, Jonnalagadda S B. Ozone-initiated disinfection kinetics of Escherichia coli in water[J]. Journal of Environmental Science and Health, Part A, 2009, 44(1): 48-56. DOI:10.1080/10934520802515335 |
| [40] |
Alexander J, Knopp G, Dötsch A, et al. Ozone treatment of conditioned wastewater selects antibiotic resistance genes, opportunistic bacteria, and induce strong population shifts[J]. Science of the Total Environment, 2016, 559(Suppl C): 103-112. |
| [41] |
Michael-Kordatou I, Andreou R, Iacovou M, et al. On the capacity of ozonation to remove antimicrobial compounds, resistant bacteria and toxicity from urban wastewater effluents[J]. Journal of Hazardous Materials, 2017, 323(Part A): 414-425. |
| [42] |
Sousa J M, Macedo G, Pedrosa M, et al. Ozonation and UV254 nm radiation for the removal of microorganisms and antibiotic resistance genes from urban wastewater[J]. Journal of Hazardous Materials, 2017, 323(Part A): 434-441. |
| [43] |
Handel N, Schuurmans J M, Brul S, et al. Compensation of the metabolic costs of antibiotic resistance by physiological adaptation in Escherichia coli[J]. Antimicrobial Agents and Chemotherapy, 2013, 57(8): 3752-3762. DOI:10.1128/AAC.02096-12 |
| [44] |
Lüddeke F, Heβ S, Gallert C, et al. Removal of total and antibiotic resistant bacteria in advanced wastewater treatment by ozonation in combination with different filtering techniques[J]. Water Research, 2015, 69(Suppl C): 243-251. |
| [45] |
Heβ S, Gallert C. Sensitivity of antibiotic resistant and antibiotic susceptible Escherichia coli, Enterococcus and Staphylococcus strains against ozone[J]. Journal of Water and Health, 2015, 13(4): 1020-1028. DOI:10.2166/wh.2015.291 |
| [46] |
Zhang Y, Zhuang Y, Geng J, et al. Reduction of antibiotic resistance genes in municipal wastewater effluent by advanced oxidation processes[J]. Science of the Total Environment, 2016, 550: 184-191. DOI:10.1016/j.scitotenv.2016.01.078 |
| [47] |
Ferro G, Guarino F, Cicatelli A, et al. Beta-lactams resistance gene quantification in an antibiotic resistant Escherichia coli water suspension treated by advanced oxidation with UV/H2O2[J]. Journal of Hazardous Materials, 2017, 323: 426-433. DOI:10.1016/j.jhazmat.2016.03.014 |
| [48] |
Liu Y Q, He X X, Fu Y S, et al. Degradation kinetics and mechanism of oxytetracycline by hydroxyl radical-based advanced oxidation processes[J]. Chemical Engineering Journal, 2016, 284: 1317-1327. DOI:10.1016/j.cej.2015.09.034 |
| [49] |
Ferro G, Fiorentino A, Alferez M C, et al. Urban wastewater disinfection for agricultural reuse:Effect of solar driven AOPs in the inactivation of a multidrug resistant E-coli strain[J]. Applied Catalysis B-Environmental, 2015, 178: 65-73. DOI:10.1016/j.apcatb.2014.10.043 |
| [50] |
Von Sonntag C. Advanced oxidation processes:Mechanistic aspects[J]. Water Science and Technology, 2008, 58(5): 1015-1021. DOI:10.2166/wst.2008.467 |
| [51] |
Nidheesh P V, Gandhimathi R, Ramesh S T. Degradation of dyes from aqueous solution by Fenton processes:A review[J]. Environmental Science and Pollution Research, 2013, 20(4): 2099-2132. DOI:10.1007/s11356-012-1385-z |
| [52] |
Fiorentino A, Ferro G, Alferez M C, et al. Inactivation and regrowth of multidrug resistant bacteria in urban wastewater after disinfection by solar-driven and chlorination processes[J]. Journal of Photochemistry and Photobiology B-Biology, 2015, 148: 43-50. DOI:10.1016/j.jphotobiol.2015.03.029 |
| [53] |
Pliego G, Zazo J A, Garcia-Munoz P, et al. Trends in the intensification of the Fenton process for wastewater treatment:An overview[J]. Critical Reviews in Environmental Science and Technology, 2015, 45(24): 2611-2692. DOI:10.1080/10643389.2015.1025646 |
| [54] |
Karaolia P, Michael-Kordatou I, Hapeshi E, et al. Investigation of the potential of a membrane bioreactor followed by solar Fenton oxidation to remove antibiotic-related microcontaminants[J]. Chemical Engineering Journal, 2017, 310: 491-502. DOI:10.1016/j.cej.2016.04.113 |
| [55] |
Karaolia P, Michael I, Garcia-Fernandez I, et al. Reduction of clarithromycin and sulfamethoxazole-resistant Enterococcus by pilotscale solar-driven Fenton oxidation[J]. Science of the Total Environment, 2014, 468: 19-27. |
| [56] |
Venieri D, Gounaki I, Bikouvaraki M, et al. Solar photocatalysis as disinfection technique:Inactivation of Klebsiella Pneumoniae in sewage and investigation of changes in antibiotic resistance profile[J]. Journal of Environmental Management, 2017, 195: 140-147. |
| [57] |
Das S, Sinha S, Das B, et al. Disinfection of multidrug resistant Escherichia coli by solar-photocatalysis using Fe-doped ZnO nanoparticles[J]. Scientific Reports, 2017(7): 104. DOI:10.1038/S41598-017-00173-0 |
| [58] |
Guo C, Wang K, Hou S, et al. H2O2 and/or TiO2 photocatalysis under UV irradiation for the removal of antibiotic resistant bacteria and their antibiotic resistance genes[J]. Journal of Hazardous Materials, 2017, 323: 710-718. DOI:10.1016/j.jhazmat.2016.10.041 |
| [59] |
Vymazal J. Constructed wetlands for wastewater treatment:Five decades of experience[J]. Environmental Science & Technology, 2011, 45(1): 61-69. |
| [60] |
Vacca G, Wand H, Nikolausz M, et al. Effect of plants and filter materials on bacteria removal in pilot-scale constructed wetlands[J]. Water Research, 2005, 39(7): 1361-1373. DOI:10.1016/j.watres.2005.01.005 |
| [61] |
Fang H, Zhang Q, Nie X, et al. Occurrence and elimination of antibiotic resistance genes in a long-term operation integrated surface flow constructed wetland[J]. Chemosphere, 2017, 173: 99-106. DOI:10.1016/j.chemosphere.2017.01.027 |
| [62] |
Vivant A L, Boutin C, Prost-Boucle S, et al. Free water surface constructed wetlands limit the dissemination of extended-spectrum betalactamase producing Escherichia coli in the natural environment[J]. Water Research, 2016, 104: 178-188. DOI:10.1016/j.watres.2016.08.015 |
| [63] |
Chen J, Ying G G, Wei X D, et al. Removal of antibiotics and antibiotic resistance genes from domestic sewage by constructed wetlands:Effect of flow configuration and plant species[J]. Science of the Total Environment, 2016, 571: 974-982. DOI:10.1016/j.scitotenv.2016.07.085 |
| [64] |
Huang X, Zheng J, Liu C, et al. Removal of antibiotics and resistance genes from swine wastewater using vertical flow constructed wetlands:Effect of hydraulic flow direction and substrate type[J]. Chemical Engineering Journal, 2017, 308: 692-699. DOI:10.1016/j.cej.2016.09.110 |
| [65] |
Liu L, Liu C, Zheng J, et al. Elimination of veterinary antibiotics and antibiotic resistance genes from swine wastewater in the vertical flow constructed wetlands[J]. Chemosphere, 2013, 91(8): 1088-1093. DOI:10.1016/j.chemosphere.2013.01.007 |
| [66] |
Nolvak H, Truu M, Tiirik K, et al. Dynamics of antibiotic resistance genes and their relationships with system treatment efficiency in a horizontal subsurface flow constructed wetland[J]. Science of the Total Environment, 2013, 461: 636-644. |
| [67] |
Liu L, Liu Y H, Wang Z, et al. Behavior of tetracycline and sulfamethazine with corresponding resistance genes from swine wastewater in pilot-scale constructed wetlands[J]. Journal of Hazardous Materials, 2014, 278: 304-310. DOI:10.1016/j.jhazmat.2014.06.015 |
| [68] |
Li N, Sheng G P, Lu Y Z, et al. Removal of antibiotic resistance genes from wastewater treatment plant effluent by coagulation[J]. Water Research, 2017, 111: 204-212. DOI:10.1016/j.watres.2017.01.010 |
| [69] |
Du J, Geng J, Ren H, et al. Variation of antibiotic resistance genes in municipal wastewater treatment plant with A(2) O-MBR system[J]. Environmental Science and Pollution Research, 2015, 22(5): 3715-3726. DOI:10.1007/s11356-014-3552-x |
| [70] |
Cheng H, Hong P Y. Removal of antibiotic-resistant bacteria and antibiotic resistance genes affected by varying degrees of fouling on anaerobic microfiltration membranes[J]. Environmental Science & Technology, 2017, 51(21): 12200-12209. |
 2018, Vol. 37
2018, Vol. 37