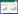文章信息
- 李艳, 谭文峰, 陈义, 吴春艳, 唐旭, 计小江
- LI Yan, TAN Wen-feng, CHEN Yi, WU Chun-yan, TANG Xu, JI Xiao-jiang
- 酶/蛋白质与土壤组分界面作用的研究进展
- Advancement in the research of enzyme binding on the interfaces of soil components
- 农业环境科学学报, 2018, 37(2): 205-214
- Journal of Agro-Environment Science, 2018, 37(2): 205-214
- http://dx.doi.org/10.11654/jaes.2017-1210
文章历史
- 收稿日期: 2017-09-05
- 修回日期: 2017-11-15
2. 华中农业大学资源与环境学院, 武汉 430070
2. College of Resources and Environment, Huazhong Agricultural University, Wuhan 430070, China
土壤酶大多数属于蛋白质,主要来自于微生物和植物根系的分泌物以及动植物残体,作为生态系统的重要组分之一,其是生态系统的生物催化剂和土壤有机体的代谢动力。这些酶参与碳、氮、磷、硫等元素的土壤生物化学循环,是土壤有机质转化的执行者和植物营养元素的活性库,其活性与土壤理化性质、土壤类型、施肥、耕作及其他农业措施等密切相关[1-2]。土壤酶可反映各种生物化学过程的方向和强度,常作为土壤肥力和自净能力的重要指标[3],由于其对环境因素改变的敏感性,也作为土壤生态系统变化的预警指标[4]。人类的活动也会直接或间接向土壤排放各种蛋白质,如转基因作物可通过根系分泌物及秸秆还田向土壤中释放Bt蛋白,一些致病蛋白(朊病毒、禽流感病毒等)也可通过动物进入土壤环境,威胁人类健康。土壤溶液中游离态酶/蛋白质含量较少,多以复合物的形式存在于土壤各组分界面上,包括矿物、有机质、矿物-有机质复合物等[5]。矿物、有机质、矿物-有机质复合物等组成了土壤中重要的化学界面,直接影响氮、磷等营养元素、有机物、生物大分子等的分布、形态、迁移转化以及生物可给性。酶/蛋白质在土壤界面上的吸附可能伴随其构象的变化,对于具有生物活性的酶/蛋白质分子,其活性和稳定性也可能在参与界面化学反应的过程中发生改变。酶/蛋白质分子构象的改变可能是影响其活性和稳定性的重要原因之一[6-7],但构象的改变对生物活性的影响机理仍不清楚,有待于进一步研究。
1 酶/蛋白质在土壤界面上的作用过程与机制 1.1 朊病毒、Bt毒素在土壤中的界面作用朊病毒是传染性海绵状脑病的病原,可通过感染动物的尿液、粪便、唾液或腐烂的尸体等进入土壤,与土壤组分间的作用可影响该致病蛋白的传播、生物可给性及在土壤中的持久性。朊病毒与模式氧化物(Al2O3、SiO2)的界面作用以静电力为主,两者的作用很大程度上取决于pH值和离子强度,土壤中朊病毒更倾向与带正电荷的铁铝氧化物发生作用[8]。腐植酸作为一种天然的聚阴离子,广泛存在于土壤与水体中,不仅与朊病毒存在较强的亲和力,且能够增加矿物对朊病毒的吸附能力[9]。与腐植酸作用后的朊病毒仍具有感染性,但可延缓疾病的感染速度[10]。为评估受感染动物尸体掩埋后在土壤中的传播水平及对生物的可及性,Jacobson等[11]采用蛋白酶处理受感染的脑组织匀浆,用合成的雨水作为淋洗剂,模拟其在5种不同土壤中的迁移能力,结果在洗出液中并未发现朊病毒,该致病蛋白基本保留在原始位置,可能成为感染其他生物的传染源。目前仍缺少有效灭活土壤中朊病毒的方法[12],Chesney等[13]最近研究发现,过硫酸盐对蛋氨酸和色氨酸残基具有氧化作用,可作为一种原位修复朊病毒污染土壤的新方法。
Bt毒素可由转基因作物通过根系分泌物、秸秆还田等方式释放进入土壤,与土壤各组分发生交互作用。Sander等[14]和Madliger等[15]研究了pH值和离子强度对Bt毒素在SiO2和多聚赖氨酸(PLL)上吸附的影响,结果表明静电作用力是主导Bt毒素吸附的驱动力,其在SiO2上的初始吸附速率和吸附量随pH值的升高而下降,与在PLL上的吸附结果相反;当Bt毒素与吸附界面带相同电荷时,吸附量随离子强度的降低而增加;吸附于SiO2上的Bt毒素仍具有较高的杀虫活性,在吸附解吸的过程中仍保持着较高的结构稳定性。土壤中的腐植酸作为天然有机物,与Bt毒素通过静电力、疏水作用而结合,两者间的作用受溶液条件(pH值、离子强度)、腐植酸极性和表面电荷等因素影响,即使当pH>6(大于Bt毒素等电点)且离子强度降低时,Bt毒素带正电荷的区域与带负电荷腐植酸间的静电作用仍促使Bt毒素的吸附量增加,腐植酸表面极性具有不均一性,疏水作用力使得Bt毒素对腐植酸非极性较高的区域具有较强亲和力[16]。
1.2 酶/蛋白质在土壤界面上的作用机制静电力、疏水作用在酶/蛋白质的吸附中起着重要的作用。酶/蛋白质是带有两性电荷的生物聚电解质分子,与荷电界面产生的静电作用是其吸附于亲水界面上的重要驱动力,且随环境pH值的变化而变化。带正电荷的溶菌酶能在静电引力的牵引下进入带相反电荷的蒙脱石层间并呈延展状态,且Al(OH)x的空间阻隔效应使得进入层间的溶菌酶随蒙脱石表面覆盖的Al(OH)x量的增加而减少[17]。在静电引力的驱动下,溶菌酶被腐植酸包被在三维网状结构中,阻碍了底物分子与活性位点的结合[18-22]。当酶/蛋白质与固体表面带相同电荷时,两者间的疏水作用也会促进酶/蛋白质的吸附,成为其吸附的另一种重要驱动力。层状硅酸盐矿物、有机质的憎水性基团可通过疏水力与酶/蛋白质的疏水性区域相结合。pH值为6时,胰岛素、核糖核酸酶与腐植酸表面均带负电荷,疏水力可通过克服分子间的静电排斥力促进腐植酸对酶的包被作用[23]。Bt毒素的吸附量随腐植酸极性的增加而降低[16],免疫球蛋白等的吸附量随界面疏水性的增强而增加[24],这均证明了疏水作用在蛋白质吸附过程中的重要性。
酶/蛋白质与土壤组分间的作用还包括配位交换、氢键、范德华力、阳离子交换等。Huang等[25]研究了酸性磷酸酶在不同粒径土壤颗粒及矿物上的吸附、解吸和酶活性,结果表明近三分之一吸附于针铁矿上的酸性磷酸酶主要通过配位交换作用结合,与高岭石间的作用以范德华力、氢键、疏水作用力为主,因而不易发生解吸。膨润土和高岭石对过氧化物酶、络氨酸酶的吸附量与阳离子交换量呈正相关[26],表明阳离子交换可能是两者相互作用的重要机制之一。
1.3 土壤酶/蛋白质界面反应的研究手段目前,研究酶/蛋白质与界面相互作用的方法主要包括表 1中的几种。酶/蛋白质的吸附量除采用差减法测定外,同位素示踪法、椭偏光度法、微晶天平、光波导模式谱也被用于吸附量的测定。同位素示踪法的优点在于其可用于多组分分析;椭偏光度法通过测定表面物质对偏振光反射时的状态变化而实现对表面吸附态物质质量和厚度的测定,根据表面吸附蛋白的浓度和吸附层厚度可以预测吸附态蛋白的状态,已用于各种表面对酶/蛋白质吸附和解吸过程的原位检测;微晶天平和光波导模式谱不仅可以测定蛋白质吸附量,还可提供蛋白质吸附、解吸的过程信息,近年来被广泛应用于酶/蛋白质与腐植酸相互作用的研究中。此外,吸附态酶/蛋白质结构的变化可采用傅里叶红外光谱、荧光光谱、核磁共振、圆二色谱等方法测定(详见4.2),酶/蛋白质吸附、解吸过程中热量的变化可采用微热量法和差示扫描量热法测定。
界面的理化性质主要包括表面结构、疏水性、荷电性等。界面的物理结构能够对酶/蛋白质的吸附产生影响,Lundqvist等[37]研究了脱水酶在具有不同曲率SiO2表面上的吸附,结果表明酶与较大直径的颗粒存在较强的交互作用,导致酶分子的二级结构发生较大的改变。受界面疏水性的影响,纤维蛋白原等在疏水的甲基表面比亲水的羟基表面吸附速率快[50]。McClellan等[51]研究发现牛血清白蛋白在疏水性界面形成的吸附层的厚度和密度大约为在亲水界面吸附条件下的一半,表明蛋白质分子与疏水界面间的作用更加强烈,蛋白质分子的变性程度更大。蛋白质在不同疏水性界面上的吸附结果表明,蛋白质的吸附量随界面疏水性的增加而增加(即-CH3>-OPh>-OCH3> -CONHCH3),且蛋白质与界面间的作用也随之增强[52-53]。
2.2 酶/蛋白质的理化性质酶/蛋白质在界面上的吸附还与本身分子量、形状、表面官能团、电荷、结构稳定性等有关。其中,酶/蛋白质结构的稳定性也是影响酶/蛋白质吸附的一个重要因素。溶菌酶、核糖核酸酶等硬蛋白内部稳定性高,在亲水表面的吸附量较小,与疏水表面相遇时产生的静电引力能使蛋白质分子结构发生改变,导致吸附量增加[28, 54];血清白蛋白等软蛋白内部稳定性较低,吸附时构象熵增加,几乎可以结合到所有物质表面[55],即使这类蛋白质与界面间存在静电排斥力时,也会产生一定的吸附量。
2.3 环境条件pH值、离子强度、酶/蛋白质浓度等溶液条件会影响酶/蛋白质的吸附量,以及在吸附界面上的排布和结构的变化程度。图 1为某种两性蛋白质在硅石/水界面上的吸附随pH值和蛋白质浓度变化的示意图。pH值对酶/蛋白质吸附的影响较大,Mori等[56]研究指出当溶液pH值为3时(低于BSA等电点),带正电荷的牛血清白蛋白快速吸附到云母表面,但分子之间的静电排斥力使得蛋白质在固体表面呈单分子吸附层,而当溶液pH值为6时(高于BSA等电点),蛋白质与云母表面之间的静电引力促使蛋白质在固体表面形成多层吸附。蛋白质浓度是影响其在界面上分布排列的另一个重要原因,当蛋白质浓度较低时,其在界面上的吸附速度较慢,能充分在界面上铺展,与界面间的作用力增强[53]。当牛血清白蛋白的浓度低于0.5 g·L-1且pH值为3~7时,该蛋白质在界面上形成均匀的单层吸附,当其浓度高于0.5 g·L-1且溶液pH值达到蛋白质等电点附近时(pH 5.1),牛血清白蛋白在亲水的硅石上的吸附量达到最大[57-58]。
|
| 图 1 两性蛋白质在硅石/水表面吸附示意图 Figure 1 A schematic graph to show adsorption of an amphoteric protein on silica/water interface |
离子强度对酶/蛋白质吸附量的影响主要取决于酶/蛋白质与界面所带电荷性质,当蛋白质与吸附界面带相同电荷时,两者之间存在静电排斥力,当增加溶液的离子强度时,静电排斥力减弱,促使酶/蛋白质在界面上的吸附量增加;相反,当酶/蛋白质与界面所带电荷不同时,离子强度的增加对两者的静电引力产生“屏蔽效应”,导致酶/蛋白质的吸附量降低[59-60]。
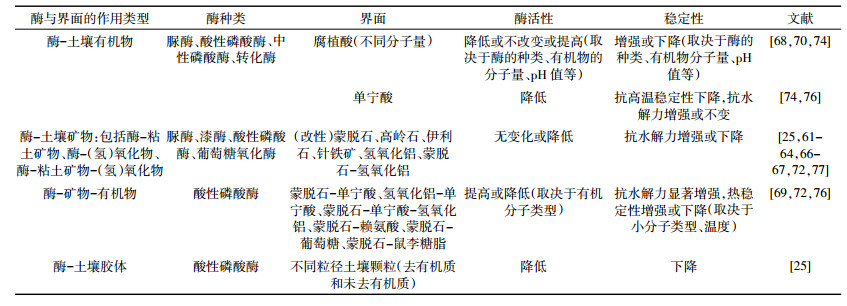
3 界面作用对酶/蛋白质生物特性的影响 3.1 界面作用对酶/蛋白质活性的影响酶/蛋白质进入土壤后,与有机质、矿物、矿物-有机质复合物、土壤胶体等结合,活性和稳定性变化的程度与酶/蛋白质种类、界面类型、环境条件等因素密切相关。吸附态酶的活性因其种类不同而不同,漆酶和过氧化酶吸附在高岭石、蒙脱石上可维持较高的活性,而吸附后的磷酸酶活性降低[61],葡萄糖氧化酶吸附于高岭石、十六烷基三甲胺-蒙脱石后,其活性较游离态酶没有明显的变化[62-64]。不同吸附界面对土壤酶/蛋白质活性的影响不同(表 2),如Bt蛋白吸附于高岭石后的杀虫活性显著高于蒙脱石,且杀虫活性还与土壤介质和蛋白质的比例有关[65]。各矿物抑制酸性磷酸酶和脲酶活性的顺序为蒙脱石>高岭石>伊利石[66],脲酶吸附于蒙脱石、氢氧化铝、蒙脱石-氢氧化铝复合物上的活性为蒙脱石>蒙脱石-氢氧化铝>氢氧化铝,蒙脱石表面由于氢氧化铝的覆盖,脲酶吸附量和活性均下降[67]。腐植酸对转化酶、酸性磷酸酶的活性表现为促进作用,对脲酶、酸性磷酸酶活性的影响与腐植酸本身的分子量、溶液pH值等因素有关。Marzadori等[68]研究了两种不同分子量的胡敏酸HA1(100~300 kDa)和HA2(10~20 kDa)对脲酶活性和稳定性的影响,结果表明pH为6.0时HA1显著抑制了脲酶的活性,在pH为7.0和8.0时不影响脲酶活性,而HA2在以上pH条件下对脲酶的活性均表现为抑制作用。Li等[20-21]研究结果表明腐植酸对溶菌酶的活性表现为抑制作用,且抑制的程度与腐植酸分子量、腐植酸/溶菌酶复合质量比、溶液pH值、离子强度有关。此外,氨基酸、葡萄糖、脂类等生物小分子能对酶与矿物间的相互作用产生影响,如赖氨酸、鼠李糖脂可以增强酸性磷酸酶在蒙脱石上的催化能力,使酶促反应能更加自发地进行,而葡萄糖分子则对酸性磷酸酶在蒙脱石上的活性产生了抑制作用[69]。
|
界面反应过程中,酶促反应动力学参数米氏常数(Km)和最大反应速率(Vmax)也可能发生变化。当土壤胶体对酶/蛋白质活性产生抑制作用时,通常表现为Vmax的降低和Km的增加,但研究结果不尽相同,如Marzadori等[70]研究发现,pH值为6~7范围内,腐植酸抑制了脲酶的活性,Vmax降低,Km降低,腐植酸增加了脲酶与底物之间的亲和力。土壤各组分通常会引起酶/蛋白质最适pH值的改变,当酶/蛋白质吸附于表面带负电荷的矿物上时,酶/蛋白质最适pH值通常向碱性方向移动[71]。
3.2 界面作用对酶/蛋白质稳定性的影响由于土壤胶体、矿物表面的保护作用,固定化酶能在一定程度上对抗蛋白酶的水解作用,并具有一定的热稳定性和化学稳定性,能够抵御高温、极端pH值的破坏,免遭光照、蛋白酶及微生物等的分解作用[72-73],这是土壤酶普遍具有较高稳定性的原因。被高岭石吸附的Bt蛋白经长期存放后仍保持较高的杀虫活性,表明高岭石对该蛋白具有稳定作用[65]。大分子量的胡敏酸与脲酶的结合有利于酶稳定性的提高,且稳定性随pH值的升高而增强[74]。Marzadori等[68]指出高分子量的胡敏酸可以提高脲酶抵抗蛋白酶的水解作用及金属离子Cu2+、Hg2+的抑制作用。酶被粘土矿物吸附后,尤其是在羟基铝存在的条件下,固定态酶的抗水解能力会更显著[75]。酸性磷酸酶与单宁酸作用后置于60 ℃下的活性显著低于游离态酶,但经水解酶处理后的吸附态酶活性较游离态酶高,且当Fe3+或MnO2与单宁酸共存时,酸性磷酸酶抗水解的能力显著增加[76]。与氢氧化铝-单宁酸、氢氧化铝-单宁酸-蒙脱石作用后,酸性磷酸酶抗水解酶的能力增强,且随时间的延长效果更加显著,这是因为酶/蛋白质通过吸附或包被在有机物-矿物胶体的内部或表面,致使酶/蛋白质构象的变化受到束缚,同时阻碍了水解酶与酶/蛋白质活性中心的接触[72]。Huang等[25]研究了不同温度(15~85 ℃)下,几种去有机质和未去有机质处理的土壤胶体对酸性磷酸酶活性的影响,结果发现不同温度下吸附态酸性磷酸酶的活性均低于游离态酶,且由于有机质的保护作用,未去有机质处理的土壤胶体对酸性磷酸酶在不同温度下活性的抑制作用低于去有机质处理的土壤胶体。
4 界面作用对酶/蛋白质构象的影响 4.1 酶/蛋白质构象变化的影响因素酶/蛋白质在界面上的吸附通常伴随构象的改变,主要表现为二、三级结构的微扰导致α-螺旋结构含量降低,螺旋结构的缺失可能增加多肽链间的柔性,导致酶/蛋白质分子构象熵的增加[78-79]。酶/蛋白质构象的变化受其种类及浓度、吸附界面表面特性、溶液条件等因素的影响[80]。首先,不同酶/蛋白质的化学性质和结构的稳定性存在一定差异,因而吸附后其结构的改变程度也可能不同。Haynes等[81]研究发现,牛奶乳清白蛋白和鸡蛋白溶解酵素吸附于憎水性聚苯乙烯表面后,两种蛋白质均发生显著的变性;当两者分别吸附于亲水的α-Fe2O3表面后,前者几乎完全变性,而后者由于良好的结构稳定性仅失去一小部分二级结构。蛋白质浓度较低时,与界面间的作用力较强,导致构象变化大于高浓度下的蛋白质溶液。其次,吸附界面的化学组成、物理结构(如形状、尺寸、比表面积)、疏水性也会影响酶/蛋白质的构象。纤维蛋白原、血清白蛋白与含不同类型基团载体间的作用力随载体表面疏水性的增加而增强,同时蛋白质的构象变化也相应增大[53]。不同直径的SiO2载体对酶构象的影响不同,脱水酶和纤维蛋白原的二级结构变化均随颗粒直径的增加而增大,脱水酶的三级结构则与颗粒大小无关,牛血清白蛋白结构的无序性也随颗粒直径的增加而增加[37]。蛋白质构型的改变程度可通过吸附于膨胀性粘粒矿物后层间距的扩张程度来判断,如直径为8.6 nm的葡萄糖氧化酶分子进入蒙脱石层间,仅使矿物层间距增加到2.3 nm[82],层间距0.96 nm的脱水钠蒙脱石吸附直径为5.0 nm的芳基硫酸酯酶后,层间距只有1.90 nm[83],表明蛋白质分子“变扁”或结构发生变形。此外,当pH值、离子强度有利于蛋白质在矿物表面上的吸附时,蛋白质呈延展状态分布于整个表面[84]。几种表面具有不同电荷量及疏水特性的矿物对葡萄糖苷酶构象影响的结果表明,除静电作用力外,疏水作用也是引起酶/蛋白质构象变化的一个主要原因[82]。
4.2 酶/蛋白质构象的主要研究手段目前,测定酶/蛋白质结构的方法可以分为两类:一可以测定酶/蛋白质高分辨率的结构特征,如X-射线衍射、小角中子衍射法、核磁共振;二能够测定酶/蛋白质二、三级结构及微环境的变化,如圆二色谱、红外光谱、荧光光谱等(部分方法见表 1)。Long等[36]采用固态核磁研究了唾液蛋白(分子量约为5 kDa)在羟磷灰石上的吸附,揭示了水分子在稳定吸附态肽链α结构上的作用。圆二色谱能提供酶/蛋白质二级结构(α-螺旋、β-折叠、自由卷曲)的信息,但其局限性不仅在于不能测定特定区域结构的变化,且在特定波长下较大的吸附剂分子存在一定的散射。Billsten等[85]发现,硅纳米颗粒的直径不超过20 nm才能进行圆二色谱测定。学者采用圆二色谱研究了几种具有不同结构稳定性的球状蛋白质在带电硅石纳米颗粒上的吸附[85-88],研究表明“软”蛋白质(牛血清白蛋白、肌球素、血红素)结构变化的程度最大,而“硬”蛋白(细胞色素C、免疫球蛋白G、核糖核酸酶)与亲水表面接触,伸展的程度较小。圆二色谱与核磁共振相结合可为酶/蛋白质在界面反应过程中的结构变化提供更多信息[37]。红外光谱适用于研究固液界面上酶/蛋白质结构的改变,唯一局限性在于颗粒直径不能超过10 μm。采用衰减全反射红外光谱可研究酶/蛋白质在固液界面上的吸附动力学和酶/蛋白质结构的变化,其中吸附动力学可通过流动或静置状态测定蛋白质酰胺键随时间的变化。荧光光谱和圆二色谱都是较成熟的溶液酶/蛋白质构象的测定方法,由于大多数酶/蛋白质都含有能发射荧光的色氨酸、酪氨酸及苯丙氨酸,其中色氨酸荧光强度最大,且对微环境的变化较敏感,因而常作为内源荧光探针研究酶/蛋白质的构象。
原子力显微镜可以分析酶/蛋白质分子在固相表面上的取向,获得酶/蛋白质的空间分布情况。此外,和频振动光谱是近年来发展起来的一项界面监测手段,被广泛应用于酶/蛋白质在固体材料上构象变化的测定,该方法具有界面单分子层的敏感性,可有效地对固体材料界面上酶/蛋白质分子的构象进行表征。
5 酶/蛋白质构象的变化与活性的关联性在土壤矿物、腐植酸等组分上的吸附可能引起酶/蛋白质构象的改变,尤其对于一些软蛋白,吸附于疏水性界面后会导致构象发生较大的变化。酶/蛋白质构象的改变可能会引起其活性和稳定性的变化,且酶/蛋白质活性的变化可能随构象变化的增加而增加,如溶菌酶、脱水酶与较大粒径SiO2作用后,α-螺旋结构减少、酶活性损失严重,而吸附于具有大曲率的小粒径SiO2上时,能最大限度地维持原有结构,从而保持较高的活性[37-38]。酶/蛋白质构象及活性的变化与吸附界面的表面特性密切相关,Noinville等[6]采用衰减全反射红外光谱发现表面带正电荷的α-胰凝乳蛋白酶吸附于带负电荷的亲水性界面时,仅5%的β-折叠结构发生损失,对酶活性的影响较低,而α-胰凝乳蛋白酶吸附于疏水性载体时,其二、三级结构均发生了较大的变化,酶活性受到抑制,脂肪酶通常以较为紧实的构象存在于溶液中,吸附于疏水性载体后则形成一种“敞开”式的构象异构体,允许底物分子接近酶的活性位点,使得酶活性增加。酶/蛋白质构象对其活性的影响可能还与本身的性质有关,木瓜蛋白酶和α-胰凝乳蛋白酶分别吸附于Teflon界面后,前者的α-螺旋结构比例降低,而后者的α-螺旋结构含量升高,且后者活性下降的程度更大[89]。
也有研究表明吸附态酶/蛋白质活性的变化不是由酶/蛋白质构象变化所导致的,空间位阻也是造成活性变化的一个重要原因。如α-胰凝乳蛋白酶在蒙脱石表面吸附后(pH为5~7)活性显著下降,该酶活性的降低可能是由催化活性位点氨基酸中的氨基和亚氨基面向带负电荷的硅氧基表面造成空间位阻所引起的[28]。
6 展望 6.1 固定化酶/蛋白质在土壤中的应用游离态酶/蛋白质在环境中的稳定性差,易发生化学、生物降解,酶/蛋白质的固定化通常使其稳定性得以增强,防止酶的损失且可反复使用。酶/蛋白质固定的机制主要包括吸附、捕获、微囊化、离子交换、交联作用、共聚作用[90]。将固定化酶/蛋白质应用于环境污染物的降解是研究热点之一,如氧化还原酶类(如过氧化物酶、漆酶等)能够降解多环芳烃类、酚类化合物、有机磷农药、偶氮类染料等环境有机污染物。固定化酶/蛋白质在水处理(尤其含酚废水)方面已有较多报道[91-92]。在土壤污染修复中,固定化酶在处理土壤持久性有机污染物、除草剂等方面也得到了广泛的应用。代云容等[93]采用可生物降解高分子外消旋聚乳酸为原料,利用乳液电纺技术将漆酶包埋固定于纳米纤维中,用于土壤多环芳烃有机污染物的修复。研究者以海藻酸钠或聚阴离子纤维素为载体,对阿特拉津降解酶和莠去津降解酶进行固定并优化了固定条件,结果表明固定化酶在土壤除草剂污染修复中能发挥较好的作用[94-96],葛世杰[97]还对固定化酶修复阿特拉津污染土壤的生态安全性进行了评价,发现固定化酶对土壤微生物活性和土壤多样性无不良影响。
固定化酶的催化能力因载体种类与特性、固定化条件不同而存在差异,可固定酶的载体材料包括多孔玻璃球、粘土矿物、海藻酸盐、有机凝胶等,但在实际土壤修复过程中,相对廉价的蒙脱石、高岭石、二氧化硅、羟磷灰石等天然载体可能成为首选。研究不同固定化载体对酶/蛋白质吸附和生物特性的影响,以节约经济成本为原则,筛选能保持酶/蛋白质较高活性和稳定性的载体,实现该技术在土壤污染修复中的应用和推广。
6.2 分子动力学模拟技术在土壤中的应用酶/蛋白质在土壤界面上的吸附是一个极其复杂的过程,不仅关系到它们在土壤中的迁移转化,也影响其生物活性的发挥。酶/蛋白质吸附于界面后的构象变化一直是人们关注的焦点,现有的技术虽能够提供一些蛋白质结构变化的信息,但多局限于蛋白质二级结构,对于三级结构及更复杂结构信息的获取较难。计算机分子模拟技术(如分子动力学模拟)在研究蛋白质构象变化方面有着不可替代的优势,它可借助合理的分子结构模型和物理原理,从微观水平上揭示蛋白质吸附的动态过程和机理。分子动力学模拟在药物小分子与蛋白质互作方面具有较多的应用,何文英等[98]采用该技术发现了胡椒碱与牛血清白蛋白的键合模式主要是疏水作用,该结论也通过热力学参数的结果得到了进一步验证。唐志华[99]和张晟瑞等[100]采用分子模拟技术与光谱学相结合的手段研究了硫酸沙丁胺醇、抑菌剂磺胺二甲嘧啶与人血清白蛋白的作用,可为酶/蛋白质结构变化提供更丰富的信息。目前,有研究者采用该技术模拟蛋白质在土壤各组分上的界面作用,如Chapron等[101]采用分子动力学模型揭示了致病性朊病毒(PrPsc 92-138)与模式矿物叶腊石间的深层作用机制,即通过蛋白质肽链上质子化的赖氨酸或组氨酸与粘土矿物表面的硅氧烷空穴中的氧原子形成的多个氢键而结合。借助于该模型,研究者发现与高岭石、蒙脱石、针铁矿发生界面作用时,免疫球蛋白Gb1结构几乎没有变化,而与水钠锰矿发生作用时,蛋白质结构被破坏[102]。虽然该技术在模拟蛋白质与土壤组分互作方面已取得了一定进展,但多应用于一些模式蛋白和模式矿物间的作用,对于土壤中多元复杂界面的吸附仍具有一定的局限性,还有待于进一步挖掘。
| [1] |
向泽宇, 王长庭, 宋文彪. 草地生态系统土壤酶活性研究进展[J]. 草业科学, 2011, 28(10): 1801-1806. XIANG Ze-yu, WANG Chang-ting, SONG Wen-biao, et al. Advances on soil enzymatic activities in grassland ecosystem[J]. Pratacultural Science, 2011, 28(10): 1801-1806. |
| [2] |
王理德, 王方琳, 郭春秀, 等. 土壤酶学研究进展[J]. 土壤, 2016, 48(1): 12-21. WANG Li-de, WANG Fang-lin, GUO Chun-xiu, et al. Review:Progress of soil enzymology[J]. Soils, 2016, 48(1): 12-21. DOI:10.11766/trxb201511240538 |
| [3] |
关松荫. 土壤酶及其研究法[M]. 北京: 中国农业出版社, 1986. GUAN Song-yin. Soil enzyme and research methods[M]. Beijing: China Agriculture Press, 1986. |
| [4] |
孟立君, 吴凤芝. 土壤酶研究进展[J]. 东北农业大学学报, 2004, 35(5): 622-626. MENG Li-jun, WU Feng-zhi. Advances on soil enzymes[J]. Journal of Northeast Agricultural University, 2004, 35(5): 622-626. |
| [5] |
Busto M D, Perez-Mateos M. Characterization of β-D-glucosidase extracted from soil fractions[J]. European Journal of Soil Science, 2000, 51(2): 193-200. DOI:10.1046/j.1365-2389.2000.00309.x |
| [6] |
Noinville S, Revault M, Baron M H, et al. Conformational changes and orientation of Humicola lanuginosa lipase on a solid hydrophobic surface:An in situ interface fourier transform infrared-attenuated total reflection study[J]. Biophysical Journal, 2002, 82(5): 2709-2719. DOI:10.1016/S0006-3495(02)75612-9 |
| [7] |
Gomes D E B, Lins R D, Pascutti P G, et al. The role of nonbonded interactions in the conformational dynamics of organophosphorous hydrolase adsorbed onto functionalized mesoporous silica surfaces[J]. The Journal of Physical Chemistry B, 2009, 114(1): 531-540. |
| [8] |
Jacobson K H, Kuech T R, Pedersen J A. Attachment of pathogenic prion protein to model oxide surfaces[J]. Environmental Science & Technology, 2013, 47(13): 6925-6934. |
| [9] |
Polano M, Anselmi C, Leita L, et al. Organic polyanions act as complexants of prion protein in soil[J]. Biochemical and Biophysical Research Communications, 2008, 367(2): 323-329. DOI:10.1016/j.bbrc.2007.12.143 |
| [10] |
Smith C B, Booth C J, Wadzinski T J, et al. Humic substances interfere with detection of pathogenic prion protein[J]. Soil Biology and Biochemistry, 2014, 68(1): 309-316. |
| [11] |
Jacobson K H, Lee S, Somerville R A, et al. Transport of the pathogenic prion protein through soils[J]. Journal of Environmental Quality, 2010, 39(4): 1145-1152. DOI:10.2134/jeq2009.0137 |
| [12] |
Booth C J, Johnson C J, Pedersen J A. Microbial and enzymatic inactivation of prions in soil environments[J]. Soil Biology and Biochemistry, 2013, 59(2): 1-15. |
| [13] |
Chesney A R, Booth C J, Lietz C B, et al. Peroxymonosulfate rapidly inactivates the disease-associated prion protein[J]. Environmental Science & Technology, 2016, 50(13): 7095-7105. |
| [14] |
Sander M, Madliger M, Schwarzenbach R P. Adsorption of transgenic insecticidal Cry1Ab protein to SiO2.1. Forces driving adsorption[J]. Environmental Science & Technology, 2010, 44(23): 8870-8876. |
| [15] |
Madliger M, Sander M, Schwarzenbach R P. Adsorption of transgenic insecticidal Cry1Ab protein to SiO2.2. Patch-controlled electrostatic attraction[J]. Environmental Science & Technology, 2010, 44(23): 8877-8883. |
| [16] |
Sander M, Tomaszewski J E, Madliger M, et al. Adsorption of insecticidal Cry1Ab protein to humic substances. 1. Experimental approach and mechanistic aspects[J]. Environmental Science & Technology, 2012, 46(18): 9923-9931. |
| [17] |
Violante A, De Cristofaro A, Rao M A, et al. Physicochemical properties of protein-smectite and protein-Al(OH)x-smectite complexes[J]. Clay Minerals, 1995, 30(4): 325-336. DOI:10.1180/claymin |
| [18] |
Tan W F, Koopal L K, Norde W. Interaction between humic acid and lysozyme, studied by dynamic light scattering and isothermal titration calorimetry[J]. Environmental Science & Technology, 2009, 43(3): 591-596. |
| [19] |
Tan W F, Norde W, Koopal L K. Interaction between lysozyme and humic acid in layer-by-layer assemblies:Effects of pH and ionic strength[J]. Journal of Colloid and Interface Science, 2014, 430: 40-46. DOI:10.1016/j.jcis.2014.05.029 |
| [20] |
Li Y, Tan W F, Wang M X, et al. Influence of lysozyme complexation with purified Aldrich humic acid on lysozyme activity[J]. European Journal of Soil Science, 2012, 63(5): 550-557. DOI:10.1111/ejs.2012.63.issue-5 |
| [21] |
Li Y, Tan W F, Koopal L K, et al. The influence of soil humic acids on the activities and stabilities of lysozyme and urease[J]. Environment Science & Technology, 2013, 47(10): 5050-5056. |
| [22] |
Tomaszewski J E, Schwarzenbach R P, Sander M. Protein encapsulation by humic substances[J]. Environmental Science & Technology, 2011, 45(14): 6003-6010. |
| [23] |
Tomaszewski J E, Madliger M, Pedersen J A, et al. Adsorption of insecticidal Cry1Ab protein to humic substances. 2. Influence of humic and fulvic acid charge and polarity characteristics[J]. Environmental Science & Technology, 2012, 46(18): 9932-9940. |
| [24] |
Jansson E, Tengvall P. Adsorption of albumin and IgG to porous and smooth titanium[J]. Colloids and Surfaces B:Biointerfaces, 2004, 35(1): 45-51. DOI:10.1016/j.colsurfb.2004.02.007 |
| [25] |
Huang Q Y, Liang W, Cai P. Adsorption, desorption and activities of acid phosphatase on various colloidal particles from an Ultisol[J]. Colloids and Surfaces B:Biointerfaces, 2005, 45(3/4): 209-214. |
| [26] |
Claus H, Filip Z. Effects of clays and other solids on the activity of phenoloxidases produced by some fungi and actinomycetes[J]. Soil Biology and Biochemistry, 1990, 22(4): 483-488. DOI:10.1016/0038-0717(90)90182-Y |
| [27] |
Quiquampoix H, Staunton S, Baron M H, et al. Interpretation of the pH dependence of protein adsorption on clay mineral surfaces and its relevance to the understanding of extracellular enzyme activity in soil[J]. Colloids and Surfaces A:Physicochemical and Engineering Aspects, 1993, 75(93): 85-93. |
| [28] |
Baron M H, Revault M, Servagent-Noinville S, et al. Chymotrypsin adsorption on montmorillonite:Enzymatic activity and kinetic FTIR structural analysis[J]. Journal of Colloid and Interface Science, 1999, 214(2): 319-332. DOI:10.1006/jcis.1999.6189 |
| [29] |
Karajanagi S S, Vertegel A A, Kane R S, et al. Structure and function of enzymes adsorbed onto single-walled carbon nanotubes[J]. Langmuir, 2004, 20(26): 11594-11599. DOI:10.1021/la047994h |
| [30] |
Maste M C L, Norde W, Visser A J W G. Adsorption-induced conformational changes in the serine proteinase savinase:A tryptophan fluorescence and circular dichroism study[J]. Journal of Colloid and Interface Science, 1997, 196(2): 224-230. DOI:10.1006/jcis.1997.5205 |
| [31] |
Bjerneld E J, Földes-Papp Z, Käll M, et al. Single-molecule surface-enhanced Raman and fluorescence correlation spectroscopy of horsera-dish peroxidase[J]. The Journal of Physical Chemistry B, 2002, 106(6): 1213-1218. DOI:10.1021/jp012268y |
| [32] |
Arnebrant T, Nylander T. Sequential and competitive adsorption of β-lactoglobulin and k-casein on metal surfaces[J]. Journal of Colloid and Interface Science, 1986, 111(2): 529-533. DOI:10.1016/0021-9797(86)90055-X |
| [33] |
Itoh H, Nagata A, Toyomasu T, et al. Adsorption of β-lactoglobulin onto the surface of stainless steel particles[J]. Bioscience Biotechnology and Biochemistry, 1995, 59(9): 1648-1651. DOI:10.1271/bbb.59.1648 |
| [34] |
Elofsson U M, Paulsson M A, Sellers P, et al. Adsorption during heat treatment related to the therma unfolding/aggregation of β-lactoglobulins A and B[J]. Journal of Colloid and Interface Science, 1996, 183(2): 408-415. DOI:10.1006/jcis.1996.0563 |
| [35] |
Lassen B, Malmsten M. Structure of protein layers during competitive adsorption[J]. Journal of Colloid and Interface Science, 1996, 180(2): 339-349. DOI:10.1006/jcis.1996.0312 |
| [36] |
Long J R, Shaw W J, Stayton P S, et al. Structure and dynamics of hydrated statherin on hydroxyapatite as determined by solid-state NMR[J]. Biochemistry, 2001, 40(51): 15451-15455. DOI:10.1021/bi010864c |
| [37] |
Lundqvist M, Sethson I, Jonsson B H. Protein adsorption onto silica nanoparticles:Conformational changes depend on the particles' curvature and the protein stability[J]. Langmuir, 2004, 20(24): 10639-10647. DOI:10.1021/la0484725 |
| [38] |
Vertegel A A, Siegel R W, Dordick J S. Silica nanoparticle size influences the structure and enzymatic activity of adsorbed lysozyme[J]. Langmuir, 2004, 20(16): 6800-6807. DOI:10.1021/la0497200 |
| [39] |
Marsh R J, Jones R A L, Sferrazza M. Neutron reflectivity study of the adsorption of β-lactoglobulinat a hydrophilic solid/liquid interface[J]. Journal of Colloid and Interface Science, 1999, 218(1): 347-349. DOI:10.1006/jcis.1999.6410 |
| [40] |
Follows D, Holt C, Nylander T, et al. Beta-casein adsorption at the silicon oxide-aqueous solution interface:Calcium ion effects[J]. Biomacromolecules, 2004, 5(2): 319-325. DOI:10.1021/bm034301n |
| [41] |
Murray B S, Deshaires C. Monitoring protein fouling of metal surfaces via a quartz crystal microbalance[J]. Journal of Colloid and Interface Science, 2000, 227(1): 32-41. DOI:10.1006/jcis.2000.6882 |
| [42] |
Tomaszewski J E, Schwarzenbach R P, Sander M. Protein encapsulation by humic substances[J]. Environmental Science & Technology, 2011, 45(14): 6003-6010. |
| [43] |
Huang H M, Lin F Y, Chen W Y, et al. Isothermal titration microcalorimetric studies of the effect of temperature on hydrophobic interaction between proteins and hydrophobic adsorbents[J]. Journal of Colloid and Interface Science, 2000, 229(2): 600-606. DOI:10.1006/jcis.2000.7017 |
| [44] |
Tsai Y S, Lin F Y, Chen W Y, et al. Isothermal titration microcalorimetric studies of the effect of salt concentrations in the interaction between proteins and hydrophobic adsorbents[J]. Colloids and Surfaces A:Physicochemical and Engineering Aspects, 2002, 197(1): 111-118. |
| [45] |
Larsericsdotter H, Oscarsson S, Buijs J. Thermodynamic analysis of proteins adsorbed on silica particles:Electrostatic effects[J]. Journal of Colloid and Interface Science, 2001, 237(1): 98-103. DOI:10.1006/jcis.2001.7485 |
| [46] |
Brandes N, Welzel P B, Werner C, et al. Adsorption-induced conformational changes of proteins onto ceramic particles:Differential scanning calorimetry and FTIR analysis[J]. Journal of Colloid and Interface Science, 2006, 299(1): 56-69. DOI:10.1016/j.jcis.2006.01.065 |
| [47] |
Takahara A, Hara Y, Kojio K, et al. Plasma protein adsorption behavior onto the surface of phase-separated organosilane monolayers on the basis of scanning force microscopy[J]. Colloids and Surfaces B:Biointerfaces, 2002, 23(2): 141-152. |
| [48] |
Ying P Q, Yu Y, Jin G, et al. Competitive protein adsorption studied with atomic force microscopy and imaging ellipsometry[J]. Colloids and Surfaces B:Biointerfaces, 2003, 32(1): 1-10. DOI:10.1016/S0927-7765(02)00133-9 |
| [49] |
Stevens P W, Hansberry M R, Kelso D M. Assessment of adsorption and adhesion of proteins to polystyrene microwells by sequential enzyme-linked-immunosorbent assay analysis[J]. Analytical Biochemistry, 1995, 225(2): 197-205. DOI:10.1006/abio.1995.1144 |
| [50] |
Roach P, Farrar D, Perry C C. Interpretation of protein adsorption:Surface-induced conformational changes[J]. Journal of the American Chemical Society, 2005, 127(22): 8168-8173. DOI:10.1021/ja042898o |
| [51] |
McClellan S J, Franses E I. Adsorption of bovine serum albumin at solid/aqueous interfaces[J]. Colloids and Surfaces A:Physicochemical and Engineering Aspects, 2005, 260(1): 265-275. |
| [52] |
Sigal G B, Mrksich M, Whitesides G M. Effect of surface wettability on the adsorption of proteins and detergents[J]. Journal of the American Chemical Society, 1998, 120(14): 3464-3473. DOI:10.1021/ja970819l |
| [53] |
Sivaraman B, Fears K P, Latour R A. Investigation of the effects of surface chemistry and solution concentration on the conformation of adsorbed proteins using an improved circular dichroism method[J]. Langmuir, 2009, 25(5): 3050-3056. DOI:10.1021/la8036814 |
| [54] |
Norde W, Anusiem A C I. Adsorption, desorption and re-adsorption of proteins on solid surfaces[J]. Colloids and Surfaces, 1992, 66(1): 73-80. DOI:10.1016/0166-6622(92)80122-I |
| [55] |
Sakiyama T, Tanino K, Urakawa M, et al. Adsorption characteristics of tryptic fragments of bovine β-lactoglobulin on a stainless steel surface[J]. Journal of Bioscience and Bioengineering, 1999, 88(5): 536-541. DOI:10.1016/S1389-1723(00)87672-0 |
| [56] |
Mori O, Imae T. AFM investigation of the adsorption process of bovine serum albumin on mica[J]. Colloids and Surfaces B:Biointerfaces, 1997, 9(1/2): 31-36. |
| [57] |
Su T J, Lu J R, Thomas R K, et al. The conformational structure of bovine serum albumin layers adsorbed at the silica-water interface[J]. The Journal of Physical Chemistry B, 1998, 102(41): 8100-8108. DOI:10.1021/jp981239t |
| [58] |
Su T J, Lu J R, Thomas R K, et al. Effect of pH on the adsorption of bovine serum albumin at the silica/water interface studied by neutron reflection[J]. The Journal of Physical Chemistry B, 1999, 103(18): 3727-3736. DOI:10.1021/jp983580j |
| [59] |
De Kruif C G, Weinbreck F, De Vries R. Complex coacervation of proteins and anionic polysaccharides[J]. Current Opinion in Colloid & Interface Science, 2004, 9(5): 340-349. |
| [60] |
Wang X, Ruengruglikit C, Wang Y W, et al. Interfacial interactions of pectin with bovine serum albumin studied by quartz crystal microbalance with dissipation monitoring:Effect of ionic strength[J]. Journal of Agricultural and Food Chemistry, 2007, 55(25): 10425-10431. DOI:10.1021/jf071714x |
| [61] |
Gianfreda L, Bollag J M. Effect of soils on the behavior of immobilized enzymes[J]. Soil Science Society of America Journal, 1994, 58(6): 1672-1681. DOI:10.2136/sssaj1994.03615995005800060014x |
| [62] |
Ross D J, McNeilly B A. Some influences of different soils and clay minerals on the activity of glucose oxidase[J]. Soil Biology and Biochemistry, 1972, 4(1): 9-18. DOI:10.1016/0038-0717(72)90037-5 |
| [63] |
Garwood G A, Mortland M M, Pinnavaia T J. Immobilization of glucose oxidase on montmorillonite clay:Hydrophobic and ionic modes of binding[J]. Journal of Molecular Catalysis, 1983, 22(2): 153-163. DOI:10.1016/0304-5102(83)83021-1 |
| [64] |
Boyd S A, Mortland M M. Selective effects of smectite-organic complexes on the activities of immobilized enzymes[J]. Journal of Molecular Catalysis, 1986, 34(1): 1-8. DOI:10.1016/0304-5102(86)87032-8 |
| [65] |
邬建敏. 蛋白质与酶在固相表面的吸附行为对其生物活性及稳定性影响的研究[D]. 杭州: 浙江大学, 2006. WU Jian-min. The adsorption of protein/enzyme on solid surface and the effect of adsorption on protein/enzyme bioactivity and stability[D]. Hangzhou:Zhejiang University, 2006. |
| [66] |
Makboul H E, Ottow J C G. Michaelis constant(Km) of acid phosphatase as aected by montmorillonite, illite and kaolinite clay minerals[J]. Microbial Ecology, 1979, 5: 207-213. DOI:10.1007/BF02013527 |
| [67] |
Gianfreda L, Rao M A, Violante A. Adsorption, activity and kinetic properties of urease on montmorillonite, aluminium hydroxide and Al(OH)x-montmorillonite complexes[J]. Soil Biology and Biochemistry, 1992, 24(1): 51-58. DOI:10.1016/0038-0717(92)90241-O |
| [68] |
Marzadori C, Francioso O, Ciavatta C, et al. Influence of the content of heavy metals and molecular weight of humic acids fractions on the activity and stability of urease[J]. Soil Biology and Biochemistry, 2000, 32: 1893-1898. DOI:10.1016/S0038-0717(00)00163-2 |
| [69] |
Kelleher B P, Willeford K O, Simpson A J, et al. Acid phosphatase interactions with organo-mineral complexes:Influence on catalytic activity[J]. Biogeochemistry, 2004, 71(3): 285-297. DOI:10.1023/B:BIOG.0000049348.53070.6f |
| [70] |
Marzadori C, Francioso O, Ciavatta C, et al. Activity and stability of jack bean urease in the presence of peat humic acids obtained using different extractants[J]. Biology & Fertility of Soils, 2000, 32: 415-420. |
| [71] |
Quiquampoix H, Burns R G. Interactions between proteins and soil mineral surfaces:Environmental and health consequences[J]. Elements, 2007, 3(6): 401-406. DOI:10.2113/GSELEMENTS.3.6.401 |
| [72] |
Rao M A, Violante A, Gianfreda L. Interaction of acid phosphatase with clays, organic molecules and organo-mineral complexes:Kinetics and stability[J]. Soil Biology and Biochemistry, 2000, 32(7): 1007-1014. DOI:10.1016/S0038-0717(00)00010-9 |
| [73] |
Tietjen T, Wetzel R G. Extracellular enzyme-clay mineral complexes:Enzyme adsorption, alteration of enzyme activity, and protection from photodegradation[J]. Aquatic Ecology, 2003, 37(4): 331-339. DOI:10.1023/B:AECO.0000007044.52801.6b |
| [74] |
Dong L H, Yang J S, Yuan H L, et al. Chemical characteristics and influences of two fractions of Chinese lignite humic acids on urease[J]. European Journal of Soil Biology, 2008, 44(2): 166-171. DOI:10.1016/j.ejsobi.2007.07.002 |
| [75] |
乔学琴. 土壤活性颗粒表面酸性磷酸酶的固定机理与特性[D]. 武汉: 华中农业大学, 2007. QIAO Xue-qin. Bingding mechanisms and properties of acid phosphatase on soil active particles[D]. Wuhan:Huazhong Agriculture University, 2007. |
| [76] |
Rao M A, Gianfreda L. Properties of acid phosphatase-tannic acid complexes formed in the presence of Fe and Mn[J]. Soil Biology and Biochemistry, 2000, 32(13): 1921-1926. DOI:10.1016/S0038-0717(00)00167-X |
| [77] |
Ahn M Y, Zimmerman A R, Martínez C E, et al. Characteristics of Trametes villosa laccase adsorbed on aluminum hydroxide[J]. Enzyme and Microbial Technology, 2007, 41(1/2): 141-148. |
| [78] |
Henzler K, Wittemann A, Breininger E, et al. Adsorption of bovine hemoglobin onto spherical polyelectrolyte brushes monitored by small-angle X-ray scattering and fourier transform infrared spectroscopy[J]. Biomacromolecules, 2007, 8(11): 3674-3681. DOI:10.1021/bm700953e |
| [79] |
Tavolaro P, Tavolaro A, Martino G. Influence of zeolite PZC and pH on the immobilization of cytochrome c:A preliminary study regarding the preparation of new biomaterials[J]. Colloids and Surfaces B:Biointerfaces, 2009, 70(1): 98-107. DOI:10.1016/j.colsurfb.2008.12.019 |
| [80] |
叶鹏, 万容兵, 王新平. 载体材料与蛋白质的相互作用及对其构象的影响[J]. 高分子通报, 2010, 11: 48-54. YE Peng, WAN Rong-bing, WANG Xin-ping. Progress of study on interaction between support and protein and its effect on protein conformation[J]. Chinese Polymer Bulletin, 2010, 11: 48-54. |
| [81] |
Haynes C A, Norde W. Structures and stabilities of adsorbed proteins[J]. Journal of Colloid and Interface Science, 1995, 169(2): 313-328. DOI:10.1006/jcis.1995.1039 |
| [82] |
Quiquampoix H, Chassin P, Ratcliffe R G. Enzyme activity and cation exchange as tools for the study of the conformation of proteins adsorbed on mineral surfaces[J]. Progress in Colloid and Polymer Science, 1989, 79: 59-63. DOI:10.1007/BFb0116176 |
| [83] |
Simpson G H, Hughes J D. Arylsulphatase clay interactions. I. Adsorption of arylsulphatase by kaolinite and montmorillonite[J]. Soil Research, 1978, 16(1): 27-33. DOI:10.1071/SR9780027 |
| [84] |
Leprince F, Quiquampoix H. Extracellular enzyme activity in soil:Effect of pH and ionic strength on the interaction with montmorillonite of two acid phosphatases secreted by the ectomycorrhizal fungus Hebeloma cylindrosporum[J]. European Journal of Soil Science, 1996, 47(4): 511-522. DOI:10.1111/ejs.1996.47.issue-4 |
| [85] |
Billsten P, Carlsson U, Elwing H. Studies on the conformation of adsorbed proteins with the use of nanoparticle technology[J]. Surfactant Science Series, 1998, 75: 627-650. |
| [86] |
Kondo A, Oku S, Higashitani K. Structural changes in protein molecules adsorbed on ultrafine silica particles[J]. Journal of Colloid and Interface Science, 1991, 143(1): 214-221. DOI:10.1016/0021-9797(91)90454-G |
| [87] |
Kondo A, Fukuda H. Effects of adsorption conditions on kinetics of protein adsorption and conformational changes at ultrafine silica particles[J]. Journal of Colloid and Interface Science, 1998, 198(1): 34-41. DOI:10.1006/jcis.1997.5278 |
| [88] |
Giacomelli C E, Norde W. The adsorption-desorption cycle. Reversibility of the BSA-silica system[J]. Journal of Colloid and Interface Science, 2001, 233(2): 234-240. DOI:10.1006/jcis.2000.7219 |
| [89] |
Zoungrana T, Findenegg G H, Norde W. Structure, stability, and activity of adsorbed enzymes[J]. Journal of Colloid and Interface Science, 1997, 190(2): 437-448. DOI:10.1006/jcis.1997.4895 |
| [90] |
Weetall H H. Immobilized enzymes. Analytical applications[J]. Analytical Chemistry, 1974, 46(7): 602A-604A. |
| [91] |
鲍腾, 彭书传, 陈冬, 等. 凹凸棒石粘土固定辣根过氧化物酶处理含酚废水[J]. 环境工程学报, 2012, 6(9): 3179-3185. BAO Teng, PENG Shu-chuan, CHEN Dong, et al. Horseradish peroxidase immobilization in palygorskite-based material for phenol wastewater treatment[J]. Chinese Journal of Environmental Engineering, 2012, 6(9): 3179-3185. |
| [92] |
王翠, 姜艳军, 周丽亚, 等. 纳米氧化硅固定辣根过氧化物酶处理苯酚废水[J]. 化工学报, 2011, 62(7): 2026-2032. WANG Cui, JIANG Yan-jun, ZHOU Li-ya, et al. Horseradish peroxidase encapsulated on nanosilica for phenol removal[J]. CIESC Journal, 2011, 62(7): 2026-2032. |
| [93] |
代云容, 丁士元, 牛军峰. 电纺纳米纤维膜固定化漆酶去除土壤中多环芳烃的研究[C]//持久性有机污染物论坛2011暨第六届持久性有机污染物全国学术研讨会论文集·哈尔滨, 2011. DAI Yun-rong, DING Shi-yuan, NIU Jun-feng. Removal of polycyclic aromatic hydrocarbons from soil by immobilized laccase on electrospun nanofiber membranes[C]//Proceedings of the 2011 symposium on persistent organic pollutants forum and the sixth national symposium on persistent organic pollutants·Harbin, 2011. |
| [94] |
Ma T T, Zhu L S, Wang J H, et al. Enhancement of atrazine degradation by crude and immobilized enzymes in two agricultural soils[J]. Environmental Earth Sciences, 2011, 64(3): 861-867. DOI:10.1007/s12665-011-0910-6 |
| [95] |
张颖, 王溪, 姜昭, 等. 固定化酶修复阿特拉津污染土壤效果及土壤细菌多样性动态变化分析[J]. 东北农业大学学报, 2013, 44(11): 1-6. ZHANG Ying, WANG Xi, JIANG Zhao, et al. Effect of atrazine-contaminated soil by immobilization enzyme and dynamic variation of soil microbial[J]. Journal of Northeast Agricultural University, 2013, 44(11): 1-6. DOI:10.3969/j.issn.1005-9369.2013.11.001 |
| [96] |
王军, 王秀国, 朱鲁生, 等. 海藻酸钠固定化莠去津降解酶的研究[J]. 农业环境科学学报, 2007, 26(5): 1733-1737. WANG Jun, WANG Xiu-guo, ZHU Lu-sheng, et al. Atrazine degrading enzyme's immobilization in sodium alginate[J]. Journal of Agro-Environment Science, 2007, 26(5): 1733-1737. |
| [97] |
葛世杰. 固定化阿特拉津降解酶制备及其土壤修复效果研究[D]. 哈尔滨: 东北农业大学, 2013. GE Shi-jie. Study on immobilized atrazine degradation enzyme properties and soil remediation effect[D]. Harbin:Northeast Agricultural University, 2013. |
| [98] |
何文英, 陈光英, 杜娟, 等. 光谱法与分子模拟研究胡椒碱对牛血清白蛋白的键合作用[J]. 化学学报, 2008, 66(21): 2365-2370. HE Wen-ying, CHEN Guang-ying, DU Juan, et al. Investigation on the binding of piperine to bovine serum albumin by optical spectroscopy and molecular modeling[J]. Acta Chimica Sinica, 2008, 66(21): 2365-2370. DOI:10.3321/j.issn:0567-7351.2008.21.009 |
| [99] |
唐志华. 分子模拟技术与光谱法结合研究硫酸沙丁胺醇与两种蛋白质的相互作用[J]. 中国实验方剂学杂志, 2014, 20(24): 114-117. TANG Zhi-hua. Studies of interaction between salbutamol sulfate and two proteins by molecular modeling and spectroscopic methods[J]. Chinese Journal of Experimental Traditional Medical Formulae, 2014, 20(24): 114-117. |
| [100] |
张晟瑞, 王芹, 季晓辉. 分子模拟技术与光谱法结合研究抑菌剂磺胺二甲嘧啶与人血清白蛋白之间的相互作用[J]. 中国实验方剂学杂志, 2014, 20(15): 104-107. ZHANG Sheng-rui, WANG Qin, JI Xiao-hui. Molecular modeling and multi-spectroscopic approaches to study interaction between antimicrobial drug sulfamethazine and human serum albumin[J]. Chinese Journal of Experimental Traditional Medical Formulae, 2014, 20(15): 104-107. |
| [101] |
Chapron Y, Charlet L, Sahai N. Fate of pathological prion(PrPsc 92-138) in soil and water:Prion-clay nanoparticle molecular dynamics[J]. Journal of Biomolecular Structure and Dynamics, 2014, 32(11): 1802-1816. DOI:10.1080/07391102.2013.836461 |
| [102] |
Andersen A, Reardon P N, Chacon S S, et al. Protein-mineral interactions:Molecular dynamics simulations capture importance of variations in mineral surface composition and structure[J]. Langmuir, 2016, 32(24): 6194-6209. DOI:10.1021/acs.langmuir.6b01198 |
 2018, Vol. 37
2018, Vol. 37