2. 复旦大学化学系, 上海 200433
2. Department of Chemistry, Fudan University, Shanghai 200433, China
近年来,人工湿地由于其造价低、便于维修管理、处理效率高、观赏性强等优点,被广泛应用于污水处理中[1-2]。然而,湿地在净化水质的同时会释放大量温室气体,越来越受到广泛关注[3]。微生物和植物是人工湿地的重要组成部分,其污水处理能力取决于湿地设计、微生物群落和植物种类等[4]。微生物在湿地生物地球化学循环中起着重要作用;不同的微生物群落会产生不同的生物化学反应,导致营养盐的消耗[5-6]。因此,湿地植物根系微生物群落结构的研究有助于改进人工湿地的设计以及管理,提高湿地水质净化能力,并对湿地温室气体排放的控制提供理论依据[7]。湿地中微生物的硝化、反硝化作用是去除氮的主要途径,硝化、反硝化细菌是反应的主要执行者[8]。因此,对湿地中硝化细菌、反硝化细菌展开分析有助于湿地污水脱氮机制的研究。
过去,人们针对湿地微生物种群的研究一直建立在传统的分离和培养方法上,这种方法费时费力,培养的种群数量也有限[9]。目前,关于湿地土壤微生物的研究大多运用传统分子生物学技术,如PCR-DGGE和FISH[10]。PCR-DGGE技术是将PCR扩增和变性梯度凝胶电泳结合起来,可对同一长度的PCR扩增片断按序列的不同在变性梯度凝胶上分离,在分析微生物群落多样性方面具有明显的优势,但是它只适用于微生物比较丰富的样本[10]。FISH技术是根据已知微生物不同分类级别上种群特异的DNA序列,以荧光标记的特异寡聚核苷酸片段作为探针,与环境基因组中DNA分子杂交,该技术可以进行样品原位杂交,但受引物以及试样不合适的限制[10]。高通量DNA测序是一种测序深度较深、覆盖度较广的方法,该技术通过大量平行序列产生大量DNA数据,其中的可操作性分类单元(OTUs)可用于有效分析样本中微生物的多样性[10]。
本研究选择美人蕉、菖蒲和水葱三种典型的湿地植物,通过构建复合垂直流人工湿地研究不同季节、上行池和下行池土壤根际微生物群落结构的变化特征,分析根系微生物结构和功能的对应关系,旨在为人工湿地的改进和优化提供参考。
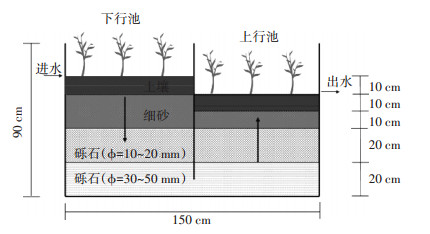
1 材料与方法 1.1 试验系统复合垂直流人工湿地系统位于复旦大学邯郸校区研究基地,在自然光照下的露天大棚内运行,示意图如图 1所示。每个系统由上、下行池组成,尺寸为150 cm×90 cm×90 cm(长×宽×高),下行池内基质自下而上分别为20 cm砾石(ϕ30~50 mm)、20 cm砾石(ϕ10~20 mm)、20 cm细砂和10 cm土壤;上行池自下而上分别为20 cm砾石(ϕ30~50 mm)、20 cm砾石(ϕ10~20 mm)、10 cm细砂和10 cm土壤。每个系统种植一种植物,分别为菖蒲、美人蕉和水葱,每平方米分别种植20棵、20棵和20簇。模拟生活污水从湿地下行池上端流入,从上行池上端流出。模拟生活污水主要是往自来水中加入葡萄糖、淀粉、鱼粉蛋白胨、硝酸钾、磷酸氢二钾、硫酸镁等。进水水质总有机碳、铵态氮、总氮和总磷的浓度分别为68~84、20~28、50~59 mg·L-1和4~6 mg·L-1。进水采用间歇流的方式,一周配一次1000 L污水,污水放置在一个1000 L的塑料桶中,水力负荷为330 mm·d-1,停留时间为5 d。系统从2015年6月开始运行,到2017年5月份结束,运行稳定,植物长势良好。
|
图 1 复合垂直流人工湿地系统示意图 Figure 1 Picture of integrated vertical flow constructed wetland |
本试验于2016年7月、10月和2017年1月用五点采样法分别采集三种植物湿地系统的上行池、下行池10 cm处根系土壤。样品标记为:ACL表示菖蒲,CIL表示美人蕉,SVV表示水葱;7、10、1表示月份;D表示下行池,U表示上行池。样品名称分别为:ACL7D、ACL7U、CIL7D、CIL7U、SVV7D、SVV7U、ACL10D、ACL10U、CIL10D、CIL10U、SVV10D、SVV10U、ACL1D、ACL1U、CIL1D、CIL1U、SVV1D、SVV1U。
1.3 DNA提取采用Omega E.Z.N.A.® Soil DNA Kit提取试剂盒提取根系土壤DNA,按说明书完成相关操作,然后将提取好的DNA保存在-20 ℃。
1.4 PCR扩增与测序本实验采用引物520F(5′-barcode+ GCACCTAAYTGGGYDTAAAGNG-3′)和802R(5′-TACNVGGGTATCTAATCC-3′)[11],扩增长度约为280 bp的16S rRNA V4区用来测序。
PCR采用NEB Q5 DNA高保真聚合酶,各组成及用量如下:0.25 μLQ5 high-fidelity DNA polymerase,5 μL 5*Reaction Buffer,5 μL 5* High GC Buffer,0.5 μL dNTP(10 mmol·L-1),1 μL模板DNA,1 μL正向引物(10 μmol·L-1),1 μL反向引物(10 μmol·L-1)和11.25 μL水。PCR扩增程序如下:将PCR反应所需的成分配制完后,在PCR仪上于98 ℃预变性30 s,使模板DNA充分变性,然后进入扩增循环。在每一个循环中,先于98 ℃保持15 s使模板变性,然后将温度降到50 ℃,保持30 s,使引物与模板充分退火;在72 ℃保持30 s,使引物在模板上延伸,合成DNA,完成一个循环。重复这样的循环25~27次,使扩增的DNA片段大量累积。最后,在72 ℃保持5 min,使产物延伸完整,4 ℃保存。
利用Illumina公司的TruSeq Nano DNA LT Library Prep Kit进行建库;在MiSeq机器上进行MiSeq Reagent Kit V3(600 cycles)2×300 bp的双端测序。
1.5 数据分析对样品进行Alpha多样性分析时,在90%的最低测序深度水平,采取对OTU丰度矩阵中全体样本进行随机重复抽样,并使用Chao1指数来表示群落丰富度。样品进行Beta多样性分析时,对Unweighted UniFrac距离矩阵和Weighted UniFrac距离矩阵进行NMDS分析,用于描述群落样本的结构分布。
2 结果与讨论样品的稀疏曲线如图 2所示,所有样品测序量在5260时,曲线已趋于平缓或者达到平台期,因此可以认为测序深度已经基本覆盖到样品中所有的物种。
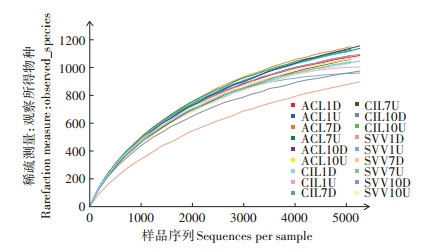
|
图 2 样品的稀疏曲线 Figure 2 The rarefaction curves for samples |
三种植物湿地微生物多样性指数如图 3所示。水葱湿地在实验期间,上行池与下行池的物种丰富度差异不大。除10月份美人蕉湿地下行池Chao1指数小于上行池Chao1指数外,菖蒲湿地与美人蕉湿地表现为其下行池的丰富度大于上行池。这是因为下行池为污水输入端,营养盐含量较高,适宜微生物代谢繁殖。流入上行池的污水经过下行池的降解处理,能被微生物利用的营养盐降低,导致微生物丰富度下降。

|
图 3 三种植物湿地微生物多样性指数 Figure 3 Diversity indices of microbes in three plant wetlands |
本研究用NMDS分析(Nonmetric Multidimensional Scaling),通过降维处理简化数据结构,在特定距离尺度下描述样本分布特征。图 4a为Unweighted UniFrac NMDS分析的样本二维排序图。除ACL7U样品,样本组均匀分布在坐标上下两侧,表明在试验期间上行池和下行池样本组群落成员相似,但是两者群落成员组成之间有差异,存在各自独有的微生物。这可能是因为下行池消耗了大量氧气,导致上行池氧气较低,使得下行池中有氧细菌远多于上行池中有氧细菌数[12]。
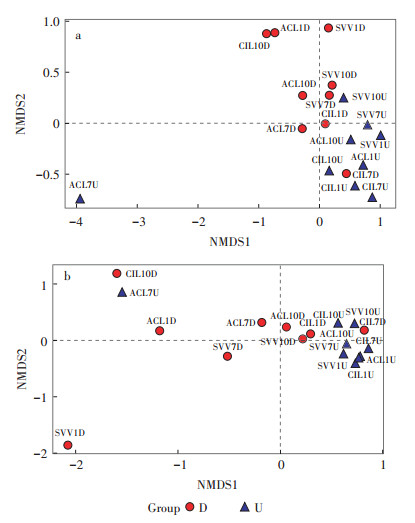
|
图 4 Unweighted UniFrac NMDS分析的样本二维排序图(a)和Weighted UniFrac NMDS分析的样本二维排序图(b) Figure 4 Unweighted UniFrac NMDS analysis of samples in two dimensions (a) and Weighted UniFrac NMDS analysis of samples in two dimensions (b) |
由图 4b可知,除ACL7U样品,上行池样品组靠的较近,表明从群落成员丰度上来看,上行池样本相似性较下行池样本相似性高,这主要是因为下行池是污水净化的主要场所,下行池的营养盐含量和溶解氧含量等相关影响因素沿程变化梯度较大,造成微生物生存环境变化较大,三种植物湿地在不同环境影响下形成了差异略大的生态系统。而经过下行池的水流,其中的污染物浓度已经较低,其浓度、溶解氧等变化较为平缓,导致微生物所处环境较为相似[13],因此上行池样品组相似性高于下行池。
门和属水平上微生物群落组成及丰度如图 5所示,在菖蒲湿地中(图 5左侧a),主要的微生物有变形菌门(Proteobacteria,38.4%)、酸杆菌门(Acidobacteria,16.9%)、绿弯菌门(Chloroflexi,13.1%)。变形菌门所占比例较大,这与已有研究一致[14-15]。酸杆菌门相对丰度在菖蒲湿地下行池夏秋冬季节、上行池夏秋冬季节占有较大比例,分别为15.6%、15.8%、17.0%、17.2%、16.5%、19.5%。绿弯菌门的相对丰度在菖蒲下行池夏秋冬季节、上行池夏秋冬季节分别占14.5%、11.7%、11.2%、11.0%、16.5%、13.8%。

|
图 5 门和属水平上微生物群落在样本中组成及丰度 Figure 5 Taxonomic composition at phylum and genus level and abundance of microbes in each sample |
美人蕉湿地的微生物群落结果如图 5左侧b所示,下行池根系的变形菌门相对丰度在秋季最大,达到50.9%;上行池根系变形菌门相对丰度在夏季、秋季和冬季差异不大,维持在33.0%到34.2%之间。总体来说,下行池变形菌门相对丰度高于上行池。酸杆菌门在下行池、上行池的相对丰度夏季最高,分别为20.4%、18.7%,且下行池酸杆菌门相对丰度高于上行池。绿弯菌门下行池夏季较高,达到14.4%,上行池秋季较高,达到16.6%。
水葱湿地根系微生物主要类别同为变形菌门(34.6%)、酸杆菌门(17.5%)、绿弯菌门(10.8%)(图 5左侧c)。随季节演替,变形菌门在下行池的相对丰度从46.3%降到31.1%。上行池相对丰度随季节变化不明显,基本维持在30.0%。下行池酸杆菌门的相对丰度夏季>秋季>冬季,分别为17.0%、16.1%、10.7%,上行池秋季>夏季>冬季,分别为21.7%、20.0%、19.6%。绿弯菌门下行池秋季>夏季>冬季,分别为13.0%、9.4%、6.8%。
三种湿地植物根系微生物组成在门水平上相似,主要有变形菌门、酸杆菌门、绿弯菌门等。变形菌门比例较高,包含许多微生物都与碳、氮、硫循环相关[16],在去除碳、氮方面有重要的作用,同时对温室气体的生成有巨大影响。酸杆菌门是严格厌氧细菌,用来发酵芳香族混合物和醋酸盐[17]。绿弯菌门在营养盐的去除过程中发挥着重要作用[18]。同时湿地中还检测到了浮霉菌门(Planctomycetes)、芽单胞菌门(Gemmatimonadetes)、放线菌门(Actinobacteria)、拟杆菌门(Bacteroidetes)、泉古菌门(Crenarchaeota)、厚壁菌门(Firmicutes)等。浮霉菌门能将NO2-作为电子受体,将氨转化为氮气,对NH3-N的去除有重要影响[19]。芽单胞菌门中的一些菌种已被确认为革兰氏阴性、好氧、积累磷酸盐微生物[20]。放线菌门将氢气作为电子供体用来去除硝酸盐,对生成N2O有重要影响。疣微菌门相对丰度为0.8%,能氧化CH4,对其排放有抑制作用[21]。
属水平上的微生物群落组成及相对丰度如图 5右侧图所示,三种湿地根系中主要的微生物属有Nitrospira、Candidatus Nitrosophaera等。其中,Nitrospira是硝化细菌;Candidatus Nitrosophaera是亚硝化细菌。不同植物湿地优势菌属有类似,也有差异。硝化细菌最主要的微生物属为Nitrospira,下行池在秋季达到最高,相对丰度为2.44%。
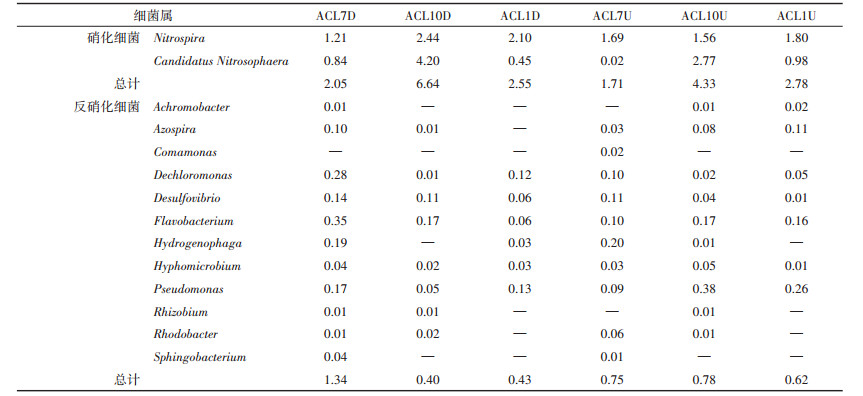
菖蒲湿地在属水平上的硝化、反硝化细菌组成及其相对丰度如表 1所示。Nitrospira和Candidatus Nitrosophaera是主要的硝化细菌,下行池、上行池相对丰度均在秋季达到最高,分别为6.64%、4.33%。反硝化微生物有Dechloromonas、Flavobacterium、Pseudomonas属等,这与已有研究结果一致[22-25]。菖蒲下行池的根系反硝化微生物在夏季所占比例最多,约1.34%,秋季与冬季差异不明显,上行池反硝化细菌相对丰度随季节变化不明显,基本维持在0.62%~0.78%。下行池反硝化微生物相对丰度高于上行池。下行池硝化细菌、反硝化细菌相对丰度都比上行池高,这可能是因为下行池是进水端,含有丰富的氮源、碳源供微生物生长活动[13]。
|
|
表 1 菖蒲湿地硝化细菌、反硝化细菌的相对丰度表(%) Table 1 The relative abundance of nitrifying bacteria and denitrifying bacteria in ACL cell(%) |
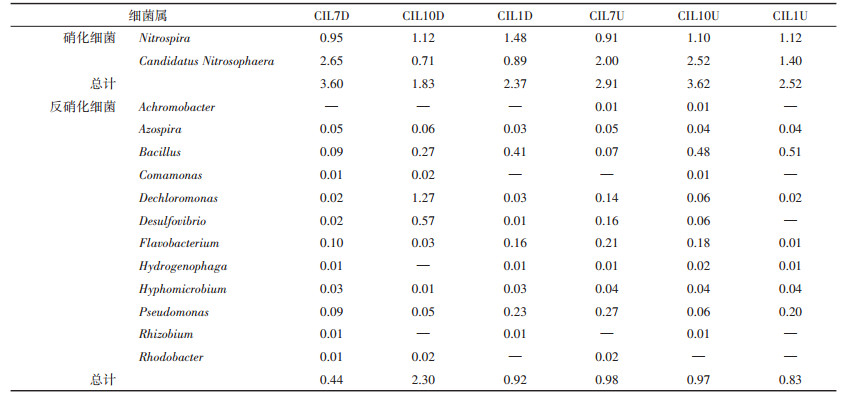
在美人蕉湿地中,下行池植物根系的硝化细菌Nitrospira和Candidatus Nitrosophaera相对丰度在夏季达到最高3.60%,上行池秋季达到最高3.62%(表 2)。反硝化细菌主要有Dechloromonas、Flavobacterium、Pseudomonas等。下行池和上行池根系反硝化细菌相对丰度分别在秋季和夏季达到最高,分别为2.30%和0.98%。
|
|
表 2 美人蕉湿地硝化细菌、反硝化细菌的相对丰度表(%) Table 2 The relative abundance of nitrifying bacteria and denitrifying bacteria in CIL cell(%) |
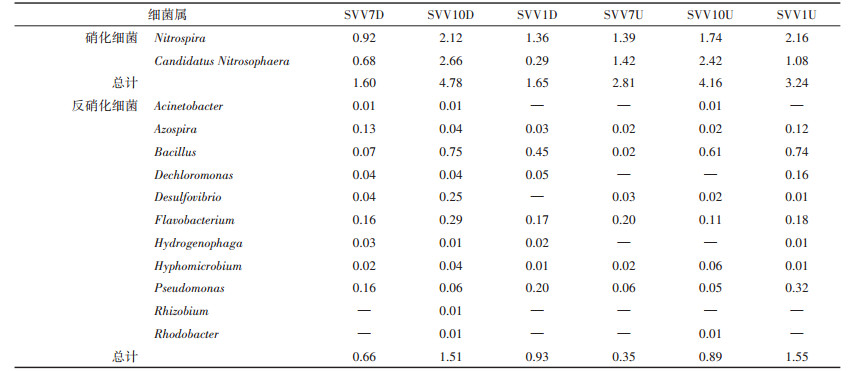
在水葱湿地中,硝化细菌为Nitrospira和Candidatus Nitrosophaera(表 3),上、下行池的相对丰度在秋季达到最高,分别占4.16%、4.78%。反硝化细菌有Dechloromonas、Flavobacterium、Pseudomonas等,下行池反硝化细菌相对丰度在秋季达到最高,为1.51%;上行池冬季达到最高,为1.55%。
|
|
表 3 水葱湿地硝化细菌、反硝化细菌的相对丰度表(%) Table 3 The relative abundance of nitrifying bacteria and denitrifying bacteria in SVV cell(%) |
总之,湿地植物根系硝化细菌属为Nitrospira、Candidatus Nitrosophaera,反硝化细菌属为Dechloromonas、Flavobacterium、Pseudomonas等。菖蒲和水葱湿地秋季硝化细菌丰度均高于夏季和冬季。
3 结论(1)菖蒲湿地、美人蕉湿地和水葱湿地系统Chao1指数均表现为秋季>夏季>冬季,即微生物丰富度秋季>夏季>冬季。水葱湿地上行池与下行池物种丰富度差异不大;除秋季的美人蕉湿地,菖蒲湿地与美人蕉湿地Chao1指数表现为下行池>上行池。
(2)在试验期间,各湿地上行池和下行池样本组群落组成均相似;但上、下行池群落成员组成之间有差异,上行池样本相似性较下行池样本高。
(3)门水平上,湿地根系微生物从大到小依次为变形菌门、酸杆菌门、绿弯菌门等。从属水平上,检测到了硝化细菌Nitrospira和Candidatus Nitrosophaera;反硝化细菌Dechloromonas、Flavobacterium和Pseudomonas等。菖蒲湿地和水葱湿地根系硝化细菌秋季的相对丰度均高于夏季和冬季。
| [1] |
Pelissari C, ávila C, Trein C M, et al. Nitrogen transforming bacteria within a full-scale partially saturated vertical subsurface flow constructed wetland treating urban wastewater[J]. Science of the Total Environment, 2016, 574: 390-399. |
| [2] |
Chen Y, Wen Y, Tang Z, et al. Effects of plant biomass on bacterial community structure in constructed wetlands used for tertiary wastewater treatment[J]. Ecological Engineering, 2015, 84: 38-45. DOI:10.1016/j.ecoleng.2015.07.013 |
| [3] |
Wu J, Zhang J, Jia W, et al. Impact of COD/N ratio on nitrous oxide emission from microcosm wetlands and their performance in removing nitrogen from wastewater[J]. Bioresource Technology, 2009, 100(12): 2910-2917. DOI:10.1016/j.biortech.2009.01.056 |
| [4] |
Ibekwe A M, Lyon S R, Leddy M, et al. Impact of plant density and microbial composition on water quality from a free water surface constructed wetland[J]. Journal of Applied Microbiology, 2007, 102(4): 921-936. |
| [5] |
Stottmeister U, Wießner A, Kuschk P, et al. Effects of plants and microorganisms in constructed wetlands for wastewater treatment[J]. Biotechnology Advances, 2003, 22(1/2): 93-117. |
| [6] |
Ligi T, Oopkaup K, Truu M, et al. Characterization of bacterial communities in soil and sediment of a created riverine wetland complex using high-throughput 16S rRNA amplicon sequencing[J]. Ecological Engineering, 2014, 72: 56-66. DOI:10.1016/j.ecoleng.2013.09.007 |
| [7] |
Truu M, Juhanson J, Truu J. Microbial biomass, activity and community composition in constructed wetlands[J]. Science of the Total Environment, 2009, 407(13): 3958-3971. DOI:10.1016/j.scitotenv.2008.11.036 |
| [8] |
刘超翔, 董春宏. 潜流式人工湿地污水处理系统硝化能力研究[J]. 环境科学, 2003, 24(1): 80-83. LIU Chao-xiang, DONG Chun-hong. Study on ability of nitrification in a subsurface constructed wetland system treating sewage[J]. Environmental Science, 2003, 24(1): 80-83. DOI:10.3321/j.issn:0250-3301.2003.01.013 |
| [9] |
雷旭, 李冰, 李晓, 等. 复合垂直流人工湿地系统中不同植物根际微生物群落结构[J]. 生态学杂志, 2015, 34(5): 1373-1381. LEI Xu, LI Bing, LI Xiao, et al. Rhizosphere microbial communities of three plants in vertical-flow constructed wetland[J]. Chinese Journal of Ecology, 2015, 34(5): 1373-1381. |
| [10] |
Margulies M, Egholm M, Altman W E, et al. Genome sequencing in microfabricated high-density picolitre reactors[J]. Nature, 2005, 437: 376-380. DOI:10.1038/nature03959 |
| [11] |
Shaw A G, Sim K, Powell E, et al. Latitude in sample handling and storage for infant faecal microbiota studies:The elephant in the room[J]. Microbiome, 2016, 4(1): 40. |
| [12] |
刘志伟, 周美修, 宋俊玲, 等. 复合垂直流人工湿地污染物去除特征及微生物群落多样性分析[J]. 环境工程, 2014, 32(6): 38-42. LIU Zhi -wei, ZHOU Mei-xiu, SONG Jun-ling, et al. Pollutants removal characteristics and analysis of microbial community diversity in integrated vertical-flow constructed wetland[J]. Environmental Engineering, 2014, 32(6): 38-42. |
| [13] |
黄德锋, 李田, 陆斌. 复合垂直流人工湿地污染物去除及微生物群落结构的PCR-DGGE分析[J]. 环境科学研究, 2007, 20(6): 137-141. HUANG De-feng, LI Tian, LU Bin. Pollutants removal and analysis of structure changes of microbial community in integrated vertical-flow constructed wetland[J]. Research of Environmental Sciences, 2007, 20(6): 137-141. |
| [14] |
Wang Q, Xie H, Ngo H H, et al. Microbial abundance and community in subsurface flow constructed wetland microcosms:Role of plant presence[J]. Environmental Science & Pollution Research International, 2016, 23(5): 1-10. |
| [15] |
Yuan J, Dong W, Sun F, et al. Bacterial communities and enzymatic activities in the vegetation-activated sludge process (V-ASP) and related advantages by comparison with conventional constructed wetland[J]. Bioresource Technology, 2016, 220: 341-351. DOI:10.1016/j.biortech.2016.08.095 |
| [16] |
Ansola G, Arroyo P, Le S D M. Characterisation of the soil bacterial community structure and composition of natural and constructed wetlands[J]. Science of the Total Environment, 2014, 473-474(3): 63-71. |
| [17] |
Hugenholtz P, Goebel B M, Pace N R. Impact of culture-independent studies on the emerging phylogenetic view of bacterial diversity[J]. Journal of Bacteriology, 1998, 180(18): 4765-4774. |
| [18] |
Ma Q, Qu Y Y, Zhang X W, et al. Identification of the microbial community composition and structure of coal-mine wastewater treatment plants[J]. Microbiological Research, 2015, 175: 1-5. DOI:10.1016/j.micres.2014.12.013 |
| [19] |
Pynaert K, Smets B F, Wyffels S, et al. Characterization of an autotrophic nitrogen-removing biofilm from a highly loaded lab-scale rotating biological contactor[J]. Applied & Environmental Microbiology, 2003, 69(6): 3626-3635. |
| [20] |
Vaz-Moreira I, Silva M E, Manaia C M, et al. Diversity of bacterial isolates from commercial and homemade vomposts[J]. Microbial Ecology, 2008, 55(4): 714-722. DOI:10.1007/s00248-007-9314-2 |
| [21] |
Liessens J, Vanbrabant J, Vos P D, et al. Mixed culture hydrogenotrophic nitrate reduction in drinking water[J]. Microbial Ecology, 1992, 24(3): 271-290. DOI:10.1007/BF00167786 |
| [22] |
Lai Q, Shao Z. Pseudomonas xiamenensis sp. nov., a denitrifying bacterium isolated from activated sludge[J]. International Journal of Systematic & Evolutionary Microbiology, 2008, 58(8): 1911-1915. |
| [23] |
Drysdale G D, Kasan H C, Bux F. Denitrification by heterotrophic bacteria during activated sludge treatment[J]. Water SA, 1999, 25(3): 357-362. |
| [24] |
Hayatsu M, Tago K, Saito M. Various players in the nitrogen cycle:Diversity and functions of the microorganisms involved in nitrification and denitrification[J]. Soil Science & Plant Nutrition, 2008, 54(1): 33-45. |
| [25] |
Pang S, Zhang S, Lv X Y, et al. Characterization of bacterial community in biofilm and sediments of wetlands dominated by aquatic macrophytes[J]. Ecological Engineering, 2016, 97: 242-250. DOI:10.1016/j.ecoleng.2016.10.011 |
 2019, Vol. 38
2019, Vol. 38