2. 中国科学院生态环境研究中心, 水污染控制实验室, 北京 100085;
3. 中国科学院大学, 北京 100049
2. Laboratory of Water Pollution Control Technology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China
我国是畜禽养殖大国,畜禽养殖污染居全国污染物排放首位[1]。畜禽养殖粪污富含有机质、氮、磷、重金属、有机污染物和病原微生物,但随着我国生态环境保护力度的不断加强和新时代的生态文明建设,畜禽粪污处理处置也发生了相应的变化。畜禽粪污污染控制早期以有机污染防控为主(COD当量),强调有机物去除、转化和腐熟,解决土地利用中的黑臭和烧苗等问题;随后发展成为以无机污染防控为主(重金属、氮、磷等),减少重金属在植物中的富集和农业面源污染等问题;目前,畜禽粪污中生物污染的防控仍未引起足够重视,亟需加强相关研究。2020年10月17日,为了维护国家安全,防范和应对生物安全风险,我国颁发了《中华人民共和国生物安全法》,并将生物安全定义为国家有效防范和应对危险生物因子及相关因素威胁,生物技术能够稳定健康发展,人民生命健康和生态系统相对处于没有危险和不受威胁的状态,生物领域具备维护国家安全和持续发展的能力[2]。危险生物因子(Dangerous biological agents,DBAs)具有潜伏周期长、隐匿性强、溯源分析难等特点,如当前对全球生物安全造成重大威胁的新型冠状病毒(SARS-CoV-2),其溯源分析是世界性科学难题[3-4]。1984年美国历史上发生的沙门氏菌感染恐袭事件,是在一年以后才被认定是人为故意传播导致的生物安全事件[5]。因此,识别潜在的DBAs,鉴定DBAs可能导致的生物安全事故,明确DBAs在环境中的归趋,进而提前开发有效管控DBAs的医疗措施等是预防生物安全事故、保障生物安全的重要研究领域。
随着时代的发展,生物安全领域的范畴逐渐从人体健康保障扩展到动植物、环境等领域[5]。尤其是近年来畜禽养殖行业的集约式、规模化发展,畜禽养殖领域的生物安全问题引起了人们的广泛关注。畜禽养殖行业是世界范围内动物疫情暴发的热点区域,如禽流感、非洲猪瘟(African swine fever,ASF)等动物疫情的暴发,不仅影响到了食品的安全供应,而且引发了人畜共患病的担忧,始终是生物安全领域不可忽视的重大问题之一[6-7]。我国生物安全法第三章中亦将防控重大新发突发传染病、动植物疫情列为生物安全重要内容[2]。此外,全球耐药性(Antibiotic resistance)问题也是当前学术界研究的热点,由此引起的“超级细菌(Super bugs)”在世界范围内的传播也成为政府部门普遍关注的潜在DBAs[8-9]。据世界卫生组织(World health organization,WHO)估算,如果不采取有效措施,到2050年,全球每年将有1 000万人因耐药性问题而失去生命,超过因癌症导致的死亡人数[10]。联合国环境署已将耐药性问题列为国际新型环境问题之一,而抗生素污染控制亦是2021年我国两会期间重要关键词,并将成为我国“十四五”期间迫切解决的环境问题之一[11-14]。因此,应对微生物耐药成为我国生物安全法适用的重要活动之一。据统计,我国约52%的抗生素用于畜禽养殖行业,这导致畜禽废弃物成为环境中抗生素抗性基因(Antibiotic resistance genes,ARGs)的重要来源[15-18],而ARGs在畜禽养殖行业的传播是当前关注的热点之一。
综上所述,基于我国生物安全法的要求,畜禽养殖行业的生物安全主要体现在动物疫情和微生物耐药问题。而畜禽粪污之于生物安全,主要体现在两个方面:(1)畜禽粪污是环境中ARGs重要的源和汇,如何防控畜禽粪污中ARGs的传播是适用于我国生物安全法中有关微生物耐药的防控;(2)导致动物疫情的病原体如禽流感病毒、非洲猪瘟病毒(African swine fever virus,ASFV)能够赋存于畜禽粪污中而进行传播,如何实现畜禽粪污中病原体的有效控制,减少动物疫情的传播是适用于我国生物安全法的内容之一。围绕DBAs在畜禽粪污中的环境行为开展研究,对畜禽养殖领域开展生物安全控制工作具有重要意义。因此,本文以畜禽粪污中DBAs(ARGs和病原体)为研究对象,从畜禽养殖行业污染物控制的角度,覆盖源头-生物处理-末端利用,通过文献综述解析DBAs在环境中的行为,总结畜禽粪污处理处置中DBAs在环境中的行为及对其控制的研究进展,为畜禽养殖行业的生物安全控制,防范畜禽养殖行业疫情,防控畜禽养殖源DBAs向环境扩散提供参考和借鉴。
1 畜禽粪污中DBAs的赋存特征根据我国生物安全法的内容,本文主要从微生物耐药性和能引起动物疫情的病毒等对DBAs进行分析总结。从微生物耐药性的角度,主要是指核酸水平的ARGs和细胞水平的耐药病原菌。从动物疫情暴发的角度,根据历年来我国动物疫情暴发的因素,主要是以病毒为主。因此,此部分内容主要是分别探讨ARGs、耐药病原微生物和病毒等DBAs在畜禽粪污中的赋存特征,以期为后续风险控制和治理提供基础数据。
1.1 ARGs由于抗生素在畜禽养殖行业的大量使用,畜禽粪便已成为环境中ARGs的重要源[15, 19]。以我国最大的畜禽养殖行业——生猪养殖为例,猪粪中检测到146种ARGs,每克干猪粪中ARGs的基因拷贝数高达1.3×1010,每万头存栏生猪养殖场,每天通过猪粪向环境中释放的ARGs的基因拷贝数高达4.0×1017±1.3× 1017,并且育肥猪释放的ARGs远超其他生猪养殖阶段[19-20]。猪粪中占主导的ARGs为大环内酯类(ermB,ermF)、四环素类(tetG,tetM)和磺胺类(sul1,sul2),每克干猪粪中ARGs的基因拷贝数(lg值),ermB含量为6.47~10.56,ermF为6.03~10.47,tetG为6.43~9.82,sul1和sul2为6.19~9.86。从抗生素抗性机制来看,猪粪中以酶失活(Antibiotic inactivation)和外排泵(Antibiotic efflux)的抗性基因占主导,分别为44.5% 和34.3%[20]。猪粪中ARGs的赋存特征与抗生素在畜禽养殖中的使用密切相关。生猪养殖过程中金霉素作为食品添加剂被广泛使用,而大环内酯类抗生素是生猪养殖过程中重要的治疗用药物,这导致了四环素类和大环内酯类ARGs在猪粪中广泛大量的存在[16]。随着研究不断深入,新型ARGs也不断被发现,如mcr-1首先在畜禽养殖领域被发现[11],而目前已在各种环境样品中被监测到,如污水处理厂、土壤、河流等[21-23]。自2016年发现mcr - 1以来,目前已发现39种MCR的变种ARGs,并推动了黏杆菌素在我国畜禽养殖领域的禁用,具有重要的科学和现实意义[24-25]。因此,如何进一步识别和鉴定畜禽养殖行业的新型ARGs,做到早发现、早预防也是当前领域的研究热点和值得深入探讨的科学问题。
ARGs作为环境中的一种新兴污染物,本质上属于脱氧核糖核酸,是一种化学污染物质,但其生物学特性决定了ARGs作为环境中污染物的特异性[26-27]。ARGs的化学特性决定了其降解性能,而其生物特性决定了其传播、增殖特性。ARGs在环境中主要是以胞内(Intracellular,iARGs)和胞外(Extracellular,eARGs)两种形式存在,同时通过转化和细胞死亡实现胞内和胞外的转变(图 1)[28]。畜禽粪污中的ARGs以iARGs为主(占比>99%),每克干猪粪中iARGs的基因拷贝数为2.15 × 1010,eARGs的基因拷贝数为6.29×107;每克干鸡粪中iARGs的基因拷贝数为1.01× 1010,eARGs的基因拷贝数为4.28×106 [29]。ARGs的化学污染性质主要体现在eARGs上,ARGs可在环境中实现降解和削减,且是胞外聚合物(Extracellular polymeric substances,EPS)和溶解性微生物产物(Soluble microbial products,SMP)的重要组成部分,但其生物学特性决定了eARGs可通过转化作用进入细胞成为iARGs。iARGs主要体现ARGs的生物学特性,通过宿主菌群的增殖实现ARGs的垂直转移(Vertical gene transfer,VGT);基于转导(Transduction)和转接(Conjugation)在不同宿主间实现基因的水平转移(Horizontal gene transfer,HGT)[30]。基于统计学的研究结果,从ARGs在畜禽粪污中的整体赋存特征来看,畜禽粪污中的微生物群落结构是ARGs赋存特性的决定性因子,即VGT对整体ARGs的赋存特性起决定作用[19-20]。如猪粪中微生物以厚壁菌门(Firmicutes)和拟杆菌门(Bacteroidetes)为主,这两类ARGs是大环内酯类和四环素类ARGs重要的宿主菌群,因此也决定了其在猪粪中的主导作用。但对于某个ARG在畜禽粪污中的赋存特征而言,其表现出了多种因素的协同作用。如水平转移元件(Mobile genetic elements,MGEs)intI1对sul1赋存的HGT作用,而典型sul1的宿主菌群Pseudomonas和Citrobacter在畜禽粪污中广泛存在,其VGT作用对于sul1在畜禽粪污中的赋存亦不可忽视。但很难界定哪种作用贡献更大,这是因为单次HGT的发生,可以通过VGT作用不断被扩大[30]。此外,尚无有关畜禽粪污中ARGs的HGT发生频率的研究,iARGs和eARGs间的转化研究也鲜有报道,这些都是值得深入研究的科学问题。
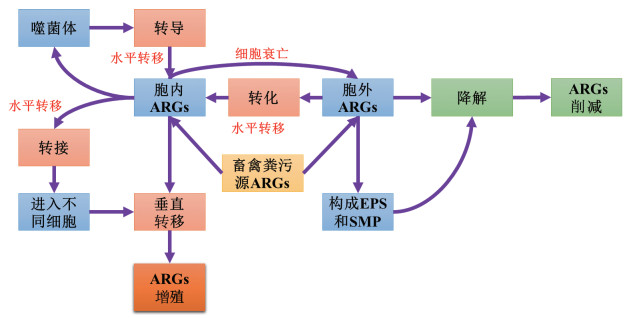
|
图 1 ARGs在环境中的赋存与归趋途径 Figure 1 The distribution and fate of ARGs in the environmental dimension |
畜禽粪污中含有大量病原微生物,典型的细菌种类包括大肠杆菌(Escherichia coli)、肠球菌(Enterococcus)、沙门氏菌(Salmonella)、金黄色葡萄球菌(Staphylococcus aureus)、弯曲杆菌(Campylobacter)等,在兽用抗生素的作用下,耐药病原菌对养殖场及周边环境造成较大威胁。肠球菌(Enterococcus)和金黄色葡萄球菌是畜禽养殖源典型的革兰氏阳性病原菌;沙门氏菌和大肠埃希氏菌(大肠杆菌)是畜禽养殖源典型的革兰氏阴性病原菌。对公众危害较大的耐药病原菌包括具有碳青霉烯、广谱头孢菌素和/或氟喹诺酮耐药性的肠杆菌科细菌,具有喹诺酮耐药性的弯曲杆菌,具有甲氧西林耐药性的金黄色葡萄球菌以及具有糖肽类耐药性的肠球菌等,其携带的ARGs如blaCTX-M、blaVIM、blaNDM、blaOXA-48-like、blaOXA-23、mcr、armA、vanA、cfr和optrA的关注度最高[31]。从猪粪水中分离的典型病原微生物,如不动杆菌(Acinetobacter)、Escherichia、丛毛单胞菌(Comamonas)、Enterococcus、假单孢菌(Pseudomonas),普遍具有多重耐药性,同时携带sul和tet等多种ARGs,并同时携带一类整合子基因intI1[32]。
近年来宏基因组方法被用于鉴定病原菌或耐药病原菌在环境中的分布,FANG等[33]分析了典型病原菌在猪粪污染的河流底泥(艰难梭菌Clostridium difficile、结核分枝杆菌Mycobacterium tuberculosis),河水(弗氏志贺氏菌Shigella flexneri、百日咳博氏菌Bordetella pertussis)及温室土壤(炭疽杆菌Bacillus anthracis,羊氏布鲁氏菌Brucella melitensis)中的分布,并发现其具有多重耐药性。宏基因组和宏转录组联用可用于揭示ARGs的赋存与表达,LIU等[34]指出质粒介导ARGs较染色体ARGs具有更高的表达。JIA等[35]发现紫外处理会导致污水中大环内酯-林可霉素- 链阳霉素B(MLSB)的抗性基因表达增高。基于此,如何区分耐药病原微生物ARGs赋存、表达与耐药病原菌毒力(Virulence)之间的关系,从耐药病原微生物的角度,解析畜禽粪污中耐药性问题可能导致的健康风险,仍是值得深入探讨的主题。
1.3 典型病毒畜禽粪污中富含大量的病毒,但有关畜禽粪污中病毒组(Virome)的研究非常有限。2021年发表的有关牛粪污中病毒组的研究表明,牛粪污中发现7 682种病毒操作分类单元(Virus operational taxonomic units,vOTUs),种类繁多,其中91% 的病毒属于有尾噬菌体目(Caudovirales),10%的vOTUs属于温和型噬菌体(Temperate phages),说明牛粪污中以烈性噬菌体(Lytic phages)为主。进一步分析发现,只有两个vOTUs为已知,分别为Mycoplasma bacteriophage L2和Streptococcus phage Javan630,绝大多数病毒无法做进一步分类,说明牛粪污中绝大多数病毒目前仍然未知[36]。有关畜禽粪污中病毒组的研究,未知领域较多,还需要进一步深入开展。
本文主要从生物安全的角度,分析导致重大动物疫情的典型病毒在畜禽粪污中的赋存特征,而非洲猪瘟病毒(African swine fever virus,ASFV)导致的非洲猪瘟(African swine fever,ASF)疫情对我国生猪养殖行业的影响巨大[37-38]。ASF目前尚无有效治疗方法和疫苗防控措施,致死率近100%[39]。ASFV属于非洲猪瘟病毒科(Asfarviridae)、非洲猪瘟病毒属(Asfivirus),是目前唯一已知的dsDNA虫媒病毒[40]。ASFV的自然宿主为非洲疣猪,ASFV在自然宿主体内表现为亚临床(subclinical)特性,但感染家猪和野猪后,表现为一种高度传染性和致死性的出血性病毒病[41]。自我国2018年8月首例ASF病例确诊以来,ASFV在我国境内传播迅速,且范围广,给畜禽养殖生产造成极大经济损失[42]。生猪感染ASFV后,ASFV能够在畜禽粪污中体现,并表现出较强的环境稳定性。有研究表明,ASFV在畜禽粪污中能够稳定存在超过100 d[6]。但也有研究表明,ASFV在猪粪中的存活时间同温度有较大关系,在4 ℃条件下保存,8 d依然有传染性,而在37 ℃下能够保留3~4 d[43]。值得注意的是,ASFV作为一种dsDNA病毒,DNA在环境中的持久性为基于畜禽粪污提前预警ASF的暴发提供了可能。例如,有研究表明,感染ASF的野外疣猪尸体填埋一段时间(18~ 440 d)后,基于PCR的方法仍能够检测到ASFV的DNA片段,但通过培养方法无法获得ASFV,这说明病毒已经被成功灭活,但是其核酸DNA却拥有更长的半衰期[44]。目前有关ASFV在畜禽粪污中的含量研究较少,疫情发展同畜禽粪污中ASFV丰度之间的关系亦尚未可知。探讨畜禽粪污中ASFV与疫情的关系,开发畜禽粪污中ASFV核酸残留的快速监测方法,对预防疫情、明确疫情发展趋势具有重要的理论和现实依据,值得深入探讨。
2 畜禽粪污处理处置过程中DBAs的归趋特征本节主要从畜禽粪污处理处置的技术路线开展DBAs的归趋特征解析。目前我国畜禽粪污的处理处置方式以最终去向划分,主要包括资源化利用和达标排放[45]。资源化利用又包括堆肥-土地利用、厌氧发酵-土地利用,堆肥和厌氧是核心生物处理工艺,而土地利用是最终归宿。达标排放指畜禽养殖废水经生物处理,达到国家相关标准后,向周边环境进行排放。畜禽粪污处理方式不同、受纳环境不同,使得畜禽粪污中的DBAs归趋存在较大差别。本节内容主要以厌氧和堆肥等生物处理工艺为主要研究对象,考察DBAs的归趋,明确关键工艺参数对生物安全风险因子的影响。
2.1 畜禽粪污厌氧消化过程中DBAs的赋存与归趋特征根据我国2030年“碳达峰”和2060年“碳中和”的需求,厌氧消化作为畜禽粪污重要的资源化利用方式,将成为我国畜禽粪污处理处置的重要手段,其对环境中ARGs传播与扩散的控制作用也得到了广泛证实[46-47]。因此,如何实现厌氧消化产甲烷和ARGs削减的协同耦合是同步实现畜禽粪污资源化和抗生素抗性污染控制的关键。
整体而言,厌氧消化能够实现ARGs的总体削减,对ARGs的HGT作用具有一定的抑制作用[48-49]。多项研究表明,厌氧消化对猪粪中高丰度ermB能够有效削减[50-52]。BENERAGAMA等[53-54]发现中温和高温厌氧消化对猪粪中多重耐药抗性基因均有很高的去除作用,去除率最高达到100%。但在某些特定ARGs和一定条件下却存在相反的结论,例如SUN等[55]研究了厌氧消化过程中tetC、sul2、ermB、gyrA的丰度变化,发现其增加了2倍以上。SUI等[56-57]的研究也指出在某一养殖场粪污厌氧消化中,ARGs、intI1的丰度(lg值)分别升高了0.41和0.51,即使高温厌氧消化也不能降低某些ARGs的丰度。这些结果同厌氧消化的工艺操作条件具有较大关系[48-49],影响畜禽粪污中ARGs归趋的因素繁多,包括反应器构型[58-59]、操作条件[52, 60-61]、底物组成差异[62]等,如单相和两相反应器对厌氧消化中ARGs归趋差别的影响,工艺操作条件如固体停留时间(SRT)、含固率(TS%)、温度等对ARGs转归的影响[59, 63],以及鸡粪和猪粪厌氧消化中ARGs归趋的差别。但综合因素解析,微生物群落结构被认为是厌氧消化中决定ARGs归趋的主导因素,特定条件下某种宿主菌群的大量增殖/衰减,不可避免地会导致所携带的ARGs的富集/削减。有研究表明,ARGs的宿主菌群同时亦是厌氧消化中重要的功能菌群[64-65]。ZHANG等[66]研究了微生物群落结构(Microbial community,MC)、MGEs、重金属(Heavy metal,HMs)和ARGs的关系,结果发现这三者对ARGs影响最大的是MC,其次是MGEs和HMs,这一结论在不同研究中均得到了证实[55, 67-68]。这说明ARGs在厌氧消化中的归趋主要是由其宿主菌群,即VGT所决定。厌氧消化条件的改变,导致微生物群落结构的演替,作为ARGs的宿主菌群必然会影响到其归趋特征。但MGEs介导的HGT对厌氧消化中ARGs的作用亦不容忽视,MGEs同ARGs共存于同一基因重叠群(Contigs)的情况广泛存在,携带有ARGs的质粒在厌氧消化中的转接现象也被广泛验证[48, 69]。但是,厌氧消化中ARGs的HGT频率有待进一步深入研究,其对ARGs归趋的总体贡献虽然有限,但不容忽视。虽然ARGs在畜禽粪污厌氧消化中存在,但是否表达并行使功能,相关研究依然缺乏。此外,在已知ARGs在厌氧消化中行为和归趋机制的基础上,如何开展ARGs的特异性或靶向控制是当前领域的研究前沿。
有关ASFV在厌氧消化中的归趋研究较少。但DAVIES等[70]基于ASFV在畜禽粪污半衰期的研究表明,畜禽粪污中的ASFV在37 ℃条件下保存3~4 d后仍然保持有感染性,并认为在厌氧消化条件下,通过pH和代谢产物的作用,能够削减ASFV在畜禽粪污中的半衰期[70]。但也有研究表明,40 ℃、4 h能够实现猪粪中ASFV的灭活,并指出高温厌氧消化在数小时内可以实现ASFV的灭活,中温厌氧消化在数天内也能实现ASFV的灭活[6]。通常畜禽粪污厌氧消化的SRT保持在15~30 d,能够实现ASFV的有效削减和控制。但目前有关厌氧消化对ASFV灭活作用的研究均为实验室添加后的观察,缺乏现场研究,开展ASFV在实际畜禽粪污厌氧消化中的行为,探讨其核酸DNA在厌氧消化过程中的行为,对于减少畜禽粪污污染导致的ASFV传播具有重要意义。
2.2 畜禽粪污堆肥过程中DBAs的赋存与归趋特征堆肥对畜禽粪污中ARGs的削减作用虽然存在相反的结果,但目前仍以削减为主。如在羊粪好氧堆肥过程中发现,虽然抗生素被有效削减,但tet和sul等ARGs的丰度富集了9倍[71]。在鸡粪堆肥的现场研究中发现,ARGs的相对丰度削减了67.3% 以上[72]。有关牛粪和猪粪堆肥的研究也都表明了堆肥工艺对ARGs的有效削减作用,但不同研究中ARGs的削减/ 去除率差别较大,这同堆肥的不同工艺条件息息相关,如辅料的类型及用量、曝气条件、工艺条件、温度变化等[73-75]。从总体来看,堆肥对于畜禽粪污中的ARGs能够实现较好的控制。如畜禽粪污中的mcr-1在规模化牛粪堆肥中,22 d后便低于检出限[76]。虽然tetX、tetM、sul1等在有关猪粪堆肥的研究中得到富集[77],但这种富集普遍归因于宿主菌群的变化,微生物群落结构的变化在堆肥过程中依然起主导作用。高温期对于畜禽粪污堆肥过程中ARGs的削减起到了关键作用,超高温堆肥(>70 ℃)对畜禽粪污中ARGs能够起到更好的削减作用[78]。此外,堆肥对畜禽粪污中ARGs的表达具有较好的削减作用,并且不同阶段ARGs的表达变化情况差别较大[79]。堆肥对四环素类ARGs表达的削减效果明显,而对磺胺类和喹诺酮类ARGs的表达无作用,并在高温期有所增加。进一步研究表明,ARGs的表达同其宿主的活性具有显著相关性。畜禽粪污中ARGs的组成在堆肥升温期、高温期、降温期和后腐熟期发生动态变化,这同ARGs宿主菌群随外界条件改变而进行的演替呈现显著相关性。但目前有关畜禽粪污中iARGs和eARGs在堆肥过程中的演替和归趋特征研究尚缺乏。
堆肥对肠道病毒具有较好的灭活作用[80-81],目前尚无有关畜禽粪污中ASFV在堆肥过程中的变化和演替研究。但ASFV的抗热性较差,在60 ℃下经过10 min,或者在55 ℃下经过30 min,ASFV便会失去活性,堆肥工艺一般需要55 ℃至少维持5 d以上,足以实现ASFV的灭活。但需要进一步开展相关实验的验证研究。此外,ASFV灭活后残留的dsDNA在堆肥中的残留也值得关注,作为ASFV在环境中行为的生物标记物,需对其开展进一步研究。
2.3 畜禽粪污土地利用过程中DBAs的赋存与归趋特征目前,畜禽粪污产品(如堆肥、沼液、沼渣等)土地利用对土壤中ARGs普遍具有富集作用,且该作用因土壤类型而异,这一点在不同研究中已经得到了充分证实[82-85]。根据已有研究,对于畜禽粪污产品的土地利用过程,可以提出如下假设:在0~10 d,土壤内源微生物因为营养物质的进入,开始大幅增殖,微生物群落结构发生显著改变;在10~30 d,源于畜禽粪污的外源微生物,由于无法适应土壤环境逐渐衰减、死亡,其所携带的ARGs也逐渐释放至环境中发生降解;随着时间的推移(>30 d),虽然绝大部分ARGs能够恢复到初始值,但由于某些eARGs在环境中的可持续性,使ARGs的丰度仍然高于空白组。猪粪施用土壤后的结果也表明,耐药性微生物在130 d后,其头孢类耐药菌的丰度仍然高于空白组,并且这类得到富集的头孢类耐药菌是来自土壤而非猪粪[86]。猪粪污中耐药沙门氏菌和大肠杆菌的赋存时间受环境因素影响较大,耐药菌浓度在沙质土壤中衰减速率受粪污施用时间和降雨频次影响[87],耐药病原菌在壤质土中的赋存时间较其他类型土壤(沙土、黏土)短[87-88]。这些都说明畜禽粪污进入土壤后对土壤中ARGs和耐药菌(Antibiotic resistant bacteria,ARB)所带来的影响的复杂性,一方面,外源微生物由于无法适应土壤环境,导致ARGs的宿主菌群死亡,释放出eARGs,由于eARGs在环境中的可持续性,半衰期远高于其宿主菌群;另一方面,土壤内源微生物由于畜禽粪污所带来的营养物质、选择压力等因素,导致内源ARB的快速增殖,从而引起ARGs和ARB的进一步增殖。这些结果进一步说明在土壤中,微生物群落结构的演替依然是ARGs归趋的主导因素,而这一结论已经得到了广泛证实[89-92]。
从长期施用畜禽粪污对土壤中ARGs归趋的影响来看,ARGs均得到富集,微生物群落也发生了显著变化,短期施用效果的研究能够说明单次施用带来的影响,这种影响虽然能够使ARGs和ARB得到富集,但作用有限;长期施用条件下,土壤环境逐渐发生改变,使得更多的畜禽粪污源微生物能够适应土壤环境,并逐渐累积,直到达到一种土壤微生物和畜禽粪污源微生物的一个平衡,这种平衡的建立,很可能需要10~20 a,甚至更长时间。但无论是长期还是短期施肥所带来的效应,微生物群落结构依然是ARGs在土壤中归趋的主导因素,即土壤内源微生物增殖、外源微生物衰减和eARGs残留综合作用下的结果。但土壤中ARGs的HGT依然在发生,并且畜禽粪污产品的施用增加了土壤中ARGs水平转移发生的频率[93]。虽然HGT发生的频率有限,对土壤中ARGs归趋的贡献有限,但其带来的风险不可预估。土壤微环境如土壤类型对于HGT的发生具有显著影响[94-95](图 2)。前期研究表明,黄土中HGT对ARGs归趋的贡献明显高于红土和黑土[96-97]。但目前有关畜禽粪污中ARGs在土壤中归趋的研究,缺乏iARGs和eARGs的区分,明确不同类型eARGs和iARGs在土壤中的归趋,对于控制其在土壤中的行为具有重要的指导作用。

|
图 2 土壤类型对畜禽粪污产品土地利用中ARGs归趋的影响 Figure 2 Effects of soil types on the fate of ARGs during the land application of livestock wastes |
虽然对畜禽粪污土地利用过程中ASFV的环境行为研究缺乏,但是对于ASFV在不同土壤类型中的稳定性研究表明,土壤pH、土壤结构和温度对ASFV的稳定性具有显著影响[98]。ASFV在无菌沙土中能够存活至少3周,沙滩沙土中存活2周以上,庭院土壤中存活1周,沼泽土壤中存活3 d,但在酸性森林土壤中并未筛选出活性ASFV,通过使用柠檬酸(Citric acid)和氢氧化钙(Calcium hydroxide)改变土壤pH后,各类型土壤样品中均未再筛选出活性ASFV。因此,在考察受ASFV感染的畜禽粪污土地利用过程中,应充分考虑到土壤类型,并采用适当的消毒措施,以减少土壤中活性ASFV所带来的风险。畜禽粪污土地利用过程中环境因素等对病毒组的影响仍然需要深入研究,无论是土壤内源病毒还是外源病毒,目前研究均比较有限,亟需深入开展相关研究,这对识别、鉴定、控制新兴DBAs在环境中的传播风险具有重要的理论意义和实践价值。
3 畜禽粪污中DBAs的控制策略研究本节将从防控动物疫情和耐药性的角度,提出畜禽粪污的处理处置策略和建议。畜禽粪污不是动物疫情暴发的原因,却是动物疫情传播的重要因素,如ASFV并不源于畜禽粪污,而是来源于自然界中的非洲疣猪,但畜禽粪污对其传播却具有重要作用;而畜禽粪污中耐药性问题却相反,畜禽粪污是耐药性和耐药病原体传播和暴发的重要源,环境中“超级细菌”的产生同畜禽养殖行业密不可分,而畜禽粪污是向环境中传播超级病原体的重要载体。本文从DBAs的不同来源,分别提出不同视角的控制策略。
3.1 畜禽粪污中耐药性的控制从生物安全风险控制的角度来说,畜禽粪污中耐药性问题是一种DBAs的源。畜禽养殖行业抗生素的广泛使用,导致畜禽粪污可能成为“超级细菌”的温床,并通过环境传播,从而引起生物安全风险。因此,加强畜禽养殖行业耐药性污染的控制至关重要。畜禽粪污中耐药性污染控制的关键在于源头减量,需要加强生物处理过程控制,明确末端利用风险。从源头明确耐药性产生的主要因素,减少抗生素的使用,提倡无抗养殖,并且由于重金属等其他共选择化合物的存在,减少饲料中重金属元素如Cu、Zn、As等的使用[99]。从ARGs在畜禽粪污赋存特征及后续生物处理、土地利用过程中的归趋来看,生物处理过程是削减ARGs的关键,因此,加强生物处理过程对ARGs和ARB的削减也是目前研究的热点之一。例如,通过铁系化合物的投加实现厌氧消化中ARGs的削减[51, 64, 66, 68, 100],高温厌氧消化对ARGs的高效削减[52, 101],沸石粉的投加实现堆肥过程中ARGs的控制[52, 102-103],延长堆肥高温期、超高温堆肥等对ARGs的有效控制[78],γ射线、紫外消毒等工艺对ARGs的削减[104],这些工艺条件的优化和改善有助于削减畜禽粪污生物处理过程中的ARGs。但目前有关畜禽粪污中ARGs的削减依然缺乏特异性,上述条件更多是对整体微生物的灭活或改变,但对于ARGs和ARB的削减缺乏靶向性和针对性。如何针对ARB或ARGs开展针对性、特异性的削减是值得深入探讨的科学问题,而噬菌体的筛选和使用为特异性、靶向性控制ARB提供了理论可能。
利用噬菌体杀灭耐药性细菌,实现抗生素抗性污染的控制是当前的一个研究热点,其对环境中抗生素抗性的靶向控制具有重要作用。如YU等[105]将广谱噬菌体“鸡尾酒”以107 pfu·mL-1的剂量添加到活性污泥中,成功控制了具有多重耐药性的E. coli NDM-1,并且没有影响污泥系统的活性。由于噬菌体在食品和医疗行业已广泛且产业化应用,人们并不担心噬菌体本身会对人体产生的不利影响[106-107]。目前影响噬菌体在畜禽粪污中耐药病原微生物防控使用的因素主要有以下几个方面:(1)噬菌体高度专一性导致的较窄宿主谱。针对这个问题,开发基于多种噬菌体并用的“鸡尾酒”疗法逐渐引起了人们的关注。有研究者提出建立“噬菌体银行”以及“噬菌体鸡尾酒银行”,即将已知的噬菌体及其相关敏感宿主进行数据分类并构建检索工具,在发现致病菌后,可以迅速找出相关敏感噬菌体并调制出合适的“噬菌体鸡尾酒”[108]。(2)噬菌体通过转导作用对环境ARGs的传播作用。转导是由噬菌体介导的ARGs水平转移的方式之一。有研究表明,噬菌体是环境中ARGs重要的库,其可能会携带有毒力基因或ARGs,并通过转导作用将ARGs水平转移给宿主菌群[109-110]。因此,在将噬菌体投入实际环境应用时,必须明确所使用的噬菌体有无毒性或ARGs。(3)噬菌体对环境较敏感,影响因素尚不清晰。环境参数的变化对噬菌体的有效性具有显著影响,如盐度、温度、钙镁离子等[111],针对不同的应用场景,环境适应性研究有待开展。例如,针对畜禽粪污已成为环境中ARGs和耐药菌的重要源这一问题,利用噬菌体的专一性,开展基于耐药菌防控的靶向噬菌体筛选,并基于畜禽粪污生物处理环境,解析噬菌体的环境适应性,对畜禽粪污资源化利用和达标排放过程中ARGs和耐药菌的控制具有重要意义。
3.2 畜禽粪污中动物疫情病原体的控制从生物安全的角度来看,畜禽粪污是动植物疫情病毒的汇,而非源,但其对动植物疫情病毒的传播起到了一定的作用。以ASFV为例,ASFV并非源于畜禽养殖行业,而是自然界中的病毒进入畜禽养殖行业并传播,而畜禽粪污起到了其向环境传播的载体作用。因此,加强动物疫情病原体的鉴别、预警和防控至关重要,而畜禽粪污是实现这一点的关键载体,其源头控制也明显区别于畜禽粪污中的耐药性问题。通过加强畜禽养殖场同外界环境的接触,加强从业相关人员入舍前的消毒至关重要。基于畜禽粪污开展动物疫情病原体的源头控制,重在病原体的鉴别和预警,因此亟需开展相关动物疫情病原体的快速检测方法研究,如适用于畜禽粪污的ASFV抗原检测和qPCR检测方法等[40-41]。
在疫情暴发期间,加强畜禽粪污生物处理过程和消毒措施,减少因畜禽粪污处置不当导致的病毒传播。例如,可通过增加厌氧消化SRT、延长堆肥高温期、开发超高温堆肥等技术手段,强化生物处理工艺对ASFV等病毒的控制力度,探讨紫外消毒、氯/臭氧等化学药剂消毒等在畜禽粪污处理不同环节的适用性等,保障在畜禽粪污土地利用前实现其完全灭活。在土地利用过程中,可通过消毒措施进一步实现土壤中ASFV等病毒的灭活。据文献报道,能够实现DNA病毒灭活的方法对于ASFV病毒均能实现较好的削减效果,如柠檬酸、氢氧化钙等[6]。在环境领域,如何实现ASFV等导致动植物疫情病毒的特异性控制,也是值得深入探讨的主题。
4 总结与展望依据我国生物安全法的相关要求,本文从环境工程和环境微生物学的角度,针对畜禽粪污中DBAs的环境行为及其控制,总结了当前研究进展,并提出今后亟需开展的研究方向。
(1)明确畜禽粪污处理处置过程中的赋存与环境归趋特征。基于DBAs的分子生物学组成特征,重在防范动物疫情的传播,全面解析核酸(如ARGs)-核酸+蛋白(如病毒)-细胞结构(如耐药菌)在畜禽粪污处理处置过程中的赋存与环境归趋特征,并深入开展风险评价研究。
(2)开发畜禽粪污中新兴DBAs的识别、鉴定和快速检测方法。畜禽粪污是DBAs的汇和源,针对其开展畜禽养殖领域新兴DBAs的识别和鉴定,具有可行性和代表性,如新型ARGs的发现、畜禽粪污中病毒组的深入研究、新型“超级细菌”的识别等。针对不同DBAs特点,充分利用DBAs遗传物质(DNA/RNA)在环境中的持久性,开发快速检测方法,实现动物疫情的快速预警,具有重要的理论意义和现实价值。
(3)加强畜禽粪污生物处理过程中DBAs控制的靶向技术研发。目前,针对畜禽粪污中DBAs的控制,主要还是依赖于对微生物的广谱灭活技术(紫外、强氧化剂等),缺乏特异性和靶向性,可以利用DBAs的特点,开展靶向性控制技术研究。如针对烈性噬菌体对耐药菌的靶向性杀灭作用,开展烈性噬菌体的筛选、组合和环境适用性研究等。
(4)加强畜禽粪污中有机污染、无机污染和生物污染的协同控制技术研究。畜禽粪污的污染防控,已经从有机污染防控(强调有机物去除、转化和腐熟),减少黑臭和施肥烧苗等现象,无机污染防控(重金属、氮、磷等),解决重金属在植物中的富集和面源污染问题,发展到了生物污染防控,因此,结合碳达峰/碳中和的需求,在资源化利用和达标处理的前提下,加强DBAs的控制,开发有机污染、无机污染和生物污染的协同控制技术势在必行。
| [1] |
中华人民共和国生态环境部, 国家统计局, 中华人民共和国农业农村部. 第二次全国污染源普查公报[R/ OL]. (2020-06-10)[2021- 09-15]. http://www.gov.cn/xinwen/2020-06/10/content_5518391.htm. Ministry of Ecology and Environment of China, National Bureau of Statistics, Ministry of Agriculture and Rural Affairs of China. Bulletion of the Second National Census of Pollution Sources[R / OL]. (2020-06- 10)[2021-09-15]. http://www.gov.cn/xinwen/2020-06/10/content_5518391.htm. |
| [2] |
中华人民共和国主席令. 中华人民共和国生物安全法[EB/OL]. (2020-10-18)[2021-09-15]. http://www.gov.cn/xinwen/2020-10/18/content_5552108.htm. Order of the President of the People' s Republic of China. Biosafety Law of the People' s Republic of China[EB/ OL]. (2020-10-18)[2021-09-15]. http://www.gov.cn/xinwen/2020-10/18/content_5552108.htm. |
| [3] |
何文巧, 陈清. SARS-CoV-2溯源新进展[J]. 南方医科大学学报, 2020, 40(12): 1838-1842. HE W Q, CHEN Q. Progress in source tracking of SARS-CoV-2[J]. Journal of Southern Medical University, 2020, 40(12): 1838-1842. DOI:10.12122/j.issn.1673-4254.2020.12.22 |
| [4] |
王晓钰, 崔立. 新型冠状病毒SARS-CoV-2的研究进展[J]. 上海交通大学学报(医学版), 2020, 40(2): 149-156. WANG X Y, CUI L. A source-seeking analysis and its implication on the transmission of severe acute respiratory syndrome coronavirus 2(SARS-CoV-2)[J]. Journal of Shanghai Jiao Tong University(Medical science), 2020, 40(2): 149-156. DOI:10.3969/j.issn.1674-8115.2020.02.002 |
| [5] |
CHYBA C F. Biological security in a changed world[J]. Science, 2001, 293(5539): 2349. DOI:10.1126/science.293.5539.2349 |
| [6] |
BLOME S, FRANZKE K, BEER M. African swine fever: A review of current knowledge[J]. Virus Research, 2020, 287: 198099. DOI:10.1016/j.virusres.2020.198099 |
| [7] |
BI Y, LI J, LI S, et al. Dominant subtype switch in avian influenza viruses during 2016—2019 in China[J]. Nature Communications, 2020, 11(1): 1-12. DOI:10.1038/s41467-019-13993-7 |
| [8] |
ABAT C, RAOULT D, ROLAIN J-M. Antibiotic resistance: Are we living a nightmare?[J]. Clinical Microbiology and Infection, 2018, 24(6): 568-569. DOI:10.1016/j.cmi.2018.01.004 |
| [9] |
LÁZÁR V, KISHONY R. Transient antibiotic resistance calls for attention[J]. Nature Microbiology, 2019, 4: 1606-1607. DOI:10.1038/s41564-019-0571-x |
| [10] |
O' NEILL J. Tackling drug-resistant infections globally: Final report and recommendations[R / OL]. (2016-05)[2021-09-15]. https://apo.org.au/sites/default/files/resource-files/2016-05/apo-nid63983.pdf.
|
| [11] |
LIU Y Y, WANG Y, WALSH T R, et al. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: A microbiological and molecular biological study[J]. The Lancet Infectious Diseases, 2016, 16(2): 161-168. DOI:10.1016/S1473-3099(15)00424-7 |
| [12] |
NG C, LE T H, GOH S G, et al. A comparison of microbial water quality and diversity for ballast and tropical harbor waters[J]. PLoS One, 2015, 10(11): e0143123. DOI:10.1371/journal.pone.0143123 |
| [13] |
TONG J, LIU J, ZHENG X, et al. Fate of antibiotic resistance bacteria and genes during enhanced anaerobic digestion of sewage sludge by microwave pretreatment[J]. Bioresource Technology, 2016, 317: 37-43. |
| [14] |
DOS SANTOS D F K, ISTVAN P, QUIRINO B F, et al. Functional metagenomics as a tool for identification of new antibiotic resistance genes from natural environments[J]. Microbial Ecology, 2017, 73(2): 479-491. DOI:10.1007/s00248-016-0866-x |
| [15] |
张俊亚, 魏源送, 陈梅雪, 等. 畜禽粪便生物处理与土地利用全过程中抗生素和重金属抗性基因的赋存与转归特征研究进展[J]. 环境科学学报, 2015, 35(4): 935-946. ZHANG J Y, WEI Y S, CHEN M X, et al. Occurrence and fate of antibiotic and heavy metal resistance genes in the total process of biological treatment and land application of animal waste: A review[J]. Acta Scientiae Circumstantiae, 2015, 35(4): 935-946. |
| [16] |
隋倩雯, 张俊亚, 魏源送, 等. 畜禽养殖过程抗生素使用与耐药病原菌及其抗性基因赋存的研究进展[J]. 生态毒理学报, 2015, 10(5): 20-34. SUI Q W, ZHANG J Y, WEI Y S, et al. Veterinary antibiotics use, occurrence of antibiotic resistance pathogen and its antibiotic resistance genes in animal production: An overview[J]. Asian Journal of Ecotoxicology, 2015, 10(5): 20-34. |
| [17] |
隋倩雯, 张俊亚, 魏源送, 等. 畜禽养殖废水生物处理与农田利用过程抗生素抗性基因的转归特征研究进展[J]. 环境科学学报, 2016, 36(1): 16-26. SUI Q W, ZHANG J Y, WEI Y S, et al. Fate of antibiotic resistance genes in the process of biological treatment and land application of animal wastewater: An overview[J]. Acta Scientiae Circumstantiae, 2016, 36(1): 16-26. |
| [18] |
ZHU Y G, JOHNSON T A, SU J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [19] |
ZHANG J Y, LU T D, CHAI Y F, et al. Which animal type contributes the most to the emission of antibiotic resistance genes in largescale swine farms in China?[J]. Science of the Total Environment, 2019, 658: 152-159. DOI:10.1016/j.scitotenv.2018.12.175 |
| [20] |
ZHAO Y, SU J Q, AN X L, et al. Feed additives shift gut microbiota and enrich antibiotic resistance in swine gut[J]. Science of the Total Environment, 2017, 621: 1224-1232. |
| [21] |
马奔, 黄雅梦, 王若楠, 等. 城市污水厂MCR-1基因及其携带菌的污染[J]. 中国环境科学, 2018, 38(4): 1433-1440. MA B, HUANG Y M, WANG R N, et al. The pollution of MCR-1 and MCR-1 hosting bacteria in municipal wastewater treatment plants[J]. China Environmental Science, 2018, 38(4): 1433-1440. |
| [22] |
HAENNI M, POIREL L, KIEFFER N, et al. Co-occurrence of extended spectrum β lactamase and MCR-1 encoding genes on plasmids[J]. The Lancet Infectious Diseases, 2016, 16(3): 281-282. |
| [23] |
WANG R, VAN DORP L, SHAW L P, et al. The global distribution and spread of the mobilized colistin resistance gene mcr-1[J]. Nature Communications, 2018, 9(1): 1-9. DOI:10.1038/s41467-017-02088-w |
| [24] |
WALSH T R, WU Y. China bans colistin as a feed additive for animals[J]. The Lancet Infectious Diseases, 2016, 16(10): 1102-1103. DOI:10.1016/S1473-3099(16)30329-2 |
| [25] |
ALCOCK B P, RAPHENYA A R, LAU T T Y, et al. CARD 2020:Antibiotic resistome surveillance with the comprehensive antibiotic resistance database[J]. Nucleic Acids Research, 2020, 48(D1): D517-D525. |
| [26] |
POVOLO V R, ACKERMANN M. Disseminating antibiotic resistance during treatment[J]. Science, 2019, 364(6442): 737-738. DOI:10.1126/science.aax6620 |
| [27] |
ANDERSSON D I, HUGHES D. Antibiotic resistance and its cost: Is it possible to reverse resistance?[J]. Nature Reviews Microbiology, 2010, 8(4): 260-271. DOI:10.1038/nrmicro2319 |
| [28] |
DONG P, WANG H, FANG T, et al. Assessment of extracellular antibiotic resistance genes(eARGs) in typical environmental samples and the transforming ability of eARG[J]. Environment International, 2019, 125: 90-96. DOI:10.1016/j.envint.2019.01.050 |
| [29] |
李萍, 卢铁东, 辛苑, 等. 底物类型对厌氧消化过程中胞内外抗生素抗性基因归趋的影响研究[J]. 环境科学学报, 2021, 41(6): 2079-2091. LI P, LU T D, XIN Y, et al. Effects of substrate types on the fate of intracellular and extracellular antibiotic resistance genes in anaerobic digestion[J]. Acta Scientiae Circumstantiae, 2021, 41(6): 2079-2091. |
| [30] |
BENGTSSON-PALME J, LARSSON D G J. Antibiotic resistance genes in the environment: Prioritizing risks[J]. Nature Reviews Microbiology, 2015, 13(6): 396. |
| [31] |
KOUTSOUMANIS K, ALLENDE A, ÁLVAREZ-ORDÓÑEZ A, et al. Role played by the environment in the emergence and spread of antimicrobial resistance(AMR)through the food chain[J]. EFSA Journal, 2021, 19(6): 6651. |
| [32] |
ZOU Y, XIAO Y, WANG H, et al. New insight into fates of sulfonamide and tetracycline resistance genes and resistant bacteria during anaerobic digestion of manure at thermophilic and mesophilic temperatures[J]. Journal of Hazardous Materials, 2020, 384: 121433. DOI:10.1016/j.jhazmat.2019.121433 |
| [33] |
FANG H, HAN L, ZHANG H, et al. Dissemination of antibiotic resistance genes and human pathogenic bacteria from a pig feedlot to the surrounding stream and agricultural soils[J]. Journal of Hazardous Materials, 2018, 357: 53-62. DOI:10.1016/j.jhazmat.2018.05.066 |
| [34] |
LIU Z, KLÜMPER U, LIU Y, et al. Metagenomic and metatranscriptomic analyses reveal activity and hosts of antibiotic resistance genes in activated sludge[J]. Environment International, 2019, 129: 208-220. DOI:10.1016/j.envint.2019.05.036 |
| [35] |
JIA S, LI T, ZHANG X X. Integrated metagenomic and metatranscriptomic analyses of ultraviolet disinfection effects on antibiotic resistance genes and bacterial communities during wastewater treatment[J]. Ecotoxicology, 2021, 30(8): 1610-1619. DOI:10.1007/s10646-020-02313-1 |
| [36] |
COOK R, HOOTON S, TRIVEDI U, et al. Hybrid assembly of an agricultural slurry virome reveals a diverse and stable community with the potential to alter the metabolism and virulence of veterinary pathogens[J]. Microbiome, 2021, 9(1): 1-17. DOI:10.1186/s40168-020-00939-1 |
| [37] |
何庆, 邹亚文, 王东亮, 等. 非洲猪瘟病毒跨膜蛋白CD2v的结构与功能研究进展[J]. 病毒学报, 2021, 37(5): 1244-1251. HE Q, ZOU Y W, WANG D L, et al. Research progress on the structure and function of the transmembrane protein CD2v of African swine fever virus[J]. Chinese Journal of Virology, 2021, 37(5): 1244-1251. |
| [38] |
GUINAT C, GOGIN A, BLOME S, et al. Transmission routes of African swine fever virus to domestic pigs: Current knowledge and future research directions[J]. Veterinary Record, 2016, 178(11): 262-267. DOI:10.1136/vr.103593 |
| [39] |
ZHANG J, RODRÍGUEZ F, NAVAS M J, et al. Fecal microbiota transplantation from warthog to pig confirms the influence of the gut microbiota on African swine fever susceptibility[J]. Scientific Reports, 2020, 10(1): 1-14. DOI:10.1038/s41598-019-56847-4 |
| [40] |
SCHODER M E, TIGNON M, LINDEN A, et al. Evaluation of seven commercial African swine fever virus detection kits and three Taq polymerases on 300 well-characterized field samples[J]. Journal of Virological Methods, 2020, 280: 113874. DOI:10.1016/j.jviromet.2020.113874 |
| [41] |
BELLINI S, RUTILI D, GUBERTI V. Preventive measures aimed at minimizing the risk of African swine fever virus spread in pig farming systems[J]. Acta Veterinaria Scandinavica, 2016, 58: 82. DOI:10.1186/s13028-016-0264-x |
| [42] |
蒙嵘, 王占斌, 张彦, 等. 从生物安全角度看非洲猪瘟的防控[J]. 当代畜禽养殖业, 2021(3): 54-56. MENG R, WANG Z B, ZHANG Y, et al. The prevention and control of African swine fever from the perspective of biosecurity[J]. Contemporary Livestock and Poultry Industry, 2021(3): 54-56. DOI:10.3969/j.issn.1005-5959.2021.03.021 |
| [43] |
MITEVA A, PAPANIKOLAOU A, GOGIN A, et al. Epidemiological analyses of African swine fever in the European Union(November 2018 to October 2019)[J]. EFSA Journal, 2020, 18(1): 1-107. |
| [44] |
ZANI L, MASIULIS M, BUŠAUSKAS P, et al. African swine fever virus survival in buried wild boar carcasses[J]. Transboundary and Emerging Diseases, 2020, 67(5): 2086-2092. |
| [45] |
畜禽养殖污染防治项目建设与投资技术指南》编制说明[Z]. Guidelines for the construction and investment technology of poultry breeding pollution prevention projects[R]. 2012. |
| [46] |
ZOU Y, TU W, WANG H, et al. Anaerobic digestion reduces extracellular antibiotic resistance genes in waste activated sludge: The effects of temperature and degradation mechanisms[J]. Environment International, 2020, 143: 105980. DOI:10.1016/j.envint.2020.105980 |
| [47] |
TIAN Z, CHI Y, YU B, et al. Thermophilic anaerobic digestion reduces ARGs in excess sludge even under high oxytetracycline concentrations[J]. Chemosphere, 2019, 222: 305-313. DOI:10.1016/j.chemosphere.2019.01.139 |
| [48] |
SYAFIUDDIN A, BOOPATHY R. Role of anaerobic sludge digestion in handling antibiotic resistant bacteria and antibiotic resistance genes: A review[J]. Bioresource Technology, 2021, 330: 124970. DOI:10.1016/j.biortech.2021.124970 |
| [49] |
CONGILOSI J L, AGA D S. Review on the fate of antimicrobials, antimicrobial resistance genes, and other micropollutants in manure during enhanced anaerobic digestion and composting[J]. Journal of Hazardous Materials, 2021, 405: 123634. DOI:10.1016/j.jhazmat.2020.123634 |
| [50] |
ZHANG J Y, WANG Z Y, WANG Y Y, et al. Effects of graphene oxide on the performance, microbial community dynamics and antibiotic resistance genes reduction during anaerobic digestion of swine manure[J]. Bioresource Technology, 2017, 245: 850-859. DOI:10.1016/j.biortech.2017.08.217 |
| [51] |
LU T D, ZHANG J Y, LI P, et al. Enhancement of methane production and antibiotic resistance genes reduction by ferrous chloride during anaerobic digestion of swine manure[J]. Bioresource Technology, 2020, 298: 122519. DOI:10.1016/j.biortech.2019.122519 |
| [52] |
ZHANG J Y, SUI Q W, ZHONG H, et al. Impacts of zero valent iron, natural zeolite and Dnase on the fate of antibiotic resistance genes during thermophilic and mesophilic anaerobic digestion of swine manure[J]. Bioresource Technology, 2018, 258: 135-141. DOI:10.1016/j.biortech.2018.03.005 |
| [53] |
BENERAGAMA N, IWASAKI M, LATEEF S A, et al. Survival of multidrug-resistant bacteria in thermophilic and mesophilic anaerobic co-digestion of dairy manure and waste milk[J]. Animal Science Journal, 2013, 84(5): 426-433. DOI:10.1111/asj.12017 |
| [54] |
LATEEF S A, BENERAGAMA N, YAMASHIRO T, et al. Biohydrogen production from co-digestion of cow manure and waste milk under thermophilic temperature[J]. Bioresource Technology, 2012, 110: 251-257. DOI:10.1016/j.biortech.2012.01.102 |
| [55] |
SUN W, QIAN X, GU J, et al. Mechanisms and effects of arsanilic acid on antibiotic resistance genes and microbial communities during pig manure digestion[J]. Bioresource Technology, 2017, 234: 217-223. DOI:10.1016/j.biortech.2017.03.025 |
| [56] |
SUI Q W, ZHANG J Y, CHEN M X, et al. Distribution of antibiotic resistance genes(ARGs)in anaerobic digestion and land application of swine wastewater[J]. Environmental Pollution, 2016, 213: 751-759. DOI:10.1016/j.envpol.2016.03.038 |
| [57] |
SUI Q W, CHEN Y L, YU D W, et al. Fates of intracellular and extracellular antibiotic resistance genes and microbial community structures in typical swine wastewater treatment processes[J]. Environment International, 2019, 133: 105183. DOI:10.1016/j.envint.2019.105183 |
| [58] |
SHI Z, ZHAO R, WAN J, et al. Metagenomic analysis reveals the fate of antibiotic resistance genes in two-stage and one-stage anaerobic digestion of waste activated sludge[J]. Journal of Hazardous Materials, 2021, 406: 124595. DOI:10.1016/j.jhazmat.2020.124595 |
| [59] |
ZHANG J Y, LIU J B, WANG Y Y, et al. Profiles and drivers of antibiotic resistance genes distribution in one-stage and two-stage sludge anaerobic digestion based on microwave-H2O2 pretreatment[J]. Bioresource Technology, 2017, 241: 573-581. DOI:10.1016/j.biortech.2017.05.157 |
| [60] |
ZHANG J Y, LIU J B, LU T D, et al. Fate of antibiotic resistance genes during anaerobic digestion of sewage sludge: Role of solids retention times in different configurations[J]. Bioresource Technology, 2019, 274: 488-495. DOI:10.1016/j.biortech.2018.12.008 |
| [61] |
SUI Q W, MENG X S, WANG R, et al. Effects of endogenous inhibitors on the evolution of antibiotic resistance genes during high solid anaerobic digestion of swine manure[J]. Bioresource Technology, 2018, 270: 328-336. DOI:10.1016/j.biortech.2018.09.043 |
| [62] |
YOUNGQUIST C P, MITCHELL S M, COGGER C G. Fate of antibiotics and antibiotic resistance during digestion and composting: A review[J]. Journal of Environment Quality, 2016, 45(2): 537-545. DOI:10.2134/jeq2015.05.0256 |
| [63] |
MA Y, WILSON C A, NOVAK J T, et al. Effect of various sludge digestion conditions on sulfonamide, macrolide, and tetracycline resistance genes and class I integrons[J]. Environmental Science & Technology, 2011, 45(18): 7855-7861. |
| [64] |
ZHANG J Y, WANG Z Y, LU T D, et al. Response and mechanisms of the performance and fate of antibiotic resistance genes to nanomagnetite during anaerobic digestion of swine manure[J]. Journal of Hazardous Materials, 2019, 366: 192-201. DOI:10.1016/j.jhazmat.2018.11.106 |
| [65] |
ZHANG K, GU J, WANG X, et al. Analysis for microbial denitrification and antibiotic resistance during anaerobic digestion of cattle manure containing antibiotic[J]. Bioresource Technology, 2019, 291: 121803. DOI:10.1016/j.biortech.2019.121803 |
| [66] |
ZHANG J Y, LU T D, WANG Z Y, et al. Effects of magnetite on anaerobic digestion of swine manure: attention to methane production and fate of antibiotic resistance genes[J]. Bioresource Technology, 2019, 291: 121847. DOI:10.1016/j.biortech.2019.121847 |
| [67] |
WANG R, CHEN M X, FENG F, et al. Effects of chlortetracycline and copper on tetracyclines and copper resistance genes and microbial community during swine manure anaerobic digestion[J]. Bioresource Technology, 2017, 238: 57-69. DOI:10.1016/j.biortech.2017.03.134 |
| [68] |
MA J, GU J, WANG X, et al. Effects of nano-zerovalent iron on antibiotic resistance genes during the anaerobic digestion of cattle manure[J]. Bioresource Technology, 2019, 289: 121688. DOI:10.1016/j.biortech.2019.121688 |
| [69] |
XU R, ZHANG Y, XIONG W, et al. Metagenomic approach reveals the fate of antibiotic resistance genes in a temperature-raising anaerobic digester treating municipal sewage sludge[J]. Journal of Cleaner Production, 2020, 277: 123504. DOI:10.1016/j.jclepro.2020.123504 |
| [70] |
DAVIES K, GOATLEY L C, GUINAT C, et al. Survival of African swine fever virus in excretions from pigs experimentally infected with the Georgia 2007/1 isolate[J]. Transboundary and Emerging Diseases, 2017, 64(2): 425-431. DOI:10.1111/tbed.12381 |
| [71] |
WANG G, LI G, CHANG J, et al. Enrichment of antibiotic resistance genes after sheep manure aerobic heap composting[J]. Bioresource Technology, 2021, 323: 124620. DOI:10.1016/j.biortech.2020.124620 |
| [72] |
ZHOU G, QIU X, WU X, et al. Horizontal gene transfer is a key determinant of antibiotic resistance genes profiles during chicken manure composting with the addition of biochar and zeolite[J]. Journal of Hazardous Materials, 2021, 408: 124883. DOI:10.1016/j.jhazmat.2020.124883 |
| [73] |
LU X M, LU P Z. Synergistic effects of key parameters on the fate of antibiotic resistance genes during swine manure composting[J]. Environmental Pollution, 2019, 252: 1277-1287. DOI:10.1016/j.envpol.2019.06.073 |
| [74] |
LIU Y, CHENG D, XUE J, et al. Changes in microbial community structure during pig manure composting and its relationship to the fate of antibiotics and antibiotic resistance genes[J]. Journal of Hazardous Materials, 2020, 389: 122082. DOI:10.1016/j.jhazmat.2020.122082 |
| [75] |
ZHANG L, LI L, SHA G, et al. Aerobic composting as an effective cow manure management strategy for reducing the dissemination of antibiotic resistance genes: An integrated meta-omics study[J]. Journal of Hazardous Materials, 2020, 386: 121895. DOI:10.1016/j.jhazmat.2019.121895 |
| [76] |
XIA X, WANG Z, FU Y, et al. Association of colistin residues and manure treatment with the abundance of mcr-1 gene in swine feedlots[J]. Environment International, 2019, 127: 361-370. DOI:10.1016/j.envint.2019.03.061 |
| [77] |
GUO H, GU J, WANG X, et al. Responses of antibiotic and heavy metal resistance genes to bamboo charcoal and bamboo vinegar during aerobic composting[J]. Environmental Pollution, 2019, 252: 1097-1105. DOI:10.1016/j.envpol.2019.05.014 |
| [78] |
LIAO H, LU X, RENSING C, et al. Hyperthermophilic composting accelerates the removal of antibiotic resistance genes and mobile genetic elements in sewage sludge[J]. Environmental Science & Technology, 2018, 52(1): 266-276. |
| [79] |
WANG C, DONG D, STRONG P J, et al. Microbial phylogeny determines transcriptional response of resistome to dynamic composting processes[J]. Microbiome, 2017, 5: 103. DOI:10.1186/s40168-017-0324-0 |
| [80] |
DOAN T T, BOUVIER C, BETTAREL Y, et al. Influence of buffalo manure, compost, vermicompost and biochar amendments on bacterial and viral communities in soil and adjacent aquatic systems[J]. Applied Soil Ecology, 2014, 73: 78-86. DOI:10.1016/j.apsoil.2013.08.016 |
| [81] |
JEONG K H, LEE D J, LEE D H, et al. Composting reduces the vitality of H9N2 in poultry manure and EMCV in pig manure allowing for an environmentally friendly use of these animal wastes: A preliminary study[J]. Microorganisms, 2020, 8(6): 1-8. |
| [82] |
CHENG J H, TANG X Y, CUI J F. Effect of long-term manure slurry application on the occurrence of antibiotic resistance genes in arable purple soil(entisol)[J]. Science of the Total Environment, 2019, 647: 853-861. DOI:10.1016/j.scitotenv.2018.08.028 |
| [83] |
JOY S R, BARTELT-HUNT S L, SNOW D D, et al. Fate and transport of antimicrobials and antimicrobial resistance genes in soil and runoff following land application of swine manure slurry[J]. Environmental Science & Technology, 2013, 47(21): 12081-12088. |
| [84] |
CHEN Q L, AN X L, LI H, et al. Do manure-borne or indigenous soil microorganisms influence the spread of antibiotic resistance genes in manured soil?[J]. Soil Biology and Biochemistry, 2017, 114: 229-237. DOI:10.1016/j.soilbio.2017.07.022 |
| [85] |
PU Q, ZHAO L X, LI Y T, et al. Manure fertilization increase antibiotic resistance in soils from typical greenhouse vegetable production bases, China[J]. Journal of Hazardous Materials, 2020, 391: 122267. DOI:10.1016/j.jhazmat.2020.122267 |
| [86] |
UDIKOVIC-KOLIC N, WICHMANN F, BRODERICK N A, et al. Bloom of resident antibiotic-resistant bacteria in soil following manure fertilization[J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(42): 15202-15207. DOI:10.1073/pnas.1409836111 |
| [87] |
BOLSTER C H, BROOKS J P, COOK K L. Effect of manure application rate and rainfall timing on the leaching of antibiotic-resistant bacteria and their associated genes[J]. Water, Air, and Soil Pollution, 2018, 229(4): 130. DOI:10.1007/s11270-018-3781-6 |
| [88] |
SUI Q W, ZHANG J Y, CHEN M X, et al. Fate of microbial pollutants and evolution of antibiotic resistance in three types of soil amended with swine slurry[J]. Environmental Pollution, 2019, 245: 353-362. DOI:10.1016/j.envpol.2018.11.003 |
| [89] |
HE L Y, HE L K, GAO F Z, et al. Dissipation of antibiotic resistance genes in manure-amended agricultural soil[J]. Science of the Total Environment, 2021, 787: 147582. DOI:10.1016/j.scitotenv.2021.147582 |
| [90] |
RAUSEO J, BARRA CARACCIOLO A, ADEMOLLO N, et al. Dissipation of the antibiotic sulfamethoxazole in a soil amended with anaerobically digested cattle manure[J]. Journal of Hazardous Materials, 2019, 378: 120769. DOI:10.1016/j.jhazmat.2019.120769 |
| [91] |
XIE W Y, YUAN S T, XU M G, et al. Long-term effects of manure and chemical fertilizers on soil antibiotic resistome[J]. Soil Biology and Biochemistry, 2018, 122: 111-119. DOI:10.1016/j.soilbio.2018.04.009 |
| [92] |
LIN H, SUN W, ZHANG Z, et al. Effects of manure and mineral fertilization strategies on soil antibiotic resistance gene levels and microbial community in a paddy – upland rotation system[J]. Environmental Pollution, 2016, 211: 332-337. DOI:10.1016/j.envpol.2016.01.007 |
| [93] |
PU C, YU Y, DIAO J, et al. Exploring the persistence and spreading of antibiotic resistance from manure to biocompost, soils and vegetables[J]. Science of the Total Environment, 2019, 688: 262-269. DOI:10.1016/j.scitotenv.2019.06.081 |
| [94] |
HEUER H, FOCKS A, LAMSHÖFT M, et al. Fate of sulfadiazine administered to pigs and its quantitative effect on the dynamics of bacterial resistance genes in manure and manured soil[J]. Soil Biology and Biochemistry, 2008, 40(7): 1892-1900. DOI:10.1016/j.soilbio.2008.03.014 |
| [95] |
MUNIR M, XAGORARAKI I. Levels of antibiotic resistance genes in manure, biosolids, and fertilized soil[J]. Journal of Environmental Quality, 2011, 40(1): 248-255. DOI:10.2134/jeq2010.0209 |
| [96] |
ZHANG J Y, SUI Q W, TONG J, et al. Soil types influence the fate of antibiotic-resistant bacteria and antibiotic resistance genes following the land application of sludge composts[J]. Environment International, 2018, 118: 34-43. DOI:10.1016/j.envint.2018.05.029 |
| [97] |
WANG L, WANG J, WANG J, et al. Soil types influence the characteristic of antibiotic resistance genes in greenhouse soil with longterm manure application[J]. Journal of Hazardous Materials, 2020, 392: 122334. DOI:10.1016/j.jhazmat.2020.122334 |
| [98] |
CARLSON J, FISCHER M, ZANI L, et al. Stability of African swine fever virus in soil and options to mitigate the potential transmission risk[J]. Pathogens, 2020, 9(11): 1-12. |
| [99] |
BAKER-AUSTIN C, WRIGHT M S, STEPANAUSKAS R, et al. Coselection of antibiotic and metal resistance[J]. Trends in Microbiology, 2006, 14(4): 176-182. DOI:10.1016/j.tim.2006.02.006 |
| [100] |
LU T D, ZHANG J Y, WEI Y S, et al. Effects of ferric oxide on the microbial community and functioning during anaerobic digestion of swine manure[J]. Bioresource Technology, 2019, 287: 121393. DOI:10.1016/j.biortech.2019.121393 |
| [101] |
WU X, TIAN Z, LV Z, et al. Effects of copper salts on performance, antibiotic resistance genes, and microbial community during thermophilic anaerobic digestion of swine manure[J]. Bioresource Technology, 2020, 300: 122728. DOI:10.1016/j.biortech.2019.122728 |
| [102] |
PENG S, LI H, SONG D, et al. Influence of zeolite and superphosphate as additives on antibiotic resistance genes and bacterial communities during factory-scale chicken manure composting[J]. Bioresource Technology, 2018, 263: 393-401. DOI:10.1016/j.biortech.2018.04.107 |
| [103] |
ZHANG J Y, CHEN M X, SUI Q W, et al. Impacts of addition of natural zeolite or a nitrification inhibitor on antibiotic resistance genes during sludge composting[J]. Water Research, 2016, 91: 339-349. DOI:10.1016/j.watres.2016.01.010 |
| [104] |
SHEN Y, CHU L, ZHUAN R, et al. Degradation of antibiotics and antibiotic resistance genes in fermentation residues by ionizing radiation: A new insight into a sustainable management of antibiotic fermentative residuals[J]. Journal of Environmental Management, 2019, 232: 171-178. |
| [105] |
YU P, MATHIEU J, LU G W, et al. Control of antibiotic-resistant bacteria in activated sludge using polyvalent phages in conjunction with a production host[J]. Environmental Science & Technology Letters, 2017, 4(4): 137-142. |
| [106] |
伍亚云, 黄勋. 噬菌体治疗细菌感染的研究进展[J]. 中国感染控制杂志, 2021, 20(2): 186-190. WU Y Y, HUANG X. Advances in the treatment of bacterial infections by phages[J]. Chinese Journal of Infection Control, 2021, 20(2): 186-190. |
| [107] |
HYMAN P. Phages for phage therapy: Isolation, characterization, and host range breadth[J]. Pharmaceuticals, 2019, 12(1): 35. DOI:10.3390/ph12010035 |
| [108] |
MATHIEU J, YU P, ZUO P, et al. Going viral: Emerging opportunities for phage-based bacterial control in water treatment and reuse[J]. Accounts of Chemical Research, 2019, 52(4): 849-857. DOI:10.1021/acs.accounts.8b00576 |
| [109] |
LARRAÑAGA O, BROWN-JAQUE M, QUIRÓS P, et al. Phage particles harboring antibiotic resistance genes in fresh-cut vegetables and agricultural soil[J]. Environment International, 2018, 115: 133-141. DOI:10.1016/j.envint.2018.03.019 |
| [110] |
CALERO-CÁCERES W, YE M, BALCÁZAR J L. Bacteriophages as environmental reservoirs of antibiotic resistance[J]. Trends in Microbiology, 2019, 27(7): 570-577. DOI:10.1016/j.tim.2019.02.008 |
| [111] |
熊文斌, 卢晗, 刘新春. 噬菌体应用于污水处理过程的探讨[J]. 净水技术, 2020, 39(11): 93-100. XIONG W B, LU H, LIU X C. Exploration of bacteriophage applied in process of wastewater treatment[J]. Water Purification Technology, 2020, 39(11): 93-100. |
 2021, Vol. 40
2021, Vol. 40


