2. 中国环境科学研究院环境基准与风险评估国家重点实验室, 北京 100012;
3. 兰州交通大学环境与市政工程学院, 兰州 730070
2. State Key Laboratory of Environmental Criteria and Risk Assessment, Chinese Research Academy of Environmental Sciences, Beijing 100012, China;
3. School of Environmental and Municipal Engineering, Lanzhou Jiaotong University, Lanzhou 730070, China
抗生素广泛存在于环境中,具有持久性,能抑制甚至杀灭多种病原菌[1]。氟喹诺酮类抗生素(Fluoroquinolones,FQs)是一类具有较广抗菌谱、高疗效的人工合成抗菌药物,长期应用于人类医学和兽用防治领域[2]。中国是世界上最大的抗生素生产国和使用国,每年动物的使用量达84 240 t。据统计估算,2013年畜禽抗生素的使用量占抗生素使用总量的52%,略高于人类使用量(48%)。在畜禽养殖中FQs使用量较多的是恩诺沙星(ENR)、环丙沙星(CIP)和诺氟沙星(NOR)[3-4]。大多数抗生素不能被动物完全代谢,其摄入剂量的30%~90%以原型药物和代谢产物形式排出[5-6]。因此,畜禽排泄物中存在大量的FQs,它们通过土壤粪肥、回用灌溉水等途径进入自然环境[7-8],进而对生态环境和人类健康产生影响。畜禽粪便和粪便堆肥产品常作为肥料使用,是抗生素进入农业领域的主要来源,抗生素在施用粪肥的农田中经雨水径流形成扩散污染[9]。施用粪肥后,农业土壤中FQs的残留量较高[10],除此之外,污泥、沉积物、水环境中也有不同程度的残留[11-12]。畜禽废物进入自然环境后造成一定水平的FQs污染,但首当其冲的是土壤环境中的FQs污染问题,近年来,畜禽粪便中的FQs残留及其进入土壤后的降解是环境科学领域的研究热点。有研究表明:FQs生物降解速率缓慢,半衰期较长(> 100 h)[13],在环境中残留时间比磺胺类和大环内酯类抗生素更长[14];土壤颗粒存在吸附作用,该作用可螯合FQs,延缓了FQs的非生物、生物降解,使得FQs在土壤和水生沉积物中存在更持久[15]。为解决环境中的FQs污染问题,国内外学者对FQs的有效去除方法进行了大量研究,FQs可经吸附、水解、光解、生物降解等一系列非生物或生物过程进行转化[16]。其中,吸附、水解、光解等非生物降解途径可降低抗生素污染物的污染水平,但这些降解作用不足以使FQs残留降低至安全浓度。FQs的生物转化是自然环境中抗生素类药物降解的重要途径,也是畜禽粪便中FQs残留物失活的主要形式[17],因此,受到国内外学者的广泛关注。目前,畜禽废物中FQs的生物降解方法主要有两种:高温堆肥和外源添加高效降解菌剂。基于文献研究,本文对畜禽养殖环境中FQs的残留情况进行资料收集,结合国内外FQs生物降解的研究现状,现阶段学术界关于FQs生物降解效果的研究已有成果性的进展,国外关于FQs生物转化机理及分子机制的深入研究,则为科学解决FQs残留污染问题提供了理论基础。文中对FQs的生物转化机制与机理进行重点阐述,并对今后FQs生物去除方法研究进行展望,以期为畜禽粪便中FQs生物转化的进一步研究提供参考。
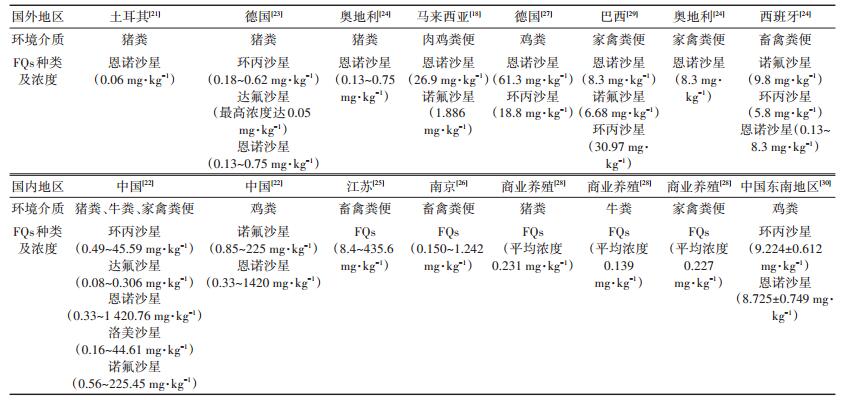
1 畜禽粪便中FQs的残留与降解 1.1 畜禽粪便中FQs的残留根据国内外各畜牧养殖场和农场的检测调查表明,在猪、鸡和牛的粪便中,氟喹诺酮抗生素(FQs)的检出率高,检出浓度范围广。诺氟沙星(NOR)、环丙沙星(CIP)、恩诺沙星(ENR)、氧氟沙星(OF)、洛美沙星(LOM)和麻保沙星(MAR)是畜禽粪便中检出率和检出浓度较高的几类FQs[17-20];而环丙沙星、恩诺沙星和诺氟沙星的残留浓度极高,是FQs降解去除中的主要研究对象。表 1列出了国内外不同畜禽养殖场的畜禽粪便中FQs的污染水平。总体而言,国外各养殖场及农场畜禽粪便中常检测到的几类FQs残留浓度低于国内;FQs在畜禽粪便中的残留浓度普遍较高。然而,调查数据中显示的FQs残留水平要远高于实验研究中的浓度值,并影响到FQs生物去除方法在外界环境中的实际应用,这一问题将是今后研究中的重要突破点。
|
|
表 1 国内外畜禽粪便中检测到的FQs种类及浓度 Table 1 Types and concentrations of FQs detected in animal manure in China and other countries |
国内外调查表明,畜禽粪便中FQs残留污染现象较为普遍,FQs残留浓度因地区和时节差异存在明显的不同。Hu等[31]检测出畜禽粪便中环丙沙星、氧氟沙星等抗生素的残留浓度会随时节而变化,夏季与冬季之间的浓度差异较明显。中国北方地区畜禽粪便中环丙沙星平均残留浓度(0.808±2.308 mg·kg-1),比东南地区畜禽粪便中环丙沙星浓度(45.6 mg·kg-1)要低[22]。Huang等[32]在施用猪粪的农田土壤中检测到恩诺沙星等抗生素的残留水平在0.189~2.669 mg·kg-1之间。刘峰等[33]调查发现,福建厦门市和莆田市部分养殖场的畜禽粪便中喹诺酮类抗生素(Quinolones,QNs)的检出浓度很高,单个样品总含量∑QNs在粪肥中最高达到2.968 mg·kg-1。在福建两市的作物土壤中检测发现,FQs浓度最高达到0.579 mg·kg-1,其中50%的样品FQs总浓度超过生态毒性效应浓度0.100 mg·kg-1,具有潜在的生态风险[33]。Hou等[34]对中国北方辽宁、天津等地区施用畜禽粪便的土壤进行检测,FQs的残留浓度为0.411±1.453 mg·kg-1。
值得关注的是,畜禽粪便中的FQs进入土壤后,在土壤介质中会发生一系列的生物作用和非生物作用,但土壤自身的降解能力较弱,使得土壤中FQs残留仍较高,为控制FQs残留所带来的污染,寻找FQs高效生物降解去除方法将需进行大量的实验研究。
1.2 畜禽粪便中FQs的降解环境中FQs的归趋行为包括:吸附、降解和植物吸收与富集。关于FQs的降解研究,有FQs的水解、光降解以及生物降解。生物降解作为一种有应用前景的FQs高效去除方法,主要有两种方式:高温堆肥和外源添加降解菌剂。这两种方法不仅能有效去除FQs,且在实际田间环境中有较好的环境效应,能很好地防止畜禽粪便中的FQs通过食物链等途径进入生态环境后引发风险问题,成为了近年来FQs生物降解研究领域的重点。
堆肥是一种畜禽粪便高效资源化的处置方法,能使土壤环境中FQs浓度显著降低[35-36]。季秋洁[20]在猪粪堆肥实验中,发现55 ℃的光照条件下,C:N为30:1时,恩诺沙星、环丙沙星、诺氟沙星的降解最快,半衰期分别为7.34、8.62、5.89 d。Ho等[18]采集大型肉鸡农场中的粪便为原料,并向其中加入标准样品恩诺沙星和诺氟沙星各5 mg·kg-1,检测出粪肥中恩诺沙星和诺氟沙星的初始浓度分别为5.09 mg·kg-1和36.77 mg· kg-1,堆肥降解40 d中,99.93%的恩诺沙星在13 d之内被降解,诺氟沙星在第9 d被快速降解,18 d以后浓度低于检出限,两类氟喹诺酮类抗生素的降解率均在99.9%以上。FQs的堆肥去除分为好氧和厌氧两种方式,好氧发酵是一种可以消减畜禽粪便中抗生素残留污染的有效途径,环丙沙星在高温好氧发酵过程中可被降解至检出限以下[37]。
进一步研究发现,在FQs的堆肥处理过程中,与添加外源菌剂相结合的生物降解方法更能有效提高FQs的去除率。冯瑶[38]以鸡粪为堆肥原料,在堆体初始C:N分别为10、15、20、25、35,以及添加不同外源抗生素降解功能菌剂(细菌T4和真菌FZC3)的条件下,研究抗生素诺氟沙星的降解情况。结果表明,C: N为25,诺氟沙星的添加量为50 mg·kg-1时,诺氟沙星去除率最高,可达80.78%;与未添加菌剂处理相比,添加了实验室筛选分离得到的两株外源菌剂后,诺氟沙星的去除率提高了0.61%~2.50%。总体而言,该研究中外源菌剂的添加对诺氟沙星的去除效果不够明显,还需对外源菌剂在不同温度下的活性及其对不同浓度诺氟沙星的去除率做进一步研究。薛南冬[39]以鸡粪为堆肥原料,与锯末按照质量比为6~8:1的比例进行混合,测出C:N为20:1,堆肥原料含水率在55%~60%之间,堆肥室温控制在25~30 ℃,并外源添加耐高温菌种产黄纤维单胞菌的条件下,外源菌剂与其他相关微生物共同作用,诺氟沙星、环丙沙星、沙拉沙星、洛美沙星、恩诺沙星的降解率提高至61.09~ 82.82%,使粪肥中的FQs降低至安全量。就外界环境中FQs的污染水平,Slana等[40]研究比较了实验室条件和田间条件下恩诺沙星的降解途径和速率,相比于受控的实验室条件,田间环境条件下恩诺沙星的降解模式多样化,降解速度较快,经两个月的田间试验培养,大约66%的恩诺沙星和92%的环丙沙星被降解。
目前,国内外学者了解到FQs的降解效果除了受降解菌种类、丰度等生物因素的控制外,受pH值、温度、总有机碳(TOC)、总氮(TN)、总磷(TP)和金属含量等非生物因素的控制也较大。关注环境因子的调控,抗生素在堆肥和外源添加降解菌的作用下可有较好的生物去除效果,为有效降低FQs生物降解过程中降解菌带来的生物风险,应加强FQs生物转化机理与机制的认知。
2 畜禽粪便中FQs的生物转化微生物的生物转化作用是降解去除FQs抗生素的有效途径。抗生素的生物转化是指在微生物降解菌的作用下,将抗生素降解转化为其他多种降解产物的过程。低效降解菌作用下,抗生素生物转化后的降解残留物及降解产物仍存在抗菌活性,在环境中有潜在的生态风险。近年来,如何发掘筛分出稳定高效的外源降解菌以提高生物转化作用成为了FQs降解研究的重点,为此众多学者开展了不同外源降解菌剂作用下,FQs生物转化过程中的降解条件、降解产物、降解途径等机理研究,并进一步分析了抗生素及其降解产物的抗菌活性。
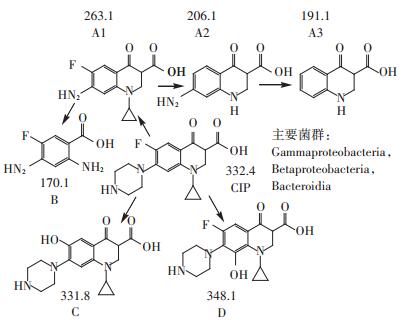
Selvam等[41]研究了在猪粪堆肥物料中,较高浓度(20 mg·kg-1)下环丙沙星降解率为82.9%,低浓度(2 mg·kg-1)下降解率仅为69%,该结果表明,实验中控制了堆肥温度、碳氮比、总有机碳、总磷等物理化学性质,金霉素和磺胺嘧啶可得到高效降解,但堆肥后环丙沙星仍存在17%~31%的残留,抗生素残留物通过牲畜粪便堆肥进入生态系统后会产生潜在风险。因此,需进一步提高环丙沙星抗生素的去除率。而Liao等[42]实验研究发现温度为25 ℃时,添加葡萄糖为碳源,氨氮为氮源,以变形菌纲(Gammaproteobacteria)、拟杆菌纲(Bacteroidia)和β-变形菌纲(Betaproteobacteria)为主要驯化降解细菌群落,28 d后100 μg·L-1环丙沙星的降解率高达89.1%,可见,高效外源菌剂在环丙沙星生物降解中发挥着极其重要的作用,该降解转化过程中环丙沙星的四种降解途径、降解产物及主要降解菌群如图 1所示。
|
图 1 环丙沙星的四种降解途径及其代表性菌群 Figure 1 Four degradation pathways and typical bacteria flora of ciprofloxacin |
Pan等[12]从制药污泥中筛分出嗜热细菌菌株C419,并在实验中添加乙酸钠,以70 ℃、pH 6.5为最佳条件,有效促进了降解菌株的生长,其中环丙沙星、诺氟沙星、恩诺沙星、氧氟沙星被高效降解,4种FQs的残留抗菌活性降低了20%~40%;实验同时进一步研究了环丙沙星抗生素的生物降解过程,经5 d的降解去除,环丙沙星的降解率达57%,并检测出7种代谢产物,降解产物的残留抗菌活性也明显降低。故C419菌株有望成为受氟喹诺酮类污染的畜禽粪便堆肥环境或其他环境中的优势修复菌种。有研究表明[43],埃希氏菌、假单胞菌和微杆菌通过特异性位点的羟基化等反应可较快地降解FQs。其中诺氟沙星经微杆菌属(Microbacterium sp.)转化生成四种代谢物:N-乙酰基诺氟沙星、去乙烯N-乙酰基诺氟沙星、8-羟基诺氟沙星和脱氟化合物6-羟基诺氟沙星[44]。
除细菌外,学者们研究发现多种真菌可用于降解FQs[45-47]。子囊菌(Xylaria longipes)作为一种能有效降解氟喹诺酮类药物的真菌,能高效降解恩诺沙星和达氟沙星,并生成降解产物恩诺沙星N-氧化物和达氟沙星N-氧化物。FQs及相应降解产物的残留抗菌活性显著降低,其中浓度3.5 μg·L-1的恩诺沙星和恩诺沙星N-氧化物的残留抗菌活性分别低于10%和25%[48],达氟沙星N-氧化物的残余抗菌活性小于母体化合物的20%[47]。
对环境有机物有较强降解能力的另一类真菌为白腐菌,其在FQs生物降解中具有较好应用[49-50]。Čvančarová等[45]研究发现白腐菌群中的白囊耙齿菌(Irpex lacteus)和变色栓菌(Trametes versicolor)对诺氟沙星、氧氟沙星和环丙沙星有很高的降解潜力,残留抗生素活性测试的结果显示,白囊耙齿菌能完全去除氧氟沙星和诺氟沙星,并未检测出它们的残留抗菌活性,而环丙沙星虽在10 d内完全降解,14 d后仍有一定的残留抗菌活性。实验表明,降解菌作用下,FQs的生物转化并非完全去除了抗生素及其降解产物的抗菌活性。
在土壤真菌和细菌作用下,环丙沙星和诺氟沙星的主要生物转化途径有亚硝化和乙酰化[51]。较早时期,就有研究发现黄孢原毛平革菌(Phanerochaete chrysosporium)、拟盘多毛孢属(Pestalotiopsis guepini)、条纹黏褶菌(Gloeophyllum striatum)这几类真菌对环丙沙星和诺氟沙星有生物降解作用[51-54],但这几类菌对FQs降解效果不显著,因此,并未对其展开长期研究。文献所述表明,FQs生物转化过程中,受不同降解菌和降解条件的影响,同种抗生素的降解途径和降解产物存在显著差异[55-56]。
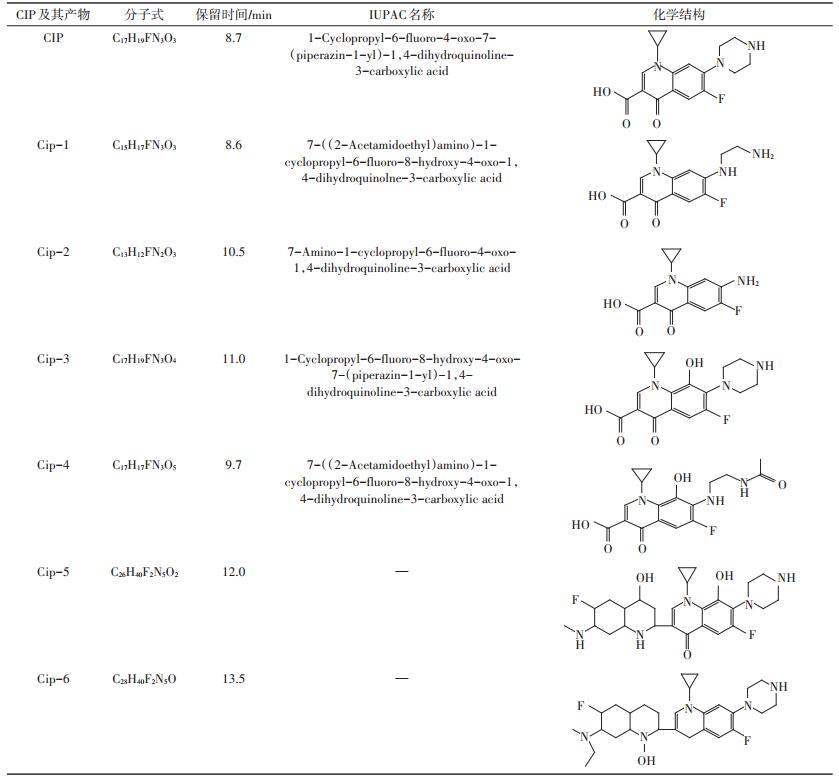
近年来,国外学者对白腐真菌[57]中的变色栓菌分泌漆酶参与CIP和NOR的降解研究关注较多,其主要通过三条途径形成转化产物:(1)哌嗪基取代基的氧化,(2)单羟基化,(3)二聚产物的形成。在不同的培养时期分别形成6种环丙沙星转化产物和3种恩诺沙星转化产物,环丙沙星及其转化产物的分子式、IUPAC命名、化学结构及保留时间见表 2。
|
|
表 2 环丙沙星及其转化产物的分子式、IUPAC命名、化学结构及保留时间 Table 2 Molecular formula, systematic IUPAC names, chemical structure and retention time of ciprofloxacin and its transformation products |
Čvančarová等[45]通过发光细菌毒性试验表明,随着CIP、OF和NOR存在浓度的增加,其对发光细菌的抑制作用不断增强。实验表明,FQs及相应代谢产物对发光细菌有一定毒性,并观察到生物体对CIP、OF更敏感,5类白腐真菌的降解作用下,生物毒性均显著降低。据报道[48],在不同细胞体系中,FQs呈现出不同的细胞毒性,超过毒性剂量会抑制细胞增殖;子囊菌(X. longipes)N-氧化不仅降低了抗微生物活性,而且能显著降低细胞毒性作用。在哺乳动物中,N-氧化物的形成是氟喹诺酮N-烷基化的主要代谢途径之一。可见,子囊菌的生物转化能有效灭活氟喹诺酮类药物。发光细菌毒性试验和细胞毒性测试是抗生素生物转化过程中生态风险评价的重要指标。研究表明,高效降解菌作用下,抗生素及转化产物降解去除过程中抗菌活性有效降低,随之生物风险显著降低。国外学者对与抗生素降解菌有关的生物风险问题虽有所关注,但相关研究尚少,日后需加大该方面的探究。
3 FQs生物转化的分子机制通过畜禽排泄物进入环境的氟喹诺酮类有机污染物,在微生物作用下得到了有效降解。明确FQs生物转化过程中的分子机制是实现抗生素及其抗性基因生物安全的关键。因此,抗生素降解过程中,微生物群落结构、丰度及真菌、细菌多样性变化对FQs的生物转化影响程度,降解菌分泌酶或所携带的酶对FQs的生物降解作用,以及起关键作用的抗性基因(Antibiotic resistance genes,ARGs)的含量变化情况及其分布与归趋,这几类问题是值得研究者们深入探讨的。
3.1 FQs生物转化中微生物群落结构与动态环境中抗生素有机污染物的有效生物衰减,通常与整个微生物群落的协同作用相关[58-59]。鸡粪堆肥过程中,诺氟沙星在50 mg·kg-1浓度下,堆肥高温期微生物群落的丰度增大,整个堆肥过程中的真菌多样性有所增加,细菌多样性变化不明显。其中与诺氟沙星降解有一定关系的细菌主要是芽孢杆菌属(Bacillus)、芽孢八叠球菌属(Sporosarcina)和拟杆菌属(Bacteroidetes),真菌包括酵母菌属(Issatchenkia)、曲霉属(Aspergillus)、节担菌属(Wallemia)以及一个未被辨识的菌属[38]。
抗生素生物转化中,起作用的微生物菌落生长为优势菌种和敏感菌种。环丙沙星的生物处理过程中,微生物群落有较大的结构变化,投加环丙沙星至300 μg·L-1高浓度时,硝化螺旋菌属(Nitrospirae)是敏感菌种,在CIP的作用下易被杀灭,而抗环丙沙星的微生物绿菌属(Chlorobi)和绿弯菌属(Chloroflexi)得以生存、适应和繁殖,逐渐成为生物膜优势菌群[60]。Liao等[42]研究发现环丙沙星生物降解过程中细菌群落结构有明显的变化,变形菌纲,拟杆菌纲和β-变形菌纲为环丙沙星降解的主要菌群;而假黄色单胞菌属(Pseudoxanthomonas)、寡养单胞菌(Stenotrophomonas)、苯基杆菌属(Phenylobacterium)和白色杆菌属(Leucobacter)参与环丙沙星的消散过程,仅为附属菌群。Meng等[61]进一步研究了微生物群落结构的变化情况,FQs生物降解中,微生物物种丰富度的急剧下降,在门和属两个级别上,细菌群落结构变化显著,两个主要门:变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)为FQs的优势菌群。
生物转化中FQs的微生物群落变化相关研究表明,FQs浓度和生长条件的不同,会对微生物群落结构和多样性产生极大影响[62];实验测试,单种或多种抗生素混合存在时,优势菌群在培养基上产生了适应性,微生物群落结构没有明显变化[63]。FQs存在时,微生物群落结构存在显著变化,但微生物多样性和丰度的长期变化还未有研究[64]。Brandt等[65]提出,微生物群落可能是一个抗生素敏感群体,在生态功能领域起着调节、供应和支撑作用。土壤中的抗生素会影响微生物群落结构和多样性,进而导致一些微生物种群及其生态功能消失[62]。FQs的微生物生态学知识有利于研究抗生素降解生物学过程,同时为环境中的微生物群落发挥更好的生态功能效应提供理论指导。
3.2 FQs生物转化中的酶作用在抗生素的微生物降解转化中,高降解能力的耐药菌含有消除大分子化合物中脆弱化学键的酶,从而降低了抗生素的活性。白腐真菌是一种能降解木质素的担子菌,并能分泌胞外木质素降解酶,它除了自身能降解诺氟沙星、氧氟沙星和环丙沙星,其木质素酶也能降解各种结构复杂、难降解的抗生素有机物。
现阶段,学者们对白腐真菌所携带的具有非特异性的酶系统已有较多关注,研究表明非特异性的酶系统作用可以转化甚至矿化应用最广泛的抗生素[66],该系统包括细胞外木质素降解酶(主要是漆酶,木质素过氧化物酶,锰过氧化物酶和通用的过氧化物酶)以及胞内酶如细胞色素P-450酶等,这些酶对抗生素有高效的降解能力[67]。有研究发现,爆竹柳和玉米两种植物对抗生素的降解由细胞色素P-450酶驱动,能发生羟基化等转化反应[68]。Čvančarová等[45]通过PCA分析木质素降解酶活性,分析表明锰过氧化物酶可能参与木质素降解菌对FQs的降解。近年来,白腐真菌全细胞和包埋在颗粒生物塑料制剂中的真菌繁殖体已经成功地应用于废水或污泥中抗生素药物的去除[69-70]。抗生素生物转化过程中,全细胞生物催化是指利用微生物有机体(即全细胞、组织甚至个体)作为催化剂进行化学转化的过程。Prieto等[57]研究了酸性条件下,使用白腐真菌的全细胞和该菌分泌酶能降解环丙沙星和诺氟沙星。为实现白腐真菌降解FQs生物技术的成功开发应用,研究并确定降解过程中起主要作用的酶是一关键点。
除此之外,Blanquez等[71]研究使用肉毒链霉菌(Streptomyces)碱性漆酶SiLA降解环丙沙星和诺氟沙星两类氟喹诺酮类抗生素。特定的SiLA-乙酰丁香酮体系能够降解超过90%的氟喹诺酮类,使用HPLC分析酶处理后的降解产物,结果证明24 h后抗菌剂完全消失,毒性测试表明该酶系统对诺氟沙星和环丙沙星的毒性分别降低了70%和90%。可见,FQs生物转化过程中,降解菌所携带的酶系统对降低抗生素毒性所发挥的作用是举足轻重的。
3.3 FQs生物转化中的ARGs为揭示抗性基因作用下抗生素耐药性的扩散分子机制,在分子生物学方面,学者们对FQs生物转化中ARGs相关作用做了大量研究。部分微生物对抗生素的降解需携带ARGs,即耐药菌才能发挥直接破坏或修饰抗生素等作用而使其失活[72]。Lucas等[73]研究检测出白腐真菌(Trametes versicolor)处理下,使对氟喹诺酮敏感性降低的抗性基因qnrS增加了163%,传统污水处理厂中抗性基因qnrS增加了302%,这一抗性基因在传统污水处理中对环丙沙星、恩诺沙星和麻保沙星这几类FQs的高效去除起关键作用[74]。
赵祥[75]研究了某地施用畜禽粪便后设施菜地土壤中的抗生素及ARGs含量,并检测发现检出率较高的喹诺酮类ARGs为qepA、qnrS、qnrB,其中qepA的含量相对较高,抗性基因qnr与重金属含量的相关性显著。生猪养殖废物中通常会有qnr(A、B和S)的存在[76-77]。Mu等[78]对中国北方家禽饲养场中的ARGs进行检测,在辽宁和天津地区的部分养殖场中,检测率较高的FQs抗性基因有qnrS和qepA。Zhang等[79]研究表明土壤中施用猪粪和家禽粪便可能会比施用牛粪产生更丰富的ARGs。在中国,畜禽粪便和堆肥后的粪便作为肥料的应用是农业土壤中抗生素的主要来源之一。更多研究表明[80-82],长期施用畜禽粪便堆肥,土壤中ARGs的多样性和丰度显著增加。
在畜禽废物的生物处理过程中,除了抗生素存在降解转化,抗性基因也会被消减[83]。好氧堆肥被广泛用于畜禽粪便的回收利用,并能有效降低进入环境中的ARGs含量。郑宁国等[84]研究了猪粪好氧高温堆肥过程中,两个喹诺酮类抗性基因qnrA和qnrS丰度的变化,堆肥过程中qnrS含量呈现急剧降低趋势,在堆肥结束时降至2.6×103 copies·g-1,去除率达到99.7%;而qnrA含量在堆肥过程中降解速率较为缓慢,堆肥结束时丰度为1.5×105 copies·g-1,去除率达到61.8%。Selvam等[85]研究探讨了猪粪堆肥中ARGs的变化,喹诺酮类抗性基因gyrA和parC在28~42 d的堆肥处理后,gyrA被完全去除,parC基因含量显著降低,但在持续56 d的检测中仍有部分残留。可见,堆肥是去除ARGs的有效方法,但基因宿主菌的嗜温性以及基因型较大等性质是少数抗性基因在堆肥过程中难以去除的潜在原因。
有研究表明,不同种类动物粪便中ARGs的多样性、丰富度和持久性有显著差异,并受重金属浓度、总氮水平以及接触抗生素的剂量和持续时间的影响[86]。生物用药过程中,抗生素、耐药菌和环境细菌的不断混合,使ARGs在环境中大量出现并广泛传播[87-88]。不同抗生素与ARGs在环境中具有不同的归趋和传播机制,抗生素的降解过程、可动遗传因子的类型(Mobile genetic elements,MGE)、微生物群落结构[89]以及环境介质理化性质(如总有机质、温度、营养元素等[90])均可影响他们之间的相关性,但不同抗生素与ARGs间的相关机制有待进一步研究。
研究表明,堆肥是去除ARGs的有效手段,为降低FQs抗性基因的污染水平,在土地施用粪肥之前,建议对动物粪便中抗生素进行适当的生物降解处理。现阶段,研究者们对FQs生物转化过程中起关键作用的ARGs有了广泛认知,并研究了该类抗性基因的含量变化情况。在今后的研究中,探究相关因素对ARGs分布和归趋机制的影响是解决ARGs污染问题的关键,这不仅能有效降低ARGs的多样性和丰度,也对减缓ARGs在环境中的传播有积极作用。
4 结论与展望近年来,畜牧养殖业迅猛发展,随之带来的畜禽粪便中抗生素污染问题不容轻视。调查数据显示,国内外畜禽粪便中FQs污染问题普遍严重,多数达到了生态毒性效应浓度阈值几倍以上,东南地区畜禽养殖场更是FQs污染的重灾区。国内畜禽粪便中FQs的残留浓度普遍高于实验室中FQs降解研究浓度水平,高浓度FQs污染条件下,高效生物处理方法的研究是一重要趋势。堆肥和外源添加高效降解菌剂作为处理畜禽粪便中FQs的常用手段,可稳定粪肥并有效降低抗生素浓度。基于最新文献所述,白腐真菌及其分泌酶对FQs和ARGs具有较高生物降解能力,且生物风险较低,相较于其他降解菌群具有更多优势。国外对FQs生物转化机理以及分子机制的认知已较广泛,但关于降解菌作用下,FQs生物转化过程中涉及人类健康和生态环境的生物风险问题的探究还不全面,以及ARGs在环境中的分布与归趋相关性研究尚有不足,生物转化机理与分子机制研究对抗生素及其抗性基因污染水平的有效降低具有重要意义,有必要加深关于机制机理研究的认知。
鉴于FQs生物降解研究现状,建议今后在FQs的生物去除方法上开展以下研究:
(1)FQs属光降解敏感型抗生素,应进行光解和生物降解相结合作用下FQs生物去除方法的研究,因土壤作为施用粪肥后残留FQs的直接受纳环境,可将降解菌与土壤中对FQs有较强吸收和富集能力的植物进行组合优化,以提高FQs的生物降解效果。
(2)由于多种外界环境因素在实验室条件下无法模拟,且实验室研究与环境中FQs的污染浓度水平不一致,畜禽粪便中FQs生物转化过程的去除研究不应仅停留在实验室水平,更应强调实际应用。
(3)生物转化过程中,受土著微生物、重金属、环境条件变化等因素影响,外源降解微生物的存活能力和降解活性会有所降低,从而可能导致外源降解菌对FQs的生物降解能力下降,为避免降解菌降解能力下降带来的生物风险,提高降解菌剂在各因素下的耐受性将成为FQs生物降解的研究重点。
| [1] |
Maia A S, Ribeiro A R, Amorim C L, et al. Degradation of fluoroquinolone antibiotics and identification of metabolites /transformation products by liquid chromatography-tandem mass spectrometry[J]. Journal of Chromatography, 2014, 1333: 87-98. DOI:10.1016/j.chroma.2014.01.069 |
| [2] |
Emmerson A M, Jones A M. The quinolones: Decades of development and use[J]. Journal of Antimicrobial Chemotherapy, 2003, 51: 13-20. |
| [3] |
GERMAP 2015-Antibiotika-Resistenz und-Verbrauch[EB/OL]. Bundesamt für Verbrauche-rschutz und Lebensmittelsicherheit. 2016. http://www.bvl.bund.de/SharedDocs/Downloads/05_Tierarzneimittel/germap2015.pdf?__blob=publicationFile&v=3
|
| [4] |
Zhang Q Q, Ying G G, Pan C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782. |
| [5] |
Zhang Y L, Lin S S, Dai C M, et al. Sorption-desorption and transport of trimethoprim and sulfonamide antibiotics in agricultural soil: Effect of soil type, dissolved organic matter, and pH[J]. Environmental Science and Pollution Research, 2014, 21(9): 5827-5835. DOI:10.1007/s11356-014-2493-8 |
| [6] |
Liu F, Ying G G, Yang J F, et al. Dissipation of sulfamethoxazole, trimethoprim and tylosin in a soil under aerobic and anoxic conditions[J]. Environmental Chemistry, 2010, 7(4): 370-376. |
| [7] |
Barton M D. Impact of antibiotic use in the swine industry[J]. Current Opinion in Microbiology, 2014, 19: 9-15. DOI:10.1016/j.mib.2014.05.017 |
| [8] |
Christou A, Aguera A, Bayona J M, et al. The potential implications of reclaimed wastewater reuse for irrigation on the agricultural environment: The knowns and unknowns of the fate of antibiotics and antibiotic resistant bacteria and resistance genes: A review[J]. Water Research, 2017, 123: 448-467. DOI:10.1016/j.watres.2017.07.004 |
| [9] |
Qiao M, Ying G G, Singer A C, et al. Review of antibiotic resistance in China and its environment[J]. Environment International, 2018, 110: 160-172. DOI:10.1016/j.envint.2017.10.016 |
| [10] |
Speltini A, Sturini M, Maraschi F, et al. Analytical methods for the determination of fluoroquinolones in solid environmental matrices[J]. Trac-Trends in Analytical Chemistry, 2011, 30(8): 1337-1350. DOI:10.1016/j.trac.2011.04.011 |
| [11] |
Almeida C M R, Santos F, Ferreira A C F, et al. Can veterinary antibiotics affect constructed wetlands performance during treatment of livestock wastewater?[J]. Ecological Engineering, 2017, 102: 583-588. DOI:10.1016/j.ecoleng.2017.02.035 |
| [12] |
Pan L J, Li J, Li C X, et al. Study of ciprofloxacin biodegradation by a Thermus sp. isolated from pharmaceutical sludge[J]. Journal of Hazardous Materials, 2018, 343: 59-67. DOI:10.1016/j.jhazmat.2017.09.009 |
| [13] |
Wang L, Qiang Z, Li Y, et al. An insight into the removal of fluoroquinolones in activated sludge process: Sorption and biodegradation characteristics[J]. Journal of Environmental Sciences, 2017, 56: 263-271. DOI:10.1016/j.jes.2016.10.006 |
| [14] |
Chee-Sanford J C, Mackie R I, Koike S, et al. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J]. Journal of Environmental Quality, 2009, 38(3): 1086-1108. |
| [15] |
Girardi C, Greve J, Lamshöft M, et al. Biodegradation of ciprofloxacin in water and soil and its effects on the microbial communities[J]. Journal of Hazardous Materials, 2011, 198: 22-30. DOI:10.1016/j.jhazmat.2011.10.004 |
| [16] |
Park J Y, Huwe B. Effect of pH and soil structure on transport of sulfonamide antibiotics in agricultural soils[J]. Environmental Pollution, 2016, 213: 561-570. DOI:10.1016/j.envpol.2016.01.089 |
| [17] |
Wohde M, Berkner S, Junker T, et al. Occurrence and transformation of veterinary pharmaceuticals and biocides in manure: A literature review[J]. Environmental Science Europe, 2016, 28(1): 23. |
| [18] |
Ho Y B, Zakaria M P, Latif P A, et al. Degradation of veterinary antibiotics and hormone during broiler manure composting[J]. Bioresource Technology, 2013, 131: 476-484. DOI:10.1016/j.biortech.2012.12.194 |
| [19] |
孟磊, 杨兵, 薛南冬, 等. 高温堆肥对鸡粪中氟喹诺酮类抗生素的去除[J]. 农业环境科学学报, 2015, 34(2): 377-383. MENG Lei, YANG Bing, XUE Nan-dong, et al. Effect of high temperature composting on removal of fluoroquinolones in chicken manures[J]. Journal of Agro-Environment Science, 2015, 34(2): 377-383. |
| [20] |
季秋洁. 3种氟喹诺酮类抗生素在猪粪中的残留与降解[D].合肥: 安徽农业大学, 2012. JI Qiu-jie. The residues and degradation of three fluoroquinolone antibiotics in pig feces[D]. Hefei: Anhui Agricultural University, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10364-1013163636.htm |
| [21] |
Karcı A, Balcıoğlu I A. Investigation of the tetracycline, sulfonamide, and fluoroquinolone antimicrobial compounds in animal manure and agricultural soils in Turkey[J]. Science of the Total Environment, 2009, 407(16): 4652-4664. DOI:10.1016/j.scitotenv.2009.04.047 |
| [22] |
Zhao L, Dong Y H, Wang H. Residues of veterinary antibiotics in manures from feedlot livestock in eight provinces of China[J]. Science of the Total Environment, 2010, 408(5): 1069-1075. |
| [23] |
Sattelberger R, Oliver G, Martínez E. Veterinärantibiotika in Wirtschaftsdünger und Boden[M]. Wien: Reports BE-272, 2005.
|
| [24] |
Elena M C, Carmen G B, Sigrid S, et al. Environmental monitoring study of selected veterinary antibiotics in animal manure and soils in Austria[J]. Environmental Pollution, 2007, 148(2): 570-579. DOI:10.1016/j.envpol.2006.11.035 |
| [25] |
Guo X Y, Hao L J, Qiu P Z, et al. Pollution characteristics of 23 veterinary antibiotics in livestock manure and manure-amended soils in Jiangsu Province, China[J]. Journal of Environmental Science and Health, 2016, 51(6): 383-392. DOI:10.1080/03601234.2016.1142743 |
| [26] |
Huang Y, Cheng M, Li W, et al. Simultaneous extraction of four classes of antibiotics in soil, manure and sewage sludge and analysis by liquid chromatography-tandem mass spectrometry with the isotope-labelled internal standard method[J]. Analytical Methods, 2013, 5(15): 3721-3731. DOI:10.1039/c3ay40220g |
| [27] |
Janusch F, Scherz G, Mohring S A I, et al. Determination of fluoroquinolones in chicken feces: A new liquid-liquid extraction method combined with LC-MS/MS[J]. Environmental Toxicology and Pharmacology, 2014, 38(3): 792-799. DOI:10.1016/j.etap.2014.09.011 |
| [28] |
Xie W Y, Yang X P, Li Q, et al. Changes in antibiotic concentrations and antibiotic resistome during commercial composting of animal manures[J]. Environmental Pollution, 2016, 219: 182-190. DOI:10.1016/j.envpol.2016.10.044 |
| [29] |
Pereira Leal R M, Figueira R F, Tornisielo V L, et al. Occurrence and sorption of fluoroquinolones in poultry litters and soils from Sao Paulo State, Brazil[J]. Science of the Total Environment, 2012, 432: 344-349. DOI:10.1016/j.scitotenv.2012.06.002 |
| [30] |
Wang N, Guo X, Xu J, et al. Pollution characteristics and environmental risk assessment of typical veterinary antibiotics in livestock farms in Southeastern China[J]. J Environ Sci Health B, 2014, 49(7): 468-479. DOI:10.1080/03601234.2014.896660 |
| [31] |
Hu X, Zhou Q, Luo Y. Occurrence and source analysis of typical veterinary antibiotics in manure, soil, vegetables and groundwater from organic vegetable bases, northern China[J]. Environmental Pollution, 2010, 158(9): 2992-2998. DOI:10.1016/j.envpol.2010.05.023 |
| [32] |
Huang X, Liu C, Li K, et al. Occurrence and distribution of veterinary antibiotics and tetracycline resistance genes in farmland soils around swine feedlots in Fujian Province, China[J]. Environmental Science and Pollution Research, 2013, 20(12): 9066-9074. DOI:10.1007/s11356-013-1905-5 |
| [33] |
刘锋, 廖德润, 李可, 等. 畜禽养殖基地磺胺类喹诺酮类和大环内酯类抗生素污染特征[J]. 农业环境科学学报, 2013, 32(4): 847-853. LIU Feng, LIAO De-run, LI Ke, et al. Pollution characteristics of the sulfonamides, quinolones and macrolides in the samples collected from livestock and poultry feedlots[J]. Journal of Agro-Environment Science, 2013, 32(4): 847-853. |
| [34] |
Hou J, Wan W, Mao D, et al. Occurrence and distribution of sulfonamides, tetracyclines, quinolones, macrolides, and nitrofurans in livestock manure and amended soils of northern China[J]. Environmental Science and Pollution Research, 2014, 22(6): 4545-4554. |
| [35] |
Zhang H, Zhou Y, Huang Y, et al. Residues and risks of veterinary antibiotics in protected vegetable soils following application of different manures[J]. Chemosphere, 2016, 152: 229-237. DOI:10.1016/j.chemosphere.2016.02.111 |
| [36] |
Dolliver H, Gupta S, Noll S. Antibiotic degradation during manure composting[J]. Journal of Environmental Quality, 2008, 37(3): 1245-1253. |
| [37] |
吴丹, 韩梅琳, 邹德勋, 等. 超高效液相色谱-串联质谱法检测鸡粪中16种残留抗生素[J]. 分析化学, 2017, 45(9): 1389-1396. WU Dan, HAN Mei-lin, ZOU De-xun, et al. Detection of 16 residual antibiotics in chicken manure by ultra performance liquid chromatography-tandem mass spectrometry[J]. Chinese Journal of Analytical Chemistry, 2017, 45(9): 1389-1396. |
| [38] |
冯瑶.鸡粪堆肥过程中诺氟沙星削减规律及微生物分子生态学机制[D].北京: 中国农业科学院, 2016. FENG Yao. Microbial ecological mechanism for degradation of norfloxacin during chicken manure composting[D]. Beijing: Chinese Academy of Agricultural Sciences, 2016. http://cdmd.cnki.com.cn/Article/CDMD-82101-1016169840.htm |
| [39] |
薛南冬.一种禽兽粪便中氟喳诺酮类抗生素的去除方法: 中国, 201510636598. 8[P]. 2016-01-20. XUE Nan-dong. A method for removing fluoroquinolone antibiotics in animal excrement: China, 201510636598. 8[P]. 2016-01-20. |
| [40] |
Slana M, Zigon D, Sollner-Dolenc M. Enrofloxacin degradation in broiler chicken manure under field conditions and its residuals effects to the environment[J]. Environmental Science and Pollution Research, 2017, 24(15): 13722-13731. DOI:10.1007/s11356-017-8722-1 |
| [41] |
Selvam A, Zhao Z, Wong J W C. Composting of swine manure spiked with sulfadiazine, chlortetracycline and ciprofloxacin[J]. Bioresource Technology, 2012, 126: 412-417. DOI:10.1016/j.biortech.2011.12.073 |
| [42] |
Liao X, Li B, Zou R, et al. Biodegradation of antibiotic ciprofloxacin: Pathways, influential factors, and bacterial community structure[J]. Environmental Science and Pollution Research, 2016, 23(8): 7911-7918. DOI:10.1007/s11356-016-6054-1 |
| [43] |
Parshikov I A, Sutherland J B. Microbial transformations of antimicrobial quinolones and related drugs[J]. Journal of Industrial Microbiology & Biotechnology, 2012, 39(12): 1731-1740. |
| [44] |
Kim D W, Heinze T M, Kim B S, et al. Modification of norfloxacin by a Microbacterium sp. strain isolated from a wastewater treatment plant[J]. Applied and Environmental Microbiology, 2011, 77: 6100-6108. DOI:10.1128/AEM.00545-11 |
| [45] |
Čvančarová M, Moeder M, Filipová A, et al. Biotransformation of fluoroquinolone antibiotics by ligninolytic fungi-Metabolites, enzymes and residual antibacterial activity[J]. Chemosphere, 2015, 136: 311-320. DOI:10.1016/j.chemosphere.2014.12.012 |
| [46] |
Čvančarová M, Moeder M, Filipová A, et al. Biotransformation of the antibiotic agent flumequine by ligninolytic fungi and residual antibacterial activity of the transformation mixtures[J]. Environmental Science & Technology, 2013, 47(24): 14128-14136. |
| [47] |
Rusch M, Kauschat A, Spielmeyer A, et al. Biotransformation of the antibiotic danofloxacin by xylaria longipes leads to an efficient reduction of its antibacterial activity[J]. Journal of Agricultural and Food Chemistry, 2015, 63(31): 6897-6904. DOI:10.1021/acs.jafc.5b02343 |
| [48] |
Rusch M, Spielmeyer A, Meissner J, et al. Efficient reduction of antibacterial activity and cytotoxicity of fluoroquinolones by fungal-mediated N-oxidation[J]. Journal of Agricultural and Food Chemistry, 2017, 65(15): 3118-3126. DOI:10.1021/acs.jafc.7b01246 |
| [49] |
Čvančarová M, Kresinova Z, Filipová A, et al. Biodegradation of PCBs by ligninolytic fungi and characterization of the degradation products[J]. Chemosphere, 2012, 88(11): 1317-1323. DOI:10.1016/j.chemosphere.2012.03.107 |
| [50] |
Muzikar M, Kresinova Z, Svobodova K, et al. Biodegradation of chlorobenzoic acids by ligninolytic fungi[J]. Journal of Hazardous Materials, 2011, 196: 386-394. DOI:10.1016/j.jhazmat.2011.09.041 |
| [51] |
Parshikov I A, Heinze T M, Moody J D, et al. The fungus Pestalotiopsis guepini as a model for biotransformation of ciprofloxacin and norfloxacin[J]. Applied Microbiology and Biotechnology, 2001, 56(3/4): 474-477. |
| [52] |
Marengo J R, Kok R A, Obrien K, et al. Aerobic biodegradation of(C-14)-sarafloxacin hydrochloride in soil[J]. Environmental Toxicology and Chemistry, 1997, 16(3): 462-471. DOI:10.1897/1551-5028(1997)016<0462:ABOCSH>2.3.CO;2 |
| [53] |
Williams A J, Parshikov I A, Moody J D, et al. Fungal transformation of an antimicrobial fluoroquinolone drug during growth on poultry litter materials[J]. Journal of Applied Poultry Research, 2004, 13(2): 235-240. DOI:10.1093/japr/13.2.235 |
| [54] |
De Witte B, Dewulf J, Demeestere K, et al. Ozonation and advanced oxidation by the peroxone process of ciprofloxacin in water[J]. Journal of Hazardous Materials, 2009, 161(2/3): 701-708. |
| [55] |
Adjei M D, Heinze T M, Deck J, et al. Transformation of the antibacterial agent norfloxacin by environmental mycobacteria[J]. Applied and Environmental Microbiology, 2006, 72(9): 5790-5793. DOI:10.1128/AEM.03032-05 |
| [56] |
Wetzstein H G, Schneider J, Karl W. Patterns of metabolites produced from the fluoroquinolone enrofloxacin by basidiomycetes indigenous to agricultural sites[J]. Applied Microbiology and Biotechnology, 2006, 71(1): 90-100. |
| [57] |
Prieto A, Moeder M, Rodil R, et al. Degradation of the antibiotics norfloxacin and ciprofloxacin by a white-rot fungus and identification of degradation products[J]. Bioresource Technology, 2011, 102(23): 10987-10995. DOI:10.1016/j.biortech.2011.08.055 |
| [58] |
Fuentes S, Mendez V, Aguila P, et al. Bioremediation of petroleum hydrocarbons: catabolic genes, microbial communities, and applications[J]. Applied Microbiology and Biotechnology, 2014, 98(11): 4781-4794. DOI:10.1007/s00253-014-5684-9 |
| [59] |
Yang Y, Wang Z, Xie S. Aerobic biodegradation of bisphenol A in river sediment and associated bacterial community change[J]. Science of the Total Environment, 2014, 470: 1184-1188. |
| [60] |
戴琦.环丙沙星废水的膜生物反应器处理特性及污泥中抗性基因的变化研究[D].上海: 上海师范大学, 2017. DAI Qi. Research on the treatment characteristics of the membrane bioreactor and the change of resistance genes in the sludge[D]. Shanghai: Shanghai Normal University, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10270-1017154083.htm |
| [61] |
Meng F, Gao G, Yang T T, et al. Effects of fluoroquinolone antibiotics on reactor performance and microbial community structure of a membrane bioreactor[J]. Chemical Engineering Journal, 2015, 280: 448-458. DOI:10.1016/j.cej.2015.06.025 |
| [62] |
Grenni P, Ancona V, Barra Caracciolo A. Ecological effects of antibiotics on natural ecosystems:A review[J]. Microchemical Journal, 2018, 136: 25-39. DOI:10.1016/j.microc.2017.02.006 |
| [63] |
Alexandrino D A M, Mucha A P, Almeida C M R, et al. Biodegradation of the veterinary antibiotics enrofloxacin and ceftiofur and associated microbial community dynamics[J]. Science of the Total Environment, 2017, 581-582: 359-368. DOI:10.1016/j.scitotenv.2016.12.141 |
| [64] |
Girardi C, Greve J, Lamshöft M, et al. Biodegradation of ciprofloxacin in water and soil and its effects on the microbial communities[J]. Journal of Hazardous Materials, 2011, 198: 22-30. DOI:10.1016/j.jhazmat.2011.10.004 |
| [65] |
Brandt K K, Amézquita A, Backhaus T, et al. Ecotoxicological assessment of antibiotics: A call for improved consideration of microorganisms[J]. Environment International, 2015, 85: 189-205. DOI:10.1016/j.envint.2015.09.013 |
| [66] |
何势.曝气生物滤池中环丙沙星去除行为及其对降解菌群抗药性的诱导作用[D].上海: 东华大学, 2016. HE Shi. Removal behavior of ciprofloxacin in the biological aerated filter and its effect on the resistance of bacteria[D]. Shanghai: Donghua University, 2016. http://cdmd.cnki.com.cn/Article/CDMD-10255-1016707970.htm |
| [67] |
Asgher M, Bhatti H N, Ashraf M, et al. Recent developments in biodegradation of industrial pollutants by white rot fungi and their enzyme system[J]. Biodegradation, 2008, 19(6): 771-783. DOI:10.1007/s10532-008-9185-3 |
| [68] |
Michelini L, Reichel R, Werner W, et al. Sulfadiazine uptake and effects on Salix fragilis L. and Zea mays L. plants[J]. Water Air and Soil Pollution, 2012, 223(8): 5243-5257. DOI:10.1007/s11270-012-1275-5 |
| [69] |
Accinelli C, Sacca M L, Batisson I, et al. Removal of oseltamivir (Tamiflu) and other selected pharmaceuticals from wastewater using a granular bioplastic formulation entrapping propagules of Phanerochaete Chrysosporium[J]. Chemosphere, 2010, 81(3): 436-443. DOI:10.1016/j.chemosphere.2010.06.074 |
| [70] |
Rodriguez-Rodriguez C E, Marco-Urrea E, Caminal G. Degradation of naproxen and carbamazepine in spiked sludge by slurry and solidphase Trametes versicolor systems[J]. Bioresource Technology, 2010, 101(7): 2259-2266. DOI:10.1016/j.biortech.2009.11.089 |
| [71] |
Blanquez A, Guillen F, Rodriguez J, et al. The degradation of two fluoroquinolone based antimicrobials by SilA, an alkaline laccase from Streptomyces ipomoeae[J]. World Journal of Microbiology & Biotechnology, 2016, 32(3): 1-8. |
| [72] |
沈怡雯, 黄智婷, 谢冰. 抗生素及其抗性基因在环境中的污染、降解和去除研究进展[J]. 应用与环境生物学报, 2015, 21(2): 181-187. SHEN Yi-wen, HUANG Zhi-ting, XIE Bing. Advances in research of pollution, degradation and removal of antibiotics and antibiotic resis tance genes in the environment[J]. Chinese Journal of Applied & Environmental Biology, 2015, 21(2): 181-187. |
| [73] |
Lucas D, Badia-Fabregat M, Vicent T, et al. Fungal treatment for the removal of antibiotics and antibiotic resistance genes in veterinary hospital wastewater[J]. Chemosphere, 2016, 152: 301-308. DOI:10.1016/j.chemosphere.2016.02.113 |
| [74] |
Rodriguez-Mozaz S, Chamorro S, Marti E, et al. Occurrence of antibiotics and antibiotic resistance genes in hospital and urban wastewaters and their impact on the receiving river[J]. Water Research, 2015, 69: 234-242. DOI:10.1016/j.watres.2014.11.021 |
| [75] |
赵祥.施用粪肥的设施菜地土壤中抗生素及抗性基因多样性及丰度的研究[D].泰安: 山东农业大学, 2017. ZHAO Xiang. Studies on the diversity and abundance of antibiotics and resistance genes in vegetable soils with facilities manure application[D]. Tai′an: Shandong Agricultural University, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10434-1017077365.htm |
| [76] |
Knapp C W, Zhang W, Sturm B S M, et al. Differential fate of erythromycin and beta-lactam resistance genes from swine lagoon waste under different aquatic conditions[J]. Environmental Pollution, 2010, 158(5): 1506-1512. DOI:10.1016/j.envpol.2009.12.020 |
| [77] |
Zhu Y G, Johnson T A, Su J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [78] |
Mu Q H, Li J, Sun Y X, et al. Occurrence of sulfonamide-, tetracycline-, plasmid-mediated quinolone-and macrolide-resistance genes in livestock feedlots in northern China[J]. Environmental Science and Pollution Research, 2015, 22(9): 6932. DOI:10.1007/s11356-014-3905-5 |
| [79] |
Zhang Y J, Hu H W, Gou M, et al. Temporal succession of soil antibiotic resistance genes following application of swine, cattle and poultry manures spiked with or without antibiotics[J]. Environmental Pollution, 2017, 231: 1621-1632. DOI:10.1016/j.envpol.2017.09.074 |
| [80] |
Chen K, Zhou J L. Occurrence and behavior of antibiotics in water and sediments from the Huangpu River, Shanghai, China[J]. Chemosphere, 2014, 95: 604-612. DOI:10.1016/j.chemosphere.2013.09.119 |
| [81] |
Wang J, Guo X, Qin J, et al. Microstructure and mechanical properties of investment casted titanium matrix composites with B4C additions[J]. Materials Science and Engineering: A, 2015, 628: 366-373. DOI:10.1016/j.msea.2015.01.067 |
| [82] |
Zhou X, Qiao M, Wang F H, et al. Use of commercial organic fertilizer increases the abundance of antibiotic resistance genes and antibiotics in soil[J]. Environmental Science and Pollution Research, 2017, 24(1): 701-710. |
| [83] |
Wang J, Ben W, Yang M, et al. Dissemination of veterinary antibiotics and corresponding resistance genes from a concentrated swine feedlot along the waste treatment paths[J]. Environment International, 2016, 92-93: 317-323. DOI:10.1016/j.envint.2016.04.020 |
| [84] |
郑宁国, 黄南, 王卫卫, 等. 高温堆肥过程对猪粪来源抗生素抗性基因的影响[J]. 环境科学, 2016, 37(5): 1986-1992. ZHENG Ning-guo, HUANG Nan, WANG Wei-wei, et al. Effects of high temperature composting on antibiotic resistance genes in pig manure[J]. Environmental Science, 2016, 37(5): 1986-1992. |
| [85] |
Selvam A, Xu D, Zhao Z, et al. Fate of tetracycline, sulfonamide and fluoroquinolone resistance genes and the changes in bacterial diversity during composting of swine manure[J]. Bioresource Technology, 2012, 126: 383-390. DOI:10.1016/j.biortech.2012.03.045 |
| [86] |
Qian X, Gu J, Sun W, et al. Diversity, abundance, and persistence of antibiotic resistance genes in various types of animal manure following industrial composting[J]. Journal of Hazardous Materials, 2018, 344: 716-722. DOI:10.1016/j.jhazmat.2017.11.020 |
| [87] |
Auerbach E A, Seyfried E E, McMahon K D. Tetracycline resistance genes in activated sludge wastewater treatment plants[J]. Water Research, 2007, 41(5): 1143-1151. DOI:10.1016/j.watres.2006.11.045 |
| [88] |
Garcia-Galan M J, Diaz-Cruz M S, Barcelo D. Occurrence of sulfonamide residues along the Ebro river basin removal in wastewater treatment plants and environmental impact assessment[J]. Environment International, 2011, 37(2): 462-473. DOI:10.1016/j.envint.2010.11.011 |
| [89] |
Su J Q, Wei B, OUYANG W Y, et al. Antibiotic resistome and its association with bacterial communities during sewage sludge composting[J]. Environmental Science & Technology, 2015, 49(12): 7356-7363. |
| [90] |
Qiao M, Ying G G, Singer A C, et al. Review of antibiotic resistance in China and its environment[J]. Environmental International, 2018, 110: 160-172. DOI:10.1016/j.envint.2017.10.016 |
 2019, Vol. 38
2019, Vol. 38