文章信息
- 杨晓燕, 李艳苓, 魏环宇, 朱昌雄, 李峰, 耿兵
- YANG Xiao-yan, LI Yan-ling, WEI Huan-yu, ZHU Chang-xiong, LI Feng, GENG Bing
- 阿特拉津降解菌CS3的分离鉴定及其降解特性的研究
- Isolation, identification, and characterization of atrazine-degrading bacterial strain CS3
- 农业环境科学学报, 2018, 37(6): 1149-1158
- Journal of Agro-Environment Science, 2018, 37(6): 1149-1158
- http://dx.doi.org/10.11654/jaes.2017-1453
文章历史
- 收稿日期: 2017-10-25
- 录用日期: 2018-01-26
阿特拉津(Atrazine)是一种被世界各地广泛应用的三嗪类除草剂,可防除农业和森林中的阔叶杂草及一些禾本科杂草[1-2]。虽然阿特拉津的杂草防除效果很好,但是由于其具有淋溶性好、易扩散、迁移率高等特点,导致其可以随排灌水和降水流失,进而对土壤[3-5]、地表水和地下水[3, 6-9]造成严重污染。此外,还有研究表明阿特拉津是一种内分泌干扰物和致癌物,能够干扰生物体内分泌系统,阻断正常的激素功能,导致先天缺陷、生殖肿瘤、两栖类和人类体重减轻等,进而影响生态平衡[10-11]。因此用生物法修复阿特拉津污染的环境受到广大研究工作者的密切关注并逐渐成为一个研究热点。
目前,已经报道的能够降解阿特拉津的细菌包括Pseudomonas sp.[12-13]、Arthrobacter sp.[14-15]、Acinetobacter sp.[16]、Rhodococcus sp.[17-18]、Pseudaminobacter sp.[19]、Nocardioides sp.[6]、Bacillus sp.[20]、Agrobacterium sp.[21-22]等许多属。其中革兰氏阴性菌Pseudomonas sp. ADP和革兰氏阳性菌Arthrobacter sp. TC1是目前研究的比较透彻的代表性菌株,前者对阿特拉津的降解是通过降解基因atzA、atzB、atzC、atzD、atzE和atzF编码的6个酶来完成,最终可以将阿特拉津完全矿化[23-24];后者则是由trzN、atzB、atzC这3个基因编码的酶催化完成,最终只能将阿特拉津降解为氰尿酸[15]。
尽管目前对阿特拉津生物降解的研究已经取得了显著的进步,但鉴于阿特拉津的应用范围及其毒性,仍需要分离更多有效的降解菌以适应不同环境需要。如Ye等[25]在2016年从中国东北的寒黑土壤中分离到Shewanella sp.菌株YJY4,为寒冷地区的阿特拉津污染修复提供新的候选菌株。本研究从河北省某农药厂排污废水中分离出一株阿特拉津降解菌Arthrobacter ureafaciens CS3,并对其生长降解特性和相关基因等进行了分析。本研究分离的菌株CS3具有较好的耐碱性,丰富了阿特拉津降解的菌种资源并为未来偏碱环境中阿特拉津污染修复提供了优良候选菌株。
1 材料与方法 1.1 材料 1.1.1 水样来源阿特拉津污染水样采集自河北省某农药厂排污河中的废水。
1.1.2 试剂与培养基阿特拉津标准样品和纯度97%的阿特拉津原药,购自北京百灵威科技有限公司。将阿特拉津原药溶解在甲醇中配制成10 000 mg·L-1的母液备用。氰尿酸(纯度98%)购自北京百灵威科技有限公司;用于高效液相色谱分析的试剂均为色谱纯,其余试剂均为分析纯。
无机盐基础培养基(MM):NaCl 1.0 g、K2HPO4 1.5 g、KH2PO4 0.5 g、MgSO4 0.2 g,蒸馏水定容至1 L,调节pH至7.0,121 ℃条件下灭菌30 min。
LB培养基:胰蛋白胨10 g、酵母粉5 g、NaCl 10 g,蒸馏水定容至1 L,调节pH至7.0,121 ℃条件下灭菌30 min。
1.2 方法 1.2.1 菌株的富集与分离在无菌条件下取10 mL阿特拉津污染废水水样加入到含有50 mg·L-1阿特拉津的100 mL MM中驯化培养,每个样品6次重复,30 ℃、150 r·min-1振荡培养7 d。然后取10 mL培养液接种到100 mL新鲜的培养基中,并逐渐增加培养基中阿特拉津浓度。按相同的富集培养方式培养,直至阿特拉津浓度增加到1000 mg·L-1,驯化结束。同时将所有浓度的富集培养物分别稀释10-1、10-2、10-3、10-4、10-5、10-6、10-7、10-8倍,各取0.1 mL稀释液分别涂布于含有相应阿特拉津浓度的LB固体平板上,置于30 ℃恒温培养箱中培养。培养结束后,挑取LB固体平板上不同形态特征的单菌落,在LB平板上连续划线纯化培养,得到纯化菌株。
1.2.2 高效降解菌株的筛选将得到的纯化菌株分别接种到LB液体培养基中30 ℃过夜培养。将培养液以5000 r·min-1离心3 min收集菌体,并用无菌水洗涤两次。接种1 mL菌悬液到含有50 mg·L-1阿特拉津的50 mL MM中,30 ℃、150 r·min-1培养7 d。用高效液相色谱法定量测定各样品中阿特拉津的残留浓度,并计算降解率,从中筛选出高效阿特拉津降解菌。
1.2.3 高效降解菌株的鉴定根据常见细菌系统鉴定手册对菌株的形态和生理生化特征进行鉴定[26]。菌株16S rRNA基因的克隆及序列分析参照如下步骤进行:(1)基因组DNA的提取按照北京全式金生物技术有限公司的细菌基因组DNA提取试剂盒说明书进行;(2)采用16S rRNA基因通用引物27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和1492R(5′-GGTTACCTTGTTACGACTT-3′),以基因组DNA为模板,进行PCR扩增。扩增条件为:95 ℃预变性2 min,然后95 ℃变性30 s,58 ℃退火30 s,72 ℃延伸90 s,共35个循环,最终72 ℃延伸10 min。使用1%琼脂糖凝胶电泳检测扩增产物大小并用北京全式金生物技术有限公司的EasyPure Quick Gel Extraction Kit试剂盒纯化PCR产物。纯化后的PCR产物由生工生物(上海)工程股份有限公司进行测序。通过NCBI网站上的BLAST程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)将菌株16S rRNA基因序列与GeneBank数据库中的已知序列进行比对,得出相似性。使用MEGA 6.0软件中的Neighbor-Joining法对菌株的16S rRNA基因序列进行聚类分析[27],并构建系统进化树,用Bootstrap值进行检验,并重复1000次。
1.2.4 外加碳氮源对菌株生长及降解阿特拉津效果的影响为了探究外加碳氮源对菌株CS3生长和降解阿特拉津的影响,本研究设立了四个不同处理,即在MM中分别加入50 mg·L-1阿特拉津作为唯一氮源,1 g·L-1蔗糖作为外加碳源(AT+C);50 mg·L-1阿特拉津作为唯一碳源,1 g·L-1的NH4NO3作为外加氮源(AT+N);50 mg·L-1阿特拉津和1 g·L-1的NH4NO3作为氮源,1 g·L-1蔗糖作为外加碳源(AT+CN);只加入50 mg·L-1阿特拉津作为唯一碳氮源(AT)。然后将1 mL浓度为1×108 CFU·mL-1的菌悬液接种到100 mL上述四种培养基中,同时设立不接种菌液的样品为空白对照。每个处理组设三个重复。将所有样品在30 ℃、180 r·min-1条件下振荡培养48 h,每隔12 h取样测定样品中的菌浓度和阿特拉津的残留浓度,并计算阿特拉津的降解率。
另外,为了研究不同碳源对菌株生长及降解阿特拉津效果的影响,本研究分别在MM中添加1 g·L-1葡萄糖、蔗糖、乳糖、柠檬酸钠作为外加碳源,然后将pH调节至7.0,121 ℃条件下灭菌30 min。将1 mL浓度为1×108 CFU·mL-1的菌悬液接种到上述100 mL含有不同外加碳源的培养基中,并加入0.5 mL浓度为10 000 mg·L-1的阿特拉津甲醇母液,使阿特拉津的终浓度为50 mg·L-1。同时设立没有添加外加碳源的样品为空白对照。每个处理组设三个重复。将所有样品在30 ℃、180 r·min-1条件下振荡培养。分别在培养24 h和48 h时取样测定样品中的菌浓度和阿特拉津的残留浓度,并计算阿特拉津的降解率。
菌浓度测定方法:通过紫外分光光度计测定样品在600 nm处的吸光值(OD600)来表示,阿特拉津降解率的计算参照方法1.2.8进行。
1.2.5 环境因素对菌株生长及降解阿特拉津效果的影响为了研究不同的环境因素如pH、温度、阿特拉津浓度等对菌株生长及降解阿特拉津的影响,本研究分别设置不同温度(4、10、20、30、37、45 ℃)、不同pH值(4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0、12.0)和不同阿特拉津浓度(5、25、50、75、100、200 mg·L-1)处理组。分别将1 mL浓度为1×108 CFU·mL-1的菌悬液接种到100 mL含1 g·L-1蔗糖的MM中,并加入0.5 mL浓度为10 000 mg·L-1的阿特拉津甲醇母液,使阿特拉津的终浓度为50 mg·L-1。同时设立不接种菌液的样品为空白对照,每个处理组设三个重复。将所有样品在30 ℃(不同温度实验除外)、180 r·min-1条件下振荡培养。分别在培养24 h和48 h时取样测定样品中的菌浓度和阿特拉津的残留浓度,并计算阿特拉津的降解率。
1.2.6 阿特拉津降解基因的检测参考前人报道的atzA、atzB、atzC、atzD、atzE、atzF、trzD和trzN基因引物序列[23, 28-30],以菌株CS3的基因组DNA为模板对上述8种降解基因进行PCR扩增。扩增条件为:95 ℃预变性5 min,然后94 ℃变性1 min,最佳退火温度退火60 s,72 ℃延伸2 min,共35个循环,最终72 ℃延伸10 min。使用1%琼脂糖凝胶电泳检测PCR产物,并由生工生物(上海)工程股份有限公司进行测序。将测序结果通过NCBI网站上的BLAST程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)与GeneBank数据库中已知序列进行同源性比对。
1.2.7 菌株的生长及降解曲线将1 mL浓度为1×108 CFU·mL-1的菌悬液接种到100 mL含1 g·L-1蔗糖的MM中,然后加入0.5 mL浓度为10 000 mg·L-1的阿特拉津甲醇母液,使阿特拉津的终浓度为50 mg·L-1。将样品分别在30 ℃、180 r·min-1条件下振荡培养。每隔6 h取样并测定样品中的菌浓度、阿特拉津的残留浓度和氰尿酸浓度。每个处理设三个重复。
1.2.8 阿特拉津和氰尿酸的测定将培养液与二氯甲烷以1:2的比例混合萃取3次,合并有机相。将合并的有机萃取液放入圆底烧瓶中,用旋转蒸发仪将液体浓缩并蒸发至干,最后用色谱纯的甲醇溶液调节体积。之后将样品通过0.22 μm尼龙膜过滤,并装进样瓶中备用。
采用HP1050型高效液相色谱(HPLC)仪检测阿特拉津和氰尿酸的浓度。阿特拉津检测的液相条件为:反相C18色谱柱(4.6 mm×250 mm,5 μm),流动相配比为甲醇:水=70:30(V/V),流速为0.8 mL·min-1,可变波长紫外检测器,检测波长为225 nm,柱温:40 ℃,进样量:10 μL。氰尿酸检测的液相条件为:Thermo氨基色谱柱(4.6 mm×250 mm,5 μm),流动相配比为甲醇:0.1%NH3·H2O=70:30(V/V),流速:1.0 mL·min-1,可变波长紫外检测器,检测波长为215 nm,柱温:30 ℃,进样量:10 μL。
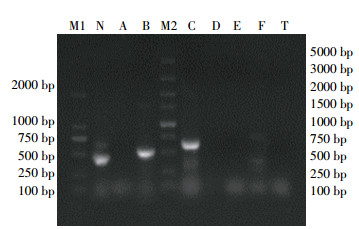
根据HPLC测定的结果,计算阿特拉津的降解率,计算公式如下:
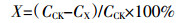
式中:X为阿特拉津的降解率,%;CX为阿特拉津的终浓度,mg·L-1;CCK为阿特拉津的初始浓度,mg·L-1。
1.3 数据分析统计方法实验结果用平均值和标准差来表示。采用软件SPSS 21.0对数据进行单因素方差分析(ANOVA)和相关性分析以确定所有处理组之间的差异。设定P<0.05为显著性差异。利用Origin 9.0软件进行作图。
2 结果与分析 2.1 高效阿特拉津降解菌的分离及鉴定经分离纯化,最终筛选出1株高效阿特拉津降解菌,命名为菌株CS3。菌株CS3为革兰氏阳性菌,在LB平板上的菌落形态特征为菌落边缘整齐,表面凸起,呈金黄色,不透明,粘稠,光滑的圆形菌落。菌株CS3的菌体形态特征如图 1所示,在培养过程中有明显的杆、球变化特征,培养2 d的幼龄菌体呈长杆状,培养6 d后的老龄菌体则呈球状或短杆状。菌株CS3不水解淀粉,不产H2S和吲哚,氧化酶阴性,甲基红试验、V-P试验、硝酸盐还原试验均为阴性;接触酶阳性,能够产氨。

|
| 左图为培养2 d的幼龄菌体;右图为培养6 d的老龄菌体 The left image represents young cells cultivated for 2 d;The right image represents aged cells cultivated for 6 d 图 1 菌株CS3的扫描电镜图(×20 000倍) Figure 1 Scanning electron micrograph of strain CS3(×20 000 times) |
通过PCR扩增得到菌株CS3的16S rRNA基因序列长度为1394 bp(GenBank登录号为MF612193)。通过NCBI的BLAST程序与已知序列比对可知,菌株CS3与产脲节杆菌(Arthrobacter ureafaciens)的16S rRNA基因序列同源,相似性在99%以上;与其余多株节杆菌属细菌的16S rRNA基因序列亦同源,相似性都在97%以上。菌株CS3的系统发育树(图 2)遗传距离显示菌株CS3与Arthrobacter ureafaciens的亲缘关系最近,并与其聚集在同一个分支上。结合其形态特征及生理生化特征,将菌株CS3初步鉴定为产脲节杆菌(Arthrobacter ureafaciens)。
|
| 采用邻近法进行计算(bootstrap number = 1000),误差棒代表每个核苷酸的碱基置换率为0.005 The calculations were performed according to a neighbour-joining analysis(bootstrap number = 1000), and the bar indicates 0.005 substitution per nucleotide position 图 2 基于16S rRNA基因序列构建的菌株CS3的系统发育树 Figure 2 Phylogenetic tree based on the 16S rRNA gene sequences of strain CS3 |
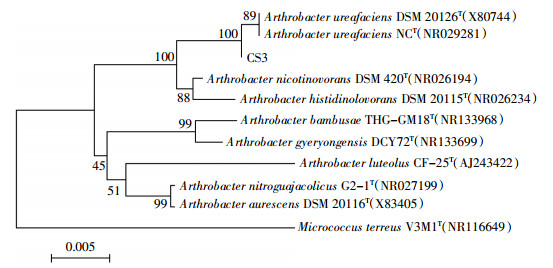
图 3显示了外加碳氮源对菌株CS3生长的影响和菌株CS3降解阿特拉津的影响。在整个培养过程中,处理组(AT+CN)和(AT+C)中菌株CS3的菌浓度都显著高于(AT+N)和(AT)处理组(P<0.05)。处理组(AT+N)和(AT)中的菌株CS3的菌浓度增长微弱,且这两个处理组之间无显著性差异(P<0.05)。以上结果表明外加碳源能够明显促进菌株CS3的生长,外加氮源对其生长影响不大,并且菌株CS3能够利用阿特拉津作为唯一氮源生长,不能利用其作为唯一碳源生长。培养36 h之前,处理组(AT+CN)和(AT+C)中的阿特拉津降解率均显著高于处理组(AT+N)和(AT)。36 h之后所有处理组中的阿特拉津基本都被完全降解,各处理组之间无显著性差异,表明外加碳源或氮源对阿特拉津降解率的影响并不大。

|
| OD600表示菌浓度。不同的小写字母表示不同处理之间存在显著性差异(P<0.05) OD600 represent concentrations of strain CS3. Different lowercase letters indicate significant difference(P < 0.05) between the treatments detected by ANOVA test 图 3 外加碳氮源对菌株CS3生长及阿特拉津降解的影响 Figure 3 Growth and atrazine degradation of strain CS3 with exogenous carbon or nitrogen sources |
外加不同碳源对菌株CS3生长的影响如图 4所示。在整个培养过程中,在含有外加蔗糖的处理组中菌株CS3的菌浓度都明显高于其他处理组;其次为乳糖。葡萄糖处理组中的菌株生长最慢,与对照组之间无显著性差异。以上结果表明外加蔗糖、乳糖和柠檬酸钠均会明显促进菌株CS3的生长,其中最佳外加碳源为蔗糖。葡萄糖对菌株CS3的生长无显著影响。图 4还显示了外加不同碳源对菌株CS3降解阿特拉津的影响。在培养24 h时,在含有外加蔗糖的处理组中菌株CS3对阿特拉津的降解效果最好且显著高于其他处理组,其次为乳糖。培养48 h之后,外加蔗糖、乳糖和柠檬酸钠的3个处理组中阿特拉津降解率均达到95%以上,无显著性差异。此时葡萄糖处理组中菌株CS3对阿特拉津的降解率最低,仅为29.53%,甚至远低于对照组的83.04%。综上可知,外加葡萄糖会抑制菌株CS3对阿特拉津的降解,外加蔗糖、乳糖和柠檬酸钠则均会促进菌株CS3对阿特拉津的降解。
|
| OD600表示菌浓度。不同的小写字母表示不同处理之间存在显著性差异(P<0.05) OD600 represent concentrations of strain CS3. Different lowercase letters indicate significant difference(P < 0.05) between the treatments detected by ANOVA test 图 4 外加不同碳源对菌株CS3生长及阿特拉津降解的影响 Figure 4 Growth and atrazine degradation of strain CS3 with different carbon sources |
图 5显示了不同pH值、不同温度和不同阿特拉津浓度对菌株CS3生长及降解阿特拉津效果的影响。当pH值为7时菌株CS3的生长最好,对阿特拉津的降解率也最高。当pH为6时,菌株生长及对阿特拉津的降解都受到较大影响,48 h后的阿特拉津降解率下降为58.90%。当pH低于4时,菌株几乎不能生长,48 h后对阿特拉津的降解率低于20%,表明该菌株不适宜在酸性环境降解阿特拉津。当pH为11时,菌株仍能较好地生长,48 h后对阿特拉津的降解率依然保持在90%以上。当pH为12时,菌株生长及对阿特拉津的降解则会受到较大影响,48 h后对阿特拉津的降解率为31.40%。由此可知菌株CS3能够生长和降解阿特拉津的pH范围在5~11之间,范围很宽且具有较好的耐碱性,偏碱性环境对阿特拉津的降解效果影响不大。因此该菌株可以作为未来偏碱环境中修复阿特拉津污染的优良候选菌株。

|
| 不同小写字母表示不同处理之间存在显著性差异(P<0.05) Different lowercase letters are different significantly between different treatments(P < 0.05) 图 5 不同环境条件对菌株CS3生长及阿特拉津降解的影响 Figure 5 Growth and atrazine degradation by strain CS3 under different culture conditions |
温度过高或过低会抑制阿特拉津降解菌的生长,同时影响阿特拉津降解菌代谢和降解酶活性[31]。由图 5可知,菌株CS3生长和降解阿特拉津的最佳温度为30 ℃,在温度范围为10~37 ℃之间,具有一定的生长和降解能力。当温度低于4 ℃或高于45 ℃时,菌株CS3几乎不能生长。显著性分析表明在实验温度条件下菌株CS3的生长与对阿特拉津的降解率之间存在极显著相关性(R=0.959**)。
由图 5可知,当阿特拉津浓度为5 mg·L-1时,菌株CS3的浓度基本保持不变,OD600的值没有明显增加,原因可能是低浓度阿特拉津不能够为菌株生长提供足够的营养。阿特拉津浓度在5~50 mg·L-1之间时,OD600的值随着阿特拉津浓度的增高而逐渐增加。当阿特拉津浓度超过50 mg·L-1后,OD600值开始下降,推测可能是因为高浓度的阿特拉津会对菌株CS3的生长产生抑制作用。当阿特拉津浓度高于75 mg·L-1时,由于其低水溶性(33 mg·L-1)而不能完全溶解在培养基中,因此之后未测定OD600的值。培养24 h时,随着阿特拉津浓度的升高,降解率逐渐降低。培养48 h后,只有阿特拉津浓度为200 mg·L-1处理组的阿特拉津降解率为96.15%,其余处理组阿特拉津降解率都达到98.81%以上,且与其余处理组之间都存在显著性差异。但在试验最高浓度(200 mg·L-1)以内,阿特拉津的降解率最终不会受到太大影响。另经实验证明,菌株CS3能在6 d内将500 mg·L-1的阿特拉津降解完全(数据未显示)。综上可知,菌株CS3对低浓度和高浓度的阿特拉津都具有较好的降解能力。
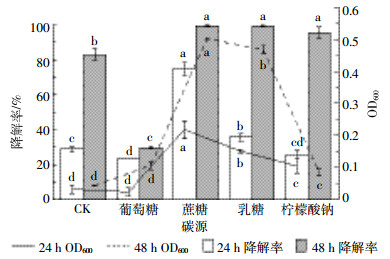
2.4 阿特拉津降解基因的检测通过PCR扩增,分别获得444 bp的trzN基因、537 bp的atzB基因和630 bp的atzC基因。尽管本研究还尝试了用不同的扩增条件和引物去扩增其他5种降解基因,但是最终都未成功(图 6)。将序列测序后与GenBank数据库中的序列比对,结果显示本研究获得的基因trzN(GenBank登录号MF774327)分别与Arthrobacter sp.SD41(KP994319)[32]和Arthrobacter sp.C3(KR263873)[33]的trzN基因同源性分别为100%、99%。基因atzB(GenBank登录号MF774328)和atzC(GenBank登录号MF774329)分别与Arthrobacter aurescens TC1(AY456696)[34]的atzB和atzC基因的同源性分别为98%和99%。
|
| M1:DL 2000 DNA marker; N:trzN; A:atzA; B:atzB; M2:DL 5000 DNA marker; C:atzC; D:atzD; E:atzE; F:atzF; T:trzD 图 6 1%琼脂糖凝胶电泳检测菌株CS3的阿特拉津降解基因 Figure 6 Agarose gel electrophoresis(1%) detection of atrazine-degrading genes of strain CS3 |
从图 7可以看出,12 h内OD600的值增加缓慢,对阿特拉津的降解效果也不明显,可能因为前12 h菌株CS3处于延滞期,对于环境需要一个适应的过程。从12 h开始菌株进入对数生长期,OD600的值增长迅速,其对阿特拉津的降解速度也相应加快。到48 h时菌株CS3进入生长稳定期,OD600的值基本保持不变,此时阿特拉津也被降解完全。在整个降解过程中,氰尿酸的浓度变化与阿特拉津浓度变化趋势完全相反,从12 h至36 h之间浓度急剧增加,到48 h后保持在30 mg·L-1左右不再增加。相关性分析也表明,菌株的生长与阿特拉津的降解之间具有极显著正相关性(R=0.991**),而氰尿酸浓度与阿特拉津浓度之间具有极显著负相关性(R=-0.989**)。此外,阿特拉津的初始平均摩尔浓度等于累积的氰尿酸的平均摩尔浓度。以上结果表明,阿特拉津的降解和菌株的生长基本同步。结合菌株CS3降解基因的检测结果,推测菌株CS3可能只能降解阿特拉津为无毒的氰尿酸,而不能进一步代谢氰尿酸为CO2和NH3。

|
| OD600表示菌浓度 OD600 represent concentrations of strain CS3 图 7 菌株CS3的生长曲线及阿特拉津降解曲线 Figure 7 Growth curve of strain CS3 and atrazine degradation curve by strain CS3 |
目前已经报道了许多从不同地方分离到的属于节杆菌属的阿特拉津降解菌,如菌株HIM[35]、GZK-1[1]、KU001[36]、TC1[34]、ADH-2[37]、AD26[38]、MCM B-436[39]、AG1[40]、FM326[41]等。另有许多报道指出,该属细菌能够降解许多其他有机污染物,如苯胺、苯酚、石油、有机磷农药等[15, 39]。这些工作显示出节杆菌属菌株在污染位点生物修复方面具有巨大潜力。
研究报道多数阿特拉津降解菌能够以阿特拉津为氮源进行生长并将它降解[14, 38, 42-44],因此外加氮源会抑制多数菌株对阿特拉津的利用和降解[45],对外源氮源不敏感的阿特拉津降解菌的报道很少。本研究中菌株CS3也能够利用阿特拉津为唯一氮源,但是外加氮源对其生长和降解阿特拉津没有太大影响,具体原因还在进一步研究中。
本研究中,外加蔗糖、乳糖和柠檬酸钠能够促进菌株CS3的生长及对阿特拉津的降解,外加葡萄糖则会抑制其对阿特拉津的降解。不同外加碳源对不同阿特拉津降解菌的影响不同。在某些情况下,外加碳源甚至可以抑制阿特拉津的降解。前人研究报道,外加蔗糖和葡萄糖能显著促进菌株ADH-2的生长,但对阿特拉津的降解表现出显著的抑制,而淀粉对其生长和阿特拉津降解均表现出促进效应[37]。Wang等[43]研究表明加入蔗糖、柠檬酸钠或半乳糖可显著促进菌株DAT1的生长,蔗糖是其生长最适合的碳源。然而,Wang等[46]2011年发现加入蔗糖并没有加速菌株HB-5对阿特拉津的降解。李绍峰等[47]研究发现降解菌株L-6可以利用葡萄糖、果糖和柠檬酸钠生长,不能利用蔗糖、乳糖和淀粉。因此,不同阿特拉津降解菌的生长及对阿特拉津的降解依赖于有机物质的类型[42]。
与许多菌株,如菌株HB-5[46]、DNS10[42]、T3AB1[48]、X-4[49]、FM326[41]等相比,菌株CS3能够生长并降解阿特拉津的pH范围更宽。菌株CS3降解阿特拉津的pH范围不仅很宽,且具有一定的嗜碱性,在pH值为11的条件下,也具有很高的降解特性,这对于未来偏碱环境中修复阿特拉津提供了更好的选择。
已经分离到的节杆菌属阿特拉津降解菌株中,除AD1[14]、MCMB436[39]、C3[33]外,许多其他Arthrobacter sp.菌株都含有trzN、atzB、atzC基因。菌株CS3中包含的阿特拉津降解基因组合与许多已经报道的节杆菌菌株如SD41[32]、TC1[34]、ADH-2[37]、AD26[38]、X-4[49]、KU001[36]、DNS10[42]、DAT1[43]等含有相同的基因组合(trzN-atzBC)。结合前人报道的阿特拉津降解基因及降解途径的研究报道[23-24],初步推测其只能将阿特拉津降解为无毒的氰尿酸,而不能完全将阿特拉津矿化。菌株CS3降解阿特拉津的实验结果也显示,当阿特拉津被降解时会积累氰尿酸,且阿特拉津的初始平均摩尔浓度等于累积的氰尿酸的平均摩尔浓度。因此可以推测菌株CS3通过trzN-atzB-atzC代谢途径,经水解脱氯和脱烷基化,将阿特拉津转化为氰尿酸,但不能进一步催化三嗪环的裂解,使氰尿酸进一步降解为CO2和NH3[1, 50-51]。
菌株CS3具有较好的降解性,对低浓度和高浓度阿特拉津都具有较好的降解能力。其降解速率远高于Zhang等[31]筛选的菌株N1以及Vaishampayan等[39]报道的菌株MCM B-436,前者96 h将20 mg·L-1阿特拉津降解66.4%,后者30 h将25 mg·L-1阿特拉津完全降解。
目前本研究只是在实验室条件下进行,下一步将重点研究其在生态环境中的实际应用效果及特性,为进一步提高其实际应用效果提供基础。此外,关于菌株CS3与其他降解菌之间是否存在共存互作以及相互之间的互作机制也在进一步研究当中。
4 结论(1) 从河北某农药厂排污河中的废水中分离出一株以阿特拉津为唯一氮源生长的高效降解阿特拉津降解菌CS3。经生理生化鉴定和16S rRNA基因序列分析,将其鉴定为产脲节杆菌(Arthrobacter ureafaciens)。
(2) 菌株CS3含有降解基因trzN、atzB、atzC,能够将阿特拉津降解为无毒的氰尿酸。
(3) 在最适条件(30℃,pH为7)下,菌株CS3能在48 h内完全降解50 mg·L-1的阿特拉津。甚至能够在6 d内将500 mg·L-1的阿特拉津完全降解,表明该菌株具有很好的降解能力。
(4) 菌株CS3生长和降解阿特拉津的温度和pH范围较宽,分别为10~37 ℃和5~11,显示出较好的环境生存能力及较好的生物修复潜力。此外,菌株CS3生长和降解阿特拉津的pH范围表明该菌株具有较好的耐碱性,为未来偏碱环境中阿特拉津污染修复提供了良好的候选菌株。
| [1] |
Getenga Z, Dörfler U, Iwobi A, et al. Atrazine and terbuthylazine mineralization by an Arthrobacter sp.isolated from a sugarcane-cultivated soil in Kenya[J]. Chemosphere, 2009, 77(4): 534-539. DOI:10.1016/j.chemosphere.2009.07.031 |
| [2] |
Lima D, Viana P, André S, et al. Evaluating a bioremediation tool for atrazine contaminated soils in open soil microcosms:The effectiveness of bioaugmentation and biostimulation approaches[J]. Chemosphere, 2009, 74(2): 187-192. DOI:10.1016/j.chemosphere.2008.09.083 |
| [3] |
Houot S, Topp E, Yassir A, et al. Dependence of accelerated degradation of atrazine on soil pH in French and Canadian soils[J]. Soil Biology & Biochemistry, 2000, 32(5): 615-625. |
| [4] |
Bastos A C, Magan N. Trametes versicolor:Potential for atrazine bioremediation in calcareous clay soil, under low water availability conditions[J]. International Biodeterioration & Biodegradation, 2009, 63(4): 389-394. |
| [5] |
Rousseaux S, Hartmann A, Soulas G. Isolation and characterisation of new Gram-negative and Gram-positive atrazine degrading bacteria from different French soils[J]. Fems Microbiology Ecology, 2001, 36(2/3): 211-222. |
| [6] |
Omotayo A E, Ilori M O, Radosevich M, et al. Metabolism of atrazine in liquid cultures and soil microcosms by Nocardioides strains isolated from a contaminated nigerian agricultural soil[J]. Soil & Sediment Contamination An International Journal, 2013, 22(4): 365-375. |
| [7] |
Singh S N, Jauhari N. Degradation of atrazine by plants and microbes[M]//Microbe-Induced Degradation of Pesticides. Springer International Publishing, 2017.
|
| [8] |
Fan X, Song F. Bioremediation of atrazine:recent advances and promises[J]. Journal of Soils & Sediments, 2014, 14(10): 1727-1737. |
| [9] |
Solomon K, Carr J P L, Giesy J, et al. Effects of atrazine on fish, amphibians, and aquatic reptiles:A critical review[J]. Critical Reviews in Toxicology, 2008, 38(9): 721-772. DOI:10.1080/10408440802116496 |
| [10] |
Shenoy K. Environmentally realistic exposure to the herbicide atrazine alters some sexually selected traits in male guppies[J]. PloS One, 2012, 7(2): e30611. DOI:10.1371/journal.pone.0030611 |
| [11] |
Hayes T B, Case P, Chui S, et al. Pesticide mixtures, endocrine disruption, and amphibian declines:Are we underestimating the impact?[J]. Environmental Health Perspectives, 2006, 114(Suppl 1): 40-50. |
| [12] |
Fernandes A F, Da S M, Martins V V, et al. Isolation and characterization of a Pseudomonas aeruginosa from a virgin Brazilian Amazon region with potential to degrade atrazine[J]. Environmental Science & Pollution Research International, 2014, 21(24): 13974-13978. |
| [13] |
Mandelbaum R T, Allan D L, Wackett L P. Isolation and characterization of a Pseudomonas sp.that mineralizes the s-triazine herbicide atrazine[J]. Applied & Environmental Microbiology, 1995, 61(4): 1451-1457. |
| [14] |
Cai B, Han Y, Liu B, et al. Isolation and characterization of an atrazine-degrading bacterium from industrial wastewater in China[J]. Letters in Applied Microbiology, 2003, 36(5): 272-276. DOI:10.1046/j.1472-765X.2003.01307.x |
| [15] |
Strong L C, Rosendahl C, Johnson G, et al. Arthrobacter aurescens TC1 metabolizes diverse s-triazine ring compounds[J]. Applied & Environmental Microbiology, 2002, 68(12): 5973-5980. |
| [16] |
Singh P, Suri C R, Cameotra S S. Isolation of a member of Acinetobacter species involved in atrazine degradation[J]. Biochemical & Biophysical Research Communications, 2004, 317(3): 697-702. |
| [17] |
Kolekar P D, Phugare S S, Jadhav J P. Biodegradation of atrazine by Rhodococcus sp.BCH2 to N -isopropylammelide with subsequent assessment of toxicity of biodegraded metabolites[J]. Environmental Science & Pollution Research International, 2014, 21(3): 2334-2345. |
| [18] |
Fazlurrahman, Batra M, Pandey J, et al. Isolation and characterization of an atrazine-degrading Rhodococcus sp.strain MB-P1 from contaminated soil[J]. Letters in Applied Microbiology, 2009, 49(6): 721-729. DOI:10.1111/lam.2009.49.issue-6 |
| [19] |
Topp E, Zhu H, Nour S M, et al. Characterization of an atrazine-degrading Pseudaminobacter sp.isolated from Canadian and French agricultural soils[J]. Applied & Environmental Microbiology, 2000a, 66(7): 2773-2782. |
| [20] |
Wang J, Zhu L, Wang Q, et al. Isolation and characterization of atrazine mineralizing Bacillus subtilis strain HB-6[J]. PloS One, 2014, 9(9): e107270. DOI:10.1371/journal.pone.0107270 |
| [21] |
Struthers J K, Jayachandran K, Moorman T B. Biodegradation of atrazine by Agrobacterium radiobacter J14a and, use of this strain in bioremediation of contaminated soil[J]. Applied & Environmental Microbiology, 1998, 64(9): 3368-3375. |
| [22] |
Siripattanakul S, Wirojanagud W, Mcevoy J M, et al. Atrazine removal in agricultural infiltrate by bioaugmented polyvinyl alcohol immobilized and free Agrobacterium radiobacter, J14a:A sand column study[J]. Chemosphere, 2009, 74(2): 308-313. DOI:10.1016/j.chemosphere.2008.09.005 |
| [23] |
Martinez B, Tomkins J, Wackett L P, et al. Complete nucleotide sequence and organization of the atrazine catabolic plasmid pADP-1 from Pseudomonas sp.strain ADP[J]. Journal of Bacteriology, 2001, 183(19): 5684-5697. DOI:10.1128/JB.183.19.5684-5697.2001 |
| [24] |
Wackett L P, Sadowsky M J, Martinez B, et al. Biodegradation of atrazine and related s-triazine compounds:from enzymes to field studies[J]. Applied Microbiology & Biotechnology, 2002, 58(1): 39-45. |
| [25] |
Ye J, Zhang J, Gao J, et al. Isolation and characterization of atrazine-degrading strain Shewanella sp.YJY4 from cornfield soil[J]. Letters in Applied Microbiology, 2016, 63(1): 45-52. DOI:10.1111/lam.2016.63.issue-1 |
| [26] |
东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001. DONG Xiu-zhu, CAI Miao-ying. Manual of systematic determinative familiar bacteriology[M]. Beijing: Science Press, 2001. |
| [27] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:Molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [28] |
De Souza M L, Seffernick J, Martinez B, et al. The atrazine catabolism genes atzABC are widespread and highly conserved[J]. Journal of Bacteriology, 1998, 180(7): 1951-1954. |
| [29] |
Mulbry W W, Zhu H, Nour S M, et al. The triazine hydrolase gene trzN from Nocardioides sp.strain C190:Cloning and construction of gene-specific primers[J]. Fems Microbiology Letters, 2002, 206(1): 75-79. DOI:10.1111/fml.2002.206.issue-1 |
| [30] |
Devers M, El A N, Kolic N U, et al. Detection and organization of atrazine-degrading genetic potential of seventeen bacterial isolates belonging to divergent taxa indicate a recent common origin of their catabolic functions[J]. Fems Microbiology Letters, 2007, 273(1): 78-86. DOI:10.1111/fml.2007.273.issue-1 |
| [31] |
Zhang H, Zhang Y, Hou Z, et al. Biodegradation of triazine herbicide metribuzin by the strain Bacillus sp.N1[J]. Journal of Environmental Science & Health Part B, 2014, 49(2): 79-86. |
| [32] |
李红梅, 张新建, 李纪顺, 等. 阿特拉津降解菌SD41的分离鉴定及土壤修复[J]. 环境科学与技术, 2014, 37(4): 38-41. LI Hong-mei, ZHANG Xin-jian, LI Ji-shun, et al. Experimental study on atrazine-degrading strain SD41:Isolation, identification and soil remediation[J]. Environmental Science & Technology, 2014, 37(4): 38-41. |
| [33] |
Wang H, Liu Y, Li J, et al. Biodegradation of atrazine by Arthrobacter sp.C3, isolated from the herbicide-contaminated corn field[J]. International Journal of Environmental Science & Technology, 2016, 13(1): 257-262. |
| [34] |
Sajjaphan K, Shapir N, Wackett L P, et al. Arthrobacter aurescens TC1 atrazine catabolism genes trzN, atzB, and atzC are linked on a 160-kilobase region and are functional in Escherichia coli[J]. Applied & Environmental Microbiology, 2004, 70(7): 4402-4407. |
| [35] |
Aislabie J, Bej A J, Lloyd N, et al. Characterization of Arthrobacter nicotinovorans, HIM, an atrazine-degrading bacterium, from agricultural soil New Zealand[J]. Fems Microbiology Ecology, 2005, 52(2): 279-286. DOI:10.1016/j.femsec.2004.11.012 |
| [36] |
Sajjaphan K, Heepngoen P, Sadowsky M J, et al. Arthrobacter sp.strain KU001 isolated from a Thai soil degrades atrazine in the presence of inorganic nitrogen sources[J]. Journal of Microbiology & Biotechnology, 2010, 20(3): 602-608. |
| [37] |
韩鹏, 洪青, 何丽娟, 等. 阿特拉津降解菌ADH-2的分离、鉴定及其特性研究[J]. 农业环境科学学报, 2009, 28(2): 406-410. HAN Peng, HONG Qing, HE Li-juan, et al. Isolation, identification and characterization of an atrazine-degrading bacteria ADH-2[J]. Journal of Agro-Environment Science, 2009, 28(2): 406-410. |
| [38] |
朱希坤, 李清艳, 蔡宝立. 节杆菌AD26的分离鉴定及其与假单胞菌ADP对阿特拉津的联合降解[J]. 农业环境科学学报, 2009, 28(3): 627-632. ZHU Xi-kun, LI Qing-yan, CAI Bao-li. Isolation and identification of Arthrobacter sp.AD26 and joint degradation of atrazine by Arthrobacter sp.AD26 and Pseudomonas sp.ADP[J]. Journal of Agro-Environment Science, 2009, 28(3): 627-632. |
| [39] |
Vaishampayan P A, Kanekar P P, Dhakephalkar P K. Isolation and characterization of Arthrobacter sp.strain MCM B-436, an atrazine-degrading bacterium, from rhizospheric soil[J]. International Biodeterioration & Biodegradation, 2007, 60(4): 273-278. |
| [40] |
代先祝, 胡江, 蒋建东, 等. 污染土壤中原位阿特拉津降解菌的分离和鉴定[J]. 土壤学报, 2006, 43(3): 467-472. DAI Xian-zhu, HU Jiang, JIANG Jian-dong, et al. Isolation and identification of in situ atrazine-degrading bacteria from contaminated soils[J]. Aeta Pedologica Sinica, 2006, 43(3): 467-472. DOI:10.11766/trxb200503160316 |
| [41] |
李明锐, 祖艳群, 陈建军, 等. 阿特拉津降解菌FM326(Arthrobacter sp.)的分离筛选、鉴定和生物学特性[J]. 农业环境科学学报, 2011, 30(11): 2242-2248. LI Ming-rui, ZU Yan-qun, CHEN Jian-jun. Isolation, screening and identification of atrazine-degrading bacterium FM326(Arthrobacter sp.)[J]. Journal of Agro-Environment Science, 2011, 30(11): 2242-2248. |
| [42] |
Zhang Y, Jiang Z, Cao B, et al. Metabolic ability and gene characteristics of Arthrobacter sp.strain DNS10, the sole atrazine-degrading strain in a consortium isolated from black soil[J]. International Biodeterioration & Biodegradation, 2011, 65(8): 1140-1144. |
| [43] |
Wang Q, Xie S. Isolation and characterization of a high-efficiency soil atrazine-degrading Arthrobacter sp.strain[J]. International Biodeterioration & Biodegradation, 2012, 71(4): 61-66. |
| [44] |
周宁, 王荣娟, 孟庆娟, 等. 寒地黑土中阿特拉津降解菌的筛选及降解特性[J]. 环境工程学报, 2008, 2(11): 1560-1563. ZHOU Ning, WANG Rong-juan, MENG Qing-juan, et al. Screening and identification of atrazine degrading bacteria and their degradation characteristics from black soil in cold aera[J]. Chinese Journal of Environmental Engineering, 2008, 2(11): 1560-1563. |
| [45] |
Yang C, Li Y, Zhang K, et al. Atrazine degradation by a simple consortium of Klebsiella sp.A1 and Comamonas sp.A2 in nitrogen enriched medium[J]. Biodegradation, 2010, 21(1): 97-105. DOI:10.1007/s10532-009-9284-9 |
| [46] |
Wang J, Zhu L, Liu A, et al. Isolation and characterization of an Arthrobacter sp.strain HB-5 that transforms atrazine[J]. Environmental Geochemistry & Health, 2011, 33(3): 259-266. |
| [47] |
李绍峰, 朱静, 李铁晶. 阿特拉津降解菌株的分离、鉴定及降解特性研究[J]. 环境科学, 2012, 33(9): 3214-3219. LI Shao-feng, ZHU Jing, LI Tie-jing. Isolation, identification and characterization of an atrazine degrading bacterium[J]. Environmental Science, 2012, 33(9): 3214-3219. |
| [48] |
闫彩芳, 娄旭, 洪青, 等. 一株阿特拉津降解菌的分离鉴定及降解特性[J]. 微生物学通报, 2011, 38(4): 493-497. YAN Cai-fang, LOU Xu, HONG Qing, et al. Isolation, identification and characterization of an atrazine-degrading strain[J]. Journal of Agro-Environment Science, 2011, 38(4): 493-497. |
| [49] |
刘春光, 杨峰山, 卢星忠, 等. 阿特拉津降解菌T3AB1的分离鉴定及土壤修复[J]. 微生物学报, 2010, 50(12): 1642-1650. LIU Chun-guang, YANG Feng-shan, LU Xing-zhong, et al. Isolation, identification and soil remediation of atrazine-degrading strain T3AB1[J]. Acta Microbiologica Sinica, 2010, 50(12): 1642-1650. |
| [50] |
Vibber L L, Pressler M J, Colores G M. Isolation and characterization of novel atrazine-degrading microorganisms from an agricultural soil[J]. Applied Microbiology & Biotechnology, 2007, 75(4): 921-928. |
| [51] |
Arbeli Z, Fuentes C. Prevalence of the gene trzN and biogeographic patterns among atrazine-degrading bacteria isolated from 13 Colombian agricultural soils[J]. Fems Microbiology Ecology, 2010, 73(3): 611-623. |
 2018, Vol. 37
2018, Vol. 37




