2. 农业农村部华南热带农业环境重点实验室, 广州 510642;
3. 华南农业大学资源环境学院生态学系, 广州 510642
2. Key Laboratory of Agroenvironment in the Tropics, Ministry of Agriculture and Rural Affairs, Guangzhou 510642, China;
3. Department of Ecology, College of Natural Resources and Environment, South China Agricultural University, Guangzhou 510642, China
农田间作系统氮素循环是国际、国内研究的热点问题,但目前的研究仅停留在间作作物氮素固定与转移等表观指标计算上[1-6],尚未深入到土壤微生物所驱动的生物固氮作用、氨化作用、硝化作用、反硝化作用等主要氮素循环过程[7],如何从复杂的土壤总微生物群落中分离出直接参与氮素循环的核心微生物及相关功能基因是亟待解决的问题。稳定性同位素核酸探针技术DNA-SIP(DNA-based stable isotope probing)是采用稳定同位素示踪复杂环境中微生物基因组DNA的分子生态学技术[8-9],利用13C或15N等稳定同位素示踪同化了标记底物的微生物作用者,将特定的物质代谢过程与微生物群落物种组成直接耦合,从而在群落水平上揭示间作系统下直接参与土壤氮、碳转化的核心功能微生物及其生理代谢的关键过程[10-12]。DNA-SIP技术的发展为农田间作系统中氮素循环的研究提供了新思路,可以利用稳定性同位素标记目标作物,进一步示踪并分离参与氮素循环相关的核心微生物,结合现代分子生物学技术深入分析其物种分布及生理代谢机制。
采用植物原位注射标记的方法可以直接示踪间作系统中氮素的流向,能够清楚地标记并分离参与氮素循环的核心微生物,是研究甘蔗//大豆间作系统土壤氮素循环的有效方法。目前DNA-SIP技术的研究中绝大多数采用13C标记底物培养环境样品来探索碳的循环和利用情况[13-16],尚未见15N原位标记探索甘蔗//大豆间作系统土壤氮素循环的研究报道。DNA-SIP技术的核心是超高速密度梯度离心分离稳定性同位素标记和非标记的DNA,如何鉴定超高速密度梯度离心后15N-DNA的富集位置是应用此项技术的关键。应用荧光实时定量PCR是鉴别超高速离心后标记DNA富集位置的最可靠技术之一[9]。本文通过室内盆栽试验,采用15N-DNA-SIP技术分离甘蔗//大豆间作系统中大豆根际土壤微生物总DNA,采用荧光定量PCR技术检测6个氮素循环关键功能基因在不同CsCl浮力密度层级中的基因丰度分布鉴定15N-DNA的富集位置,通过比较鉴定效果,筛选出有效鉴定甘蔗//大豆间作系统DNA-SIP中超高速离心后15N-DNA富集位置的指示功能基因,旨在为甘蔗//大豆间作系统氮素循环的研究提供一种新的技术方法。
1 材料和方法 1.1 试验材料供试甘蔗品种为“粤糖00-236”(Saccharum sinensis Roxb. cv. Yuetang 00-236),大豆品种为“上海青”(Glycine max L. cv. Shanghaiqing)。供试土壤取自于华南农业大学试验农场(23°8′N,113°15′E)试验田的长期定位试验小区,土壤类型为赤红壤,pH为5.90,有机质含量12.57 g·kg-1,碱解氮58.25 mg·kg-1,速效磷81.10 mg·kg-1,速效钾27.98 mg·kg-1。
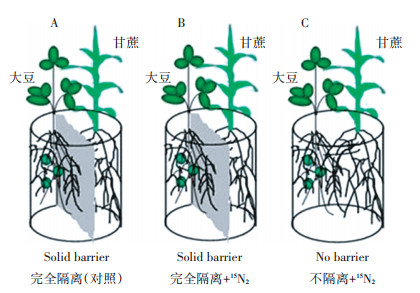
1.2 试验设计试验设置两种不同的根系隔离模式,第一种模式是将甘蔗和大豆的根系完全隔离,作物根系之间没有任何物质交换;第二种模式是甘蔗和大豆之间根系无隔离,作物根系之间物质可以自由完全交换。甘蔗和大豆之间根系无隔离的可视为甘蔗//大豆间作,完全隔离的甘蔗和大豆可视为单作甘蔗和单作大豆。使用(15NH4)2SO4分别标记完全分隔和无隔离盆栽大豆,甘蔗不做标记,以未标记单作大豆为参比对照,每个处理重复3次。试验种植模式如图 1所示。试验所用塑料盆规格为33.3 cm×22.3 cm×13.0 cm,完全分隔盆子中间使用3 mm厚塑料隔板沿宽度方向分隔为两室,每小室规格为33.3 cm×11.0 cm×13.0 cm,每个盆子底部打16个直径为4 mm的小孔以利于透气排水。
本试验于2017年7月27日至9月13日在广州华南农业大学生态农场温室内进行。土壤过2 mm筛,按体积比掺20%的小石子(Φ3~6 mm,121 ℃灭菌30 min),每盆装混匀后的土壤10 kg。每盆移栽甘蔗2株,大豆5株。植物生长期间每日定量浇水,保持土壤湿度在40%~65%之间,温度控制在25~30 ℃之间,每隔7 d调换一次盆子位置。
1.3 作物15N同位素标记本试验采用15N溶液叶柄注射的方法标记[18-19]。将15N丰度为99.14%的(15NH4)2SO4(上海化工研究院生产)配制成浓度为88 mmol·L-1的稀溶液,大豆始花期(R1,35 d)开始标记,每日使用10 μL的微量进样器(上海安亭微量进样器厂,上海)从大豆的叶柄或茎中注射10 μL的15N标记溶液,连续注射9 d,共注射90 μL的(15NH4)2SO4溶液。最后一次标记后6 d收获取样。
1.4 样品15N同位素丰度测定收获时将甘蔗、大豆植株分为地上部和根两部分装于牛皮纸袋105 ℃下杀青30 min,70 ℃烘至恒量,用高速万用粉碎机(天津泰斯特仪器有限公司,中国)磨碎,过0.18 mm筛;收集大豆根围土,室温风干后用研钵磨碎,过0.18 mm筛。采用同位素比值质谱仪(IRMS)Thermo Scientific Delta V Advantage(Thermo Fisher Scientific Inc.,美国)测定植物和土壤样品15N同位素丰度。
1.5 根际土微生物总DNA提取收获时用灭菌毛刷轻轻刷取紧贴于大豆根(≤2 mm)的土作为大豆根际土。采用PowerMax® Soil DNA Isolation Kit 12988-10(MoBio Laboratories Inc.,Carlsbad,CA,USA)提取大豆根际土中微生物DNA。洗脱后分装3 μL用于检测DNA质量和浓度,其他部分DNA存于-20 ℃备用。
使用NanoDropTM 2000c微量紫外分光光度计(Thermo Scientific,DK)对所提取的DNA浓度和纯度进行测定,当A260/A280处于1.8~2.0,A260/A230处于2.0~2.2时可认为目标DNA样品纯度较好,可用于下游试验。
1.6 超高速密度梯度离心 1.6.1 DNA-CsCl溶液制备参考Neufeld等[21]的方法,取适量CsCl(AR)溶于pH=8.0的TE溶液,通过AR200 Digital(Reichert,Inc.,Buffalo,NY,USA)手持折光仪测定折光率,将溶液浮力密度调节至1.780~1.800 g·mL-1之间作为母液,取适量母液加入TE,将浮力密度调节为1.728 g·mL-1作为补充液。
折光率与浮力密度的关系如下:

|
式中:BD为溶液浮力密度;RI为溶液折光率[22]。
将6 μg大豆根际土DNA样品,配制成浮力密度为1.728 g·mL-1的DNA-CsCl离心液。用5 mL的无针注射器将调配好的DNA-CsCl离心液转移至超高速离心试管(Reorder no. 344058,Beckman Coulter,Inc. USA)中,使用浮力密度为1.728 g·mL-1的CsCl溶液尽可能充满离心试管且无气泡产生,以防止超高速离心过程中试管爆裂,将两个对称位置离心试管称质量配平,使其质量之差小于±0.01 g,随后用热封仪(Beckman Coulter,cat. no.349646)将离心管封好,确保不漏液。
1.6.2 等密度梯度超高速离心本试验选用20 ℃,178 000×g离心力条件下离心48 h,使用Optima L-100 XP超速离心机(Beckman Coulter Incorporated,Brea,CA,USA),NVT 100离心转子(Beckman Coulter Incorporated,Brea,CA,USA)。
1.7 不同浮力密度DNA分层与纯化将离心液均等分为20层到2 mL离心管,每管体积约为280 μL,对每层溶液用AR200 Digital(Reichert,Inc.,Buffalo,NY,USA)手持折光仪测定RI值,根据预试验经验,DNA离心后处于RI为1.400 0~1.404 0的浮力密度层级,因此保存该层级的DNA离心液做进一步DNA纯化。纯化所用试剂为EZNA MicroElute DNA clean-up kit纯化试剂盒(Omega Bio-Tek,Norcross,GA,USA)。
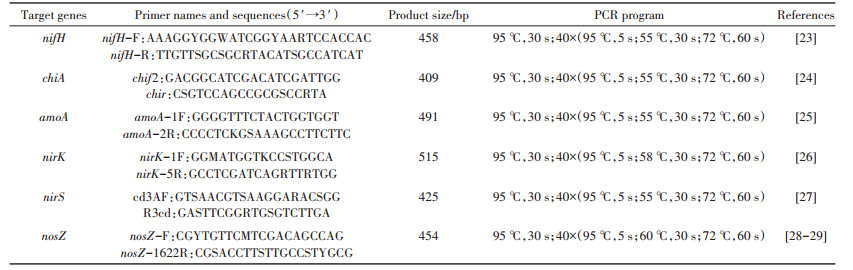
1.8 功能基因qPCR引物选择本试验选择检测调控固氮过程(nifH)、有机氮矿化过程(chiA)、硝化过程(amoA)和反硝化过程(nirK、nirS和nosZ)的关键基因,引物信息及反应程序如表 1。
|
|
表 1 qPCR引物及反应程序 Table 1 Primers and qPCR conditions used in the experiment |
反应结束后添加溶解曲线程序(95 ℃,15 s;60 ℃,60 s;95 ℃,30 s;60 ℃,15 s)。
1.9 qPCR标准曲线制备 1.9.1 功能基因片段的扩增以对照组大豆根际土DNA为模板,分别用6对引物进行相应功能基因片段扩增。采用Recombinant Taq DNA Polymerase(TaKaRa Bio Inc.)在Bio-Rad S1000 PCR系统(Bio-Rad,Foster,CA,USA)扩增,PCR反应体系为25 μL:2.5 μL 10×PCR Buffer,2.5 μL dN TP MIX,1 μL DNA模板,引物上游和下游各1 μL,ddH2O 16.8 μL,Taq酶0.2 μL。PCR反应程序为:95 ℃,30 min→35×(95 ℃,30 s;Tm;72 ℃,90 s)→ 72 ℃,10 min,退火温度按照表 1中相应引物Tm设定。反应结束后,将扩增产物经1.2 %的琼脂糖凝胶电泳观察,在紫外灯下对目标条带切胶,使用TaKaRa MiniBEST Agarose Gel DNA Extraction Kit Ver. 4.0(TaKaRa Bio Inc.)胶回收试剂盒对PCR产物进行纯化回收。
1.9.2 载体连接转化使用pMDTM 18-T Vector Cloning Kit(TaKaRa Bio Inc.)试剂盒将回收纯化后的PCR产物与pMD18载体连接,连接反应体系为:1 μL pMD18-T Vector,纯化DNA 4 μL,SolutionⅠ 5 μL,总体积10 μL。
1.9.3 质粒提取及其拷贝数计算使用MiniBEST Plasmid Purification Kit Ver. 4.0(TaKaRa Bio Inc.)质粒纯化试剂盒对菌液进行质粒提取与纯化。使用NanoDropTM 2000c微量紫外分光光度计(Thermo Scientific,DK)测定质粒DNA浓度和纯度,按照如下公式[30]计算质粒拷贝数:

|
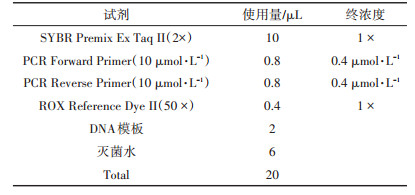
以等密度梯度超高速离心分层、纯化后的DNA为模板,使用SYBR® Premix Ex TaqTM Ⅱ(TaKaRa Biotechnology,Otsu,Shiga,Japan)Real Time PCR的专用试剂盒在Applied Biosystems 7500 / 7500 Fast Real-Time PCR System(Thermo Fisher Scientific Inc. UK)上进行绝对定量PCR分析,检测各功能基因在不同浮力密度层级的拷贝数。为了准确比对各处理不同浮力密度层级DNA中功能基因拷贝数,进行qPCR操作时,对15N标记大豆单作、15N标记大豆//甘蔗间作以及不含15N标记的大豆单作对照组中各浮力密度层级DNA在96孔PCR板上同步进行点样,每个样品3次重复。标准曲线使对应功能基因的克隆重组质粒进行10倍稀释,8个稀释梯度,每个梯度3个重复,选用无菌去离子水作为阴性对照,按照表 1设置扩增程序,扩增体系如表 2。待PCR结束后根据标准曲线的浓度计算出样品中的基因拷贝数。最终呈现数据使用基因相对丰度表示,即相同处理中各层级DNA基因拷贝数与所有层级中最大基因拷贝数的比值[31]。
|
|
表 2 功能基因qPCR扩增体系 Table 2 qPCR reaction system of functional gene used in the experiment |
试验所采集的数据使用Microsoft office excel 2013和SPSS 23.0进行相关分析与作图,处理之间的平均值差异采用One-Way ANOVA单因素方差分析,P<0.05表示显著差异。
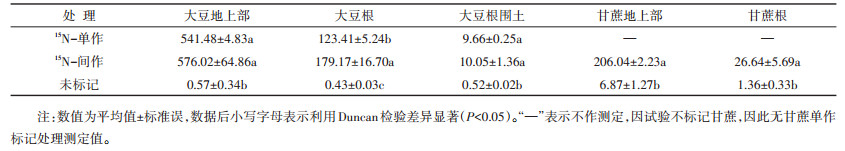
2 结果与分析 2.1 植株和土壤样品中15N同位素丰度不同处理下植株和土壤样品同位素丰度差异显著(表 3),标记单作处理和间作处理的大豆地上部、大豆根和大豆根围土样品均检测到较高的15N丰度,且都显著高于对照组。间作处理的15N同位素丰度高于单作处理。大豆地上部、大豆根和大豆根围土中15N同位素丰度依次递减,大豆地上部最高,大豆根围土中最低。同时在15N标记间作处理中的甘蔗根和甘蔗地上部中也检测到较高的15N同位素丰度,且标记处理的丰度显著高于对照处理。
|
|
表 3 不同处理中植株及土壤15N同位素丰度 Table 3 δ15N of plant and soil in different treatments |
以各处理的大豆根际土壤微生物DNA为模板,采用功能基因特异性引物定量6个氮素循环功能基因在不同CsCl浮力密度层级中的拷贝数,将原始基因拷贝数据转化为功能基因相对丰度后作图分析。不同处理和标记方式下功能基因相对丰度随着浮力密度增大均呈现出先增大后减小的趋势,形状如抛物线,各功能基因在不同浮力密度层级的分布差异较大,但都主要集中于峰值附近(图 2)。从不同密度梯度分层样品中功能基因的相对丰度显示,调控固氮过程的nifH基因(图 2a)和调控硝化过程amoA基因(图 2c)无论是在甘蔗//大豆间作还是大豆单作处理,其相对丰度高峰值位置均与参比对照处理存在明显的偏移,15N标记的两种种植模式中nifH基因相对丰度最高峰出现在1.723 g·mL-1的浮力密度层,而参比对照的nifH基因相对丰度高峰出现在1.713 g·mL-1的浮力密度层,两个层级密度差约为0.010 g·mL-1;amoA基因在15N标记的两种种植模式中相对丰度高峰出现在1.718 g·mL-1的浮力密度层,而参比对照的amoA基因相对丰度高峰出现在1.713 g·mL-1的浮力密度层,两个层级密度差为0.005 g·mL-1;调控有机氮矿化过程的chiA基因(图 2b)相对丰度高峰在15N标记的大豆单作处理中与参比对照组存在偏移,15N单作处理的chiA基因相对丰度高峰出现在浮力密度为1.718 g· mL-1的层级,参比对照组中基因相对丰度高峰在浮力密度为1.713 g·mL-1的层级,两个层级密度差为0.005 g·mL-1,而15N标记的间作处理与参比对照组中基因相对丰度高峰重叠;调控反硝化过程的nirK(图 2d)、nirS(图 2e)和nosZ(图 2f)基因中,根据相对丰度高峰位置显示,参比对照组基因相对丰度高峰所对应浮力密度高于15N标记的单作和间作处理,与预期判断结果相反。
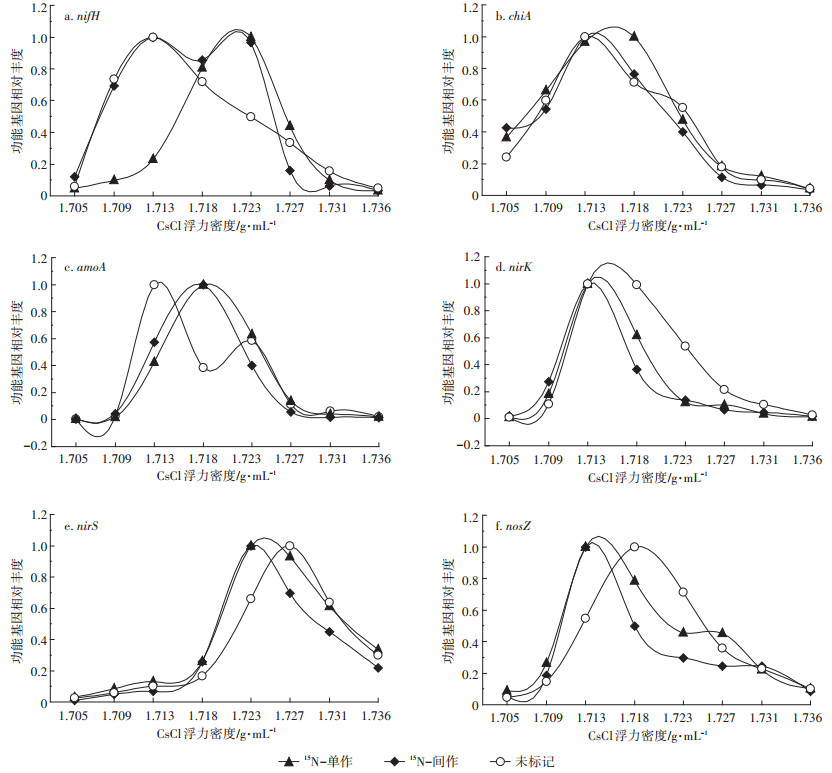
|
图中浮力密度及功能基因相对丰度数值均为3次重复平均值 图 2 不同浮力密度层级中功能基因相对丰度的分布 Figure 2 Distribution of the relative abundance of functional gene in CsCl gradient |
通过样品同位素丰度测定结果可知,标记处理中15N丰度显著升高,大豆叶柄注射标记的15N被大豆同化吸收后参与其新陈代谢,通过根系分泌物、细胞死亡凋落等方式释放到土壤中,进入土壤氮素循环,经土壤微生物吸收转化后,被间作的甘蔗吸收利用,在甘蔗//大豆间作系统中发生氮素转移,因此,证明15N成功标记到作物、土壤及土壤微生物,并被作物、土壤及土壤微生物所富集。
使用DNA-SIP技术分离被标记的土样微生物DNA,并采用qPCR检测不同浮力密度层级DNA中氮素循环功能基因的相对丰度分布,基因相对丰度高的密度层级代表着DNA富集程度也高,因此可根据基因相对丰度峰的位置判断DNA在离心液中富集位置。与参比对照组N-DNA相比,被标记的15N-DNA理论上应集中于浮力密度更高的层级,通过对比两者在不同浮力密度梯度区带中的位移,也即通过对比对照处理与标记处理所对应的功能基因相对丰度高峰的位置鉴别未标记N-DNA与15N-DNA分离情况,若对照组与标记组中功能基因相对丰度高峰有偏移,则代表分离成功,且基因相对丰度高峰所对应的高浮力密度层级即为15N-DNA富集区带,反之则分离失败。
nifH基因和amoA基因在甘蔗//大豆间作和大豆单作处理中,15N标记处理的基因相对丰度高峰值位置均与参比对照处理存在明显的偏移,因此说明nifH基因和amoA基因能够清晰判断甘蔗//大豆间作和大豆单作DNA-SIP中15N-DNA富集位置。chiA基因相对丰度高峰仅在15N标记的大豆单作处理中与参比对照组存在偏移,而15N标记的间作处理与参比对照组中基因相对丰度高峰重叠,因此说明chiA基因只能够鉴别单作处理下DNA-SIP中15N-DNA富集位置,造成这种结果的原因可能是注射相同量的15N溶液,单作处理的根室体积较间作小,根际土壤少,因此土壤中15N浓度较高,容易被富集。nirK、nirS和nosZ在14N对照组基因相对丰度高峰所对应浮力密度高于15N标记的单作和间作处理,与预期判断结果相反,Buckley等[32]用15N2-DNA-SIP研究甲烷氧化菌时也曾出现类似的结果,因此调控反硝化过程的nirK、nirS和nosZ基因不能够有效鉴别间作系统中DNA-SIP的15N-DNA富集位置。
由于室内盆栽试验严格控制了土壤水热条件,因此盆栽土壤中主要发生固氮作用、氨化作用和硝化作用等生态过程,反硝化作用相对较弱,进而调控反硝化过程的功能基因丰度相对低,因此标记的15N进入土壤后,主要被固氮作用、氨化作用和硝化作用等的微生物所同化,进而调控该过程的功能基因富集了更多的15N。在本试验中,注射的15N溶液进入大豆体内后,会很容易被共生固氮菌同化吸收,当根系分泌物或凋落物进入土壤后,被有机氮矿化微生物矿化分解,被分解后的无机氮会进一步发生硝化作用,因此调控这些生态过程的nifH基因、amoA基因和chiA基因相对容易被标记,进而在DNA-SIP的分层中检测相对灵敏。而调控反硝化过程的功能基因丰度较低,因此nirK、nirS和nosZ基因富集到15N的量较少,所以在DNA-SIP分层中检测灵敏度相对低,不易检测。
4 结论本文通过qPCR技术定量检测了调控氮素循环的功能基因在超高速离心后不同浮力密度层级中的丰度分布,通过对比分析筛选出nifH基因和amoA基因作为指示基因,可准确鉴定甘蔗//大豆间作系统中DNA-SIP的15N-DNA富集位置,为甘蔗//大豆间作系统中土壤氮素循环核心微生物的分离及其下游研究工作的开展提供了新方法。在同位素原位标记分析的基础上结合现代分子生物学技术揭示农田间作系统中参与氮素循环核心微生物的多样性,将为科学揭示农田氮素循环过程的微生物调控机理奠定技术基础。
| [1] |
Chu G X, Shen Q R, Cao J L. Nitrogen fixation and N transfer from peanut to rice cultivated in aerobic soil in an intercropping system and its effect on soil N fertility[J]. Plant & Soil, 2004, 263(1): 17-27. |
| [2] |
Li Y Y, Yu C B, Cheng X, et al. Intercropping alleviates the inhibitory effect of N fertilization on nodulation and symbiotic N2 fixation of faba bean[J]. Plant and Soil, 2009, 323(1/2): 295-308. |
| [3] |
Fan F, Zhang F, Song Y, et al. Nitrogen fixation of faba bean (Vicia faba L.) interacting with a non-legume in two contrasting intercropping systems[J]. Plant & Soil, 2006, 283(1/2): 275-286. |
| [4] |
Høgh-Jensen H. The nitrogen transfer between plants:An important but difficult flux to quantify[J]. Plant & Soil, 2006, 282(1/2): 1-5. |
| [5] |
Schipanski M E, Drinkwater L E, Russelle M P. Understanding the variability in soybean nitrogen fixation across agroecosystems[J]. Plant & Soil, 2010, 329(1/2): 379-397. |
| [6] |
雍太文, 杨文钰, 向达兵, 等. 小麦/玉米/大豆和小麦/玉米/甘薯套作对土壤氮素含量及氮素转移的影响[J]. 作物学报, 2012, 38(1): 148-158. YONG Tai-wen, YANG Wen-yu, XIANG Da -bing, et al. Effect of wheat/maize/soybean and wheat/maize/sweet potato relay strip intercropping on soil nitrogen content and nitrogen transfer[J]. Acta Agronomica Sinica, 2012, 38(1): 148-158. DOI:10.3969/j.issn.1000-2561.2012.01.030 |
| [7] |
贺纪正, 张丽梅. 土壤氮素转化的关键微生物过程及机制[J]. 微生物学通报, 2013, 40(1): 98-108. HE Ji-zheng, ZHANG Li-mei. Key processes and microbial mechanisms of soil nitrogen transformation[J]. Microbiology China, 2013, 40(1): 98-108. |
| [8] |
Peoples M B, Chalk P M, Unkovich M J, et al. Can differences in 15N natural abundance be used to quantify the transfer of nitrogen from legumes to neighbouring non-legume plant species?[J]. Soil Biology & Biochemistry, 2015, 87: 97-109. |
| [9] |
贾仲君. 稳定性同位素核酸探针技术DNA-SIP原理与应用[J]. 微生物学报, 2011, 51(12): 1585-1594. JIA Zhong-jun. Principle and application of DNA-based stable isotope probing[J]. Acta Microbiologica Sinica, 2011, 51(12): 1585-1594. |
| [10] |
Dumont M G, Murrell J C. Stable isotope probing-linking microbial identity to function[J]. Nat Rev Microbiol, 2005, 3(6): 499-504. DOI:10.1038/nrmicro1162 |
| [11] |
Bernard L, Mougel C, Maron P A, et al. Dynamics and identification of soil microbial populations actively assimilating carbon from 13C-labelled wheat residue as estimated by DNA-and RNA-SIP techniques.[J]. Environmental Microbiology, 2007, 9(3): 752-764. DOI:10.1111/emi.2007.9.issue-3 |
| [12] |
Venter Z S, Scott S L, Strauss J, et al. Increasing crop diversity increased soil microbial activity, nitrogen-sourcing and crop nitrogen, but not soil microbial diversity[J]. South African Journal of Plant & Soil, 2017, 34: 1-8. |
| [13] |
Chen S C, Duan G L, Ding K, et al. DNA stable-isotope probing identifies uncultivated members of Pseudonocardia associated with biodegradation of pyrene in agricultural soil[J]. Fems Microbiology Ecology, 2018, 94(3): 1-10. |
| [14] |
Feng Y, Lin X, Zhu J, et al. A phototrophy-driven microbial food web in a rice soil[J]. Journal of Soils & Sediments Protection Risk Assessment & Rem, 2011, 11(2): 301-311. |
| [15] |
钱明媚, 肖永良, 彭文涛, 等. 免耕水稻土固定CO2自养微生物多样性[J]. 中国环境科学, 2015, 35(12): 3754-3761. QIAN Ming-mei, XIAO Yong-liang, PENG Wen-tao, et al. Diversities of autotrophic CO2-fixing microbes in no-tillage paddy soils[J]. China Environmental Science, 2015, 35(12): 3754-3761. DOI:10.3969/j.issn.1000-6923.2015.12.029 |
| [16] |
郑燕, 贾仲君. 基于核酸DNA/RNA同位素示踪技术的水稻土甲烷氧化微生物研究[J]. 土壤学报, 2016, 53(2): 490-501. ZHENG Yan, JIA Zhong-jun. The application of biomarker genes for DNA/RNA-stable isotope probing of active methaneotrophs responsible for aerobic methane oxidation in six paddy soils[J]. Acta Pedologica Sinica, 2016, 53(2): 490-501. |
| [17] |
Meng L, Zhang A, Wang F, et al. Arbuscular mycorrhizal fungi and rhizobium facilitate nitrogen uptake and transfer in soybean/maize intercropping system[J]. Frontiers in Plant Science, 2015, 6: 339. |
| [18] |
Frey B, Schuepp H. A role of vesicular-arbuscular (VA) mycorrhizal fungi in facilitating interplant nitrogen transfer[J]. Soil Biology & Biochemistry, 1993, 25(6): 651-658. |
| [19] |
肖焱波.豆科/禾本科间作体系中养分竞争和氮素转移研究[D].北京: 中国农业大学, 2003. XIAO Yan-bo. Interspecific competition for nutriens and nitrogen transfer between the intercropped legume and cereal[D]. Beijing: China Agriculture University, 2003. http://cdmd.cnki.com.cn/Article/CDMD-10019-2003092777.htm |
| [20] |
Tran-Nguyen L T T, Schneider B. Cesium chloride-bisbenzimide gradients for separation of phytoplasma and plant DNA[J]. Methods Mol Biol, 2013, 938(938): 381-393. |
| [21] |
Neufeld J D, Vohra J, Dumont M G, et al. DNA stable-isotope probing[J]. Nature Protocols, 2007, 2(4): 860-866. DOI:10.1038/nprot.2007.109 |
| [22] |
Gore N R, Wray J L. Leucine:tRNA Ligase from cultured cells of nicotiana tabacum var. Xanthi:Evidence for de novo synthesis and for loss of functional enzyme molecules[J]. Plant Physiology, 1978, 61(1): 20-24. DOI:10.1104/pp.61.1.20 |
| [23] |
Rösch C, Mergel A, Bothe H. Biodiversity of denitrifying and dinitrogen-fixing bacteria in an acid forest soil[J]. Applied & Environmental Microbiology, 2002, 68(8): 3818-3829. |
| [24] |
Williamson N, Brian P, Wellington E M H. Molecular detection of bacterial and streptomycete chitinases in the environment[J]. Antonie Van Leeuwenhoek, 2000, 78(3): 315-321. |
| [25] |
Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker:Molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied & Environmental Microbiology, 1997, 63(12): 4704-4712. |
| [26] |
Braker G, Fesefeldt A, Witzel K P. Development of PCR primer systems for amplification of nitrite reductase genes (nirK and nirS) to detect denitrifying bacteria in environmental samples[J]. Appl Environ Microbiol, 1998, 64(10): 3769-3775. |
| [27] |
Michotey V, Méjean V, Bonin P. Comparison of methods for quantification of cytochrome cd 1-denitrifying bacteria in environmental marine samples[J]. Applied and Environmental Microbiology, 2000, 66(4): 1564-1571. DOI:10.1128/AEM.66.4.1564-1571.2000 |
| [28] |
Kloos K, Mergel A, Rösch C, et al. Denitrification within the genus Azospirillum and other associative bacteria[J]. Australian Journal of Plant Physiology, 2001, 28(9): 991-998. |
| [29] |
Throbäck I N, Enwall K, Jarvis A, et al. Reassessing PCR primers targeting nirS, nirK and nosZ genes for community surveys of denitrifying bacteria with DGGE[J]. Fems Microbiology Ecology, 2004, 49(3): 401-417. DOI:10.1016/j.femsec.2004.04.011 |
| [30] |
Vaitomaa J, Rantala A, Halinen K, et al. Quantitative Real-Time PCR for determination of microcystin synthetase E copy numbers for microcystis and anabaena in lakes[J]. Applied & Environmental Microbiology, 2003, 69(12): 7289-7297. |
| [31] |
Zhang L M, Hu H W, Shen J P, et al. Ammonia-oxidizing archaea have more important role than ammonia-oxidizing bacteria in ammonia oxidation of strongly acidic soils[J]. Isme Journal, 2012, 6(5): 1032-1045. DOI:10.1038/ismej.2011.168 |
| [32] |
Buckley D H, Huangyutitham V, Hsu S F, et al. 15N2-DNA-stable isotope probing of diazotrophic methanotrophs in soil[J]. Soil Biology & Biochemistry, 2008, 40(6): 1272-1283. |
 2019, Vol. 38
2019, Vol. 38