2. 天津大学化工学院, 天津 300072
2. School of Chemical Engineering and Technology, Tianjin University, Tianjin 300072, China
规模化、集约化的畜禽养殖业发展产生了大量养殖废水,相对固体粪便来说,养殖废水处理和利用率极低,严重制约了畜禽养殖业的可持续发展。因养殖废水含丰富的腐植酸等有机质和氮、磷等植物营养元素[1-2],直接排放会对环境造成水体富营养化的污染。在我国生态型畜禽养殖业发展的指导下,将养殖废水进行厌氧生物降解处理,产生的沼液养分全面作为一种优质的有机肥料源以肥水方式回归土壤,既有效利用养殖废水减少资源浪费,又避免直接排放养殖废水对环境的污染,是最佳的废水处理和利用途径,在农业生产上具有很好的应用前景[3]。因此,在提倡生态农业的今天,如何以沼液作为水肥安全高效地应用于农业生产,成为众多学者的研究方向[4-5]。
反硝化过程与土壤氮素损失和温室气体排放密切相关,是施肥和土壤研究中的重点问题[6]。有研究者估计,全球70%的N2O排放来自农田,其中农业生态系统的排放量约占25%[7];此外,反硝化作用致使土壤丧失20%~30%的氮肥,是土壤肥力下降的重要原因[8]。反硝化是在硝酸盐还原酶、亚硝酸盐还原酶、NO还原酶和N2O还原酶连续催化下将NO3-还原为N2的过程。其中亚硝酸盐还原酶(Nir)是反硝化作用中最关键的一步反应,因而其编码基因(nir)在反硝化研究中被广泛用作分子标记[9]。亚硝酸还原酶分为cd1-亚硝酸还原酶和Cu-亚硝酸还原酶两种,分别由nirS和nirK基因编码,两者不能共存于一种微生物中[10],nirS基因在反硝化菌中的存在比nirK更广泛[10]。nirK基因存在于许多亲缘关系较远的菌株中,且分子量变化较大;而含有nirS基因的细菌以假单胞菌占优势,且在不同菌株中分子大小相似,形态结构相对保守[11]。由nosZ基因编码的N2O还原酶催化N2O转化成N2,是反硝化过程的最后一步,对于减少N2O的排放意义重大[12-13]。
本研究以华北地区小麦-玉米轮作大田为研究对象,采用T-RFLP技术分析了不同牛场肥水浓度和灌溉次数下0~20 cm以及20~40 cm土层中nirS和nosZ群落结构及多样性变化,为牛场肥水的合理施用提供科学依据。
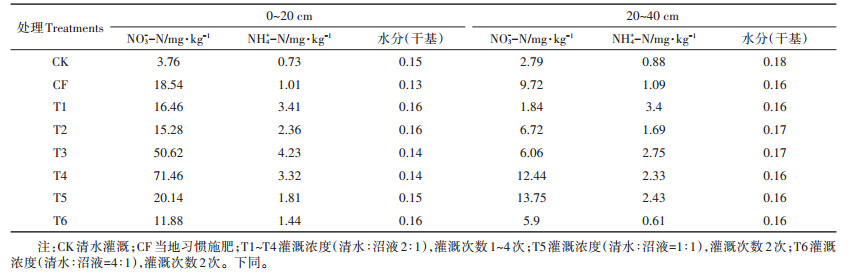
1 材料与方法 1.1 试验区概况试验于2012年10月至2015年6月进行,采样地点为河北省保定市徐水县梁家营长期定位牛场肥水灌溉试验田(115°32′6.67″E,38°56′43.63″N),土壤类型为潮褐土。徐水县位于太行山东麓,河北省中部,属暖温带季风型大陆性气候,四季分明,光照充足,海拔1200~2768 m,年平均气温12.3 ℃,年无霜期平均184 d,年均降水量546.9 mm,年日照时数平均2 744.9 h,属华北平原典型农业种植区。冬小麦-夏玉米轮作为当地主要的农业种植方式,当年10月上旬种植冬小麦,次年6月中旬收获小麦,约一周后种植夏玉米,当年9月底收获,玉米秸秆用于奶牛的饲养。该地区奶牛养殖业发达,奶牛场41座,奶牛存栏数达到2.5×104头,每年排放废水5×105 t。试验地种植前耕层土壤有机质质量分数24.5 g·kg-1、pH值7.76、全氮质量分数1.39 g·kg-1、硝态氮质量分数13.09 mg· kg-1、铵态氮质量分数2.24 mg·kg-1、速效磷质量分数64.19 mg·kg-1。试验土壤主要理化性质见表 1。
|
|
表 1 试验土壤基本性质 Table 1 Soil basic properties |
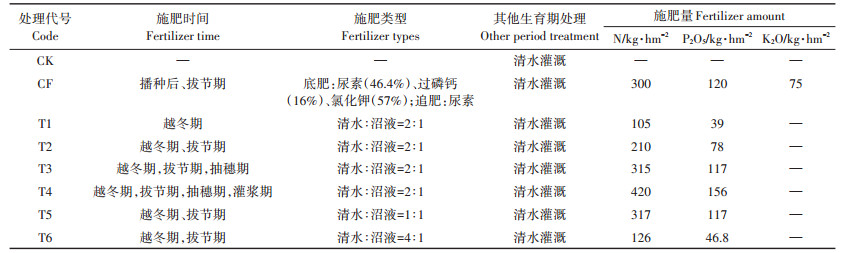
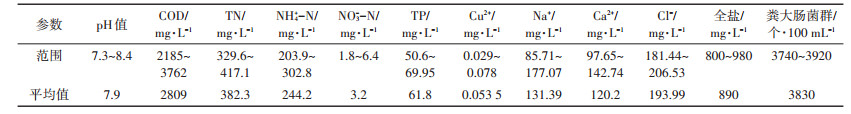
试验共设8个处理,表 2为各处理下的施肥时间、类型及成分。每处理设置3次重复,24个田间小区采用完全随机区组排列。每个小区长9 m,宽6 m,面积54 m2。试验小区灌溉采用畦灌,根据《华北地区冬小麦公顷产量6000 kg(亩产400 kg)栽培技术规程》(NY/T 205—1992),计算冬小麦需水量,所有处理灌水定额均为830 m3·hm-2,灌水量利用超声波流量计计量,灌溉误差1%以内。冬小麦全生育期灌水4次,玉米生育期灌水1次。供试冬小麦品种为济麦22、玉米为农大221。试验灌溉用牛场肥水为经过厌氧处理的牛粪尿和挤奶车间冲洗水,经检测符合《农田灌溉水质标准》(GB 5084—2005),表 3为厌氧处理后的灌溉肥水原液成分及含量。
|
|
表 2 各个处理施肥量 Table 2 Fertilizer amount in treatments |
|
|
表 3 灌溉肥水原液成分及含量 Table 3 The composition and content of cattle fertilizer biogas slurry |
土壤样品的采集:样品采自小麦收获期(2015年6月12日),采集深度为0~20、20~40 cm两个土层。采用五点取样法,每个实验小区处理中采集5个土样,将5点采集的土壤混合为1个样品。田间采集的土样保存于保鲜盒内并迅速送至实验室,于实验室内将土样去除根系、杂草等杂质后混匀,-20 ℃冷冻保存用于土壤微生物分析。
1.3 分析方法 1.3.1 土壤总DNA的提取土壤DNA的提取采用FastDNA SPIN Kit For Soil(MP Biomedicals,LLC)试剂盒方法。称取500 mg的土壤样品,按试剂盒提供的操作步骤进行,以FastPrep® Instrument进行细胞破碎处理,速度为6 m·s-1,时间40 s。提取的DNA在0.7%的琼脂糖凝胶中进行电泳检测。DNA保存于-20 ℃的冰箱中备用。
1.3.2 反硝化菌群落结构分析反硝化菌群落结构分析采用末端限制性酶切片段长度多态性方法分析(Terminal restriction fragment length polymorphism,T-RFLP),利用反硝化菌nirS、nosZ基因特异引物进行PCR扩增(表 4)。PCR产物用Mini BEST DNA Fragment Purification Kit VER 4.0(TaKaRa)试剂盒进行纯化回收,回收后的目的基因经限制性内切酶HhaI(TaKaRa)酶切分析。酶切后所得产物送至生工生物工程(上海)股份有限公司进行毛细管电泳检测。将获得的T-RFLP数据继续进行处理,去除结果中小于50 bp及相对丰度小于1%的酶切片段。
|
|
表 4 PCR反应中的引物及反应条件 Table 4 Primers and PCR conditions used for the PCR amplification |
将各处理下的PCR扩增产物均匀混合,经切胶回收后连接至pMD19®-T Vector质粒(Takara,大连)、转入大肠杆菌JM109感受态细胞DH5a内,37 ℃过夜培养,经蓝白斑筛选,挑取约100个阳性克隆子送至生工生物工程(上海)股份有限公司进行测序分析,构建克隆文库。将测序所得序列利用Vector NTI 11.5.1软件分析检查去除嵌合及错误序列后,在GeneBank数据库(http://www.ncbi.com)中进行Blast比对。
1.3.4 数据处理群落结构分析通过计算3个群落多样性指数(Diversity index):香农维纳指数(Shannon-Wiener index)、辛普森优势度指数(Simpson index)、均匀性指数(Pielou index)。
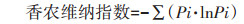
|
(1) |
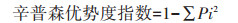
|
(2) |
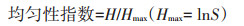
|
(3) |
式中:Pi表示某个峰的峰面积占该样点所有峰的总面积的比例;S表示T-RFs(T-RFs表示末端限制性片段)的个数。T-RFLP片段分布的影响的显著性采用SPSS 17.0(IBM SPSS)单因素方差分析检验,并进行多重比较。利用CANOCO 4.5(Ter Braak & Smilauer,2012)基于T-RFLP图谱中酶切片段的数值进行两土层中反硝化细菌群落结构的主成分分析(PCA)。系统发育分析采用MEGA 5.0(Molecular Evolutionary Genetics Analysis),根据BLAST同源性比对的结果,从核酸数据库中下载同源性高的不同分类来源的代表性nirS、nosZ序列,用邻接法(Neighbor-joining,NJ)构建nirS、nosZ反硝化细菌的系统发育树。
2 结果与分析 2.1 不同施肥处理对两土层nirS型反硝化菌群落的影响利用反硝化细菌nirS基因特异引物对两土层反硝化细菌群落结构进行T-RFLP分析,结果如图 1所示。经HhaI酶切后,0~20 cm和20~40 cm土层分别得到22种和27种片段长度,两土层中都存在着丰富的nirS型反硝化细菌基因资源。0~20 cm土层中nirS基因酶切片段68、83、102、112、160、166、169 bp和206 bp占全部片段百分比含量的75.60%~83.26%,为主要优势片段。不同肥水处理对0~20 cm土层中nirS型反硝化菌相对丰度均无显著差异,说明0~20 cm土层中nirS型反硝化细菌对施肥种类、肥水浓度及灌溉次数等不同处理条件不敏感。20~40 cm土层中nirS基因代表的反硝化微生物群落组成在各施肥处理下发生变化,经HhaI酶切后均得到6个主要片段68、98、112、160、166 bp和169 bp,占全部片段百分比含量的46.99%~60.85%,说明这些片段所代表的nirS型反硝化细菌在土壤中占优势。不同处理下6个主要片段相对丰度未发生明显变化,但CF、T1、T2、T4和T5处理下出现片段58 bp且相对丰度较高,片段185 bp在T3处理下相对丰度显著增加,片段83 bp比例在T4处理下相对丰度显著增加,这些片段均上升为优势菌种;T1和T2处理下片段317 bp相对丰度明显增加;T2、T4和T6处理下片段126 bp相对丰度显著降低。
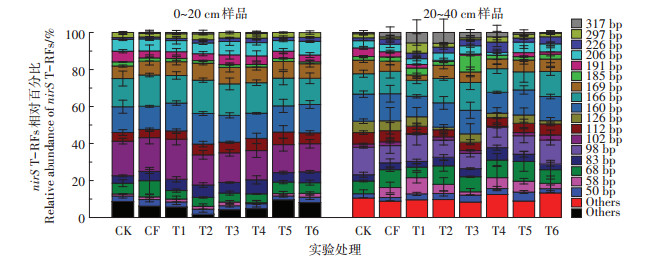
|
Others表示相对丰度小于5%的酶切片段。下同 图 1 不同处理条件下nirS型反硝化菌的T-RFLP图谱 Figure 1 T-RFLP fingerprints of nirS gene from soils under different regimes |
比较两土层nirS型反硝化菌群落组发现,0~20 cm土层中片段102 bp相对丰度在各处理下平均为17.13%,显著高于20~40 cm土层(1.55%);20~40 cm的土层中出现了0~20 cm土层中没有的76、91、98、105、126、235 bp新片段,其中98 bp和126 bp在各处理下的相对丰度较高,分别占11.31%、3.91%。上述T-RFLP结果表明,nirS型反硝化细菌群落结构在0~ 20、20~40 cm土层中存在一定的垂直分布差异,且0~ 20 cm土层中nirS型反硝化细菌群落结构对施肥种类、肥水浓度及灌溉次数不敏感,而20~40 cm土层中nirS型反硝化细菌群落结构对施肥条件敏感性提高。
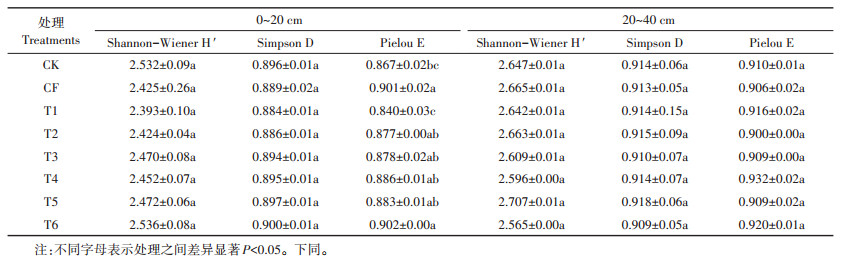
对不同处理下两土层中nirS型反硝化菌群落多样性指数分析(表 5)发现,无论是0~20 cm土层还是20~40 cm土层,大多数处理下nirS型反硝化菌的Shannon-Wiener H′指数、Simpson D指数和Pielou E指数没有显著的差别,表明实验条件下施肥种类及方式(肥水灌溉)对nirS基因多样性影响不显著。
|
|
表 5 不同施肥水平处理下两土层nirS基因多样性指数 Table 5 The diversity index of soil nirS denitrifying bacteria under different fertilizer treatments |
根据nirS基因T-RFs数据进行主成分分析(图 2)发现,在0~20 cm土层中,第一主成分(PC1)解释了37.2%的物种组成变化,第二主成分(PC2)解释了22.2%的物种组成。其中,CF与CK较为接近,而其他牛场肥水处理点与CK相距较远,说明肥水处理组对0~20 cm土层中nirS群落结构影响比常规施肥CF更大。在20~40 cm土层中,第一主成分(PC1)解释了31.0%的物种组成变化,第二主成分(PC2)解释了25.1%的物种组成。所有处理点均远离CK,其中T5、T6与CF较为接近,说明施肥处理使20~40 cm土层中nirS群落结构发生改变,其中T5、T6处理下nirS群落结构与常规施肥CF较为接近。
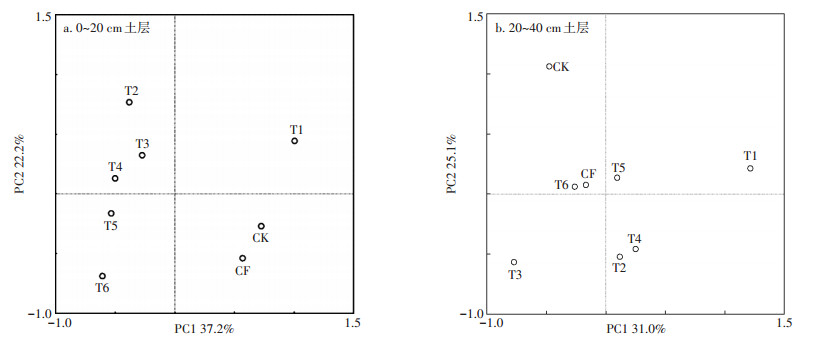
|
图 2 不同施肥处理下两土层中nirS 型反硝化菌群落的PCA分析 Figure 2 The PCA analysis of nirS denitrifying bacteria under different fertilizer treatments in soils |
基于T-RFLP图谱中处理的片段长度的数量和比例的均匀性,选取0~20 cm和20~40 cm土层nirS基因建立克隆文库,在两土层nirS基因文库中均选取约100个克隆子测序,通过登陆NCBI网站进行BLAST分析,剔出假阳性克隆,与Genbank数据库中已存在的序列进行同源性比对,最终获得了42个有代表性的操作分类单元(OTUs),进行系统发育分析,结果如图 3所示,测序获得的nirS序列与其相似菌株的相似性在84%~99%之间。
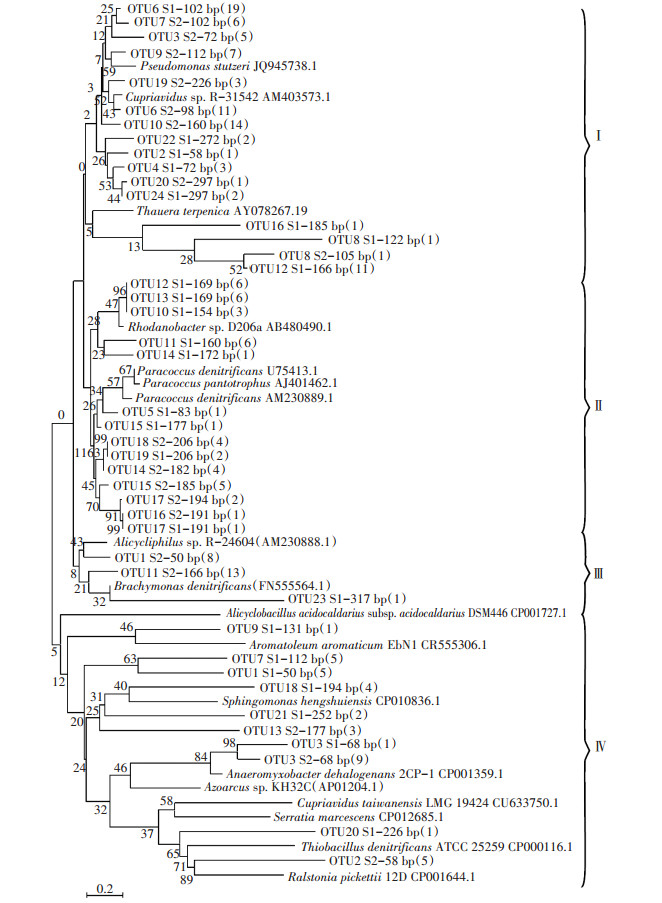
|
S1、S2分别表示0~20 cm和20~40 cm土层;bp表示酶切片段;括号中的数字表示克隆子数 图 3 nirS型反硝化菌系统发育树(邻接法) Figure 3 Neighbour-joining phylogenetic tree of nirS gene sequences |
根据序列在进化树上的分布,将nirS基因系统发育树分为4个类群:其中,总克隆的46.3%分布在第Ⅰ个类群,主要包括0~20 cm土层的优势菌群102 bp(19)、166 bp(11)和20~40 cm土层的优势菌群98 bp(11)、112 bp(7)、160 bp(14),在系统进化树中,这些序列与β-变形菌纲的贪铜菌属(Cupriavidus)、Thauera菌属和γ-变形菌纲的假单胞菌属(Pseudomonas)聚类在一起,有较近的亲缘关系。23.2%的克隆分布在第Ⅱ个类群,主要包括0~20 cm土层的优势菌群160 bp(6)、169 bp(6)和20~40 cm土层的优势菌群169 bp(6),这些序列与α -变形菌纲的副球菌属(Paracoccus)和γ-变形菌纲的Rhodanobacter聚类在一起,有较近的亲缘关系。11.2%的克隆分布在第Ⅲ个类群,此类群中20~40 cm土层的反硝化菌占到11.05%,包括20~40 cm土层的优势菌群50 bp(8)、166 bp(13),而0~20 cm土层仅占0.15%,与此类群相近的微生物为β-变形菌纲的Brachymonas、Alicycliphilus菌属。18.9%的克隆分布在第Ⅳ个类群,主要包括0~20 cm土层的优势菌群112 bp(5)和20~40 cm土层的优势菌群68 bp(9),与此类群相近的微生物为β-变形菌纲的固氮弧菌属(Azoarcus)、贪铜菌属(Cupriavidus)、Ralstonia菌属和γ-变形菌纲的Serratia菌属。
2.3 不同施肥处理对两土层nosZ型反硝化菌群落的影响利用反硝化细菌nosZ基因特异引物对两土层反硝化细菌群落结构进行T-RFLP分析,结果如图 4。在0~20 cm土层中nosZ基因经HhaI酶切得到16个T-RFs片段。不同处理下片段59、177 bp和265 bp均出现且相对丰度较高,是共有的优势菌种,且在各处理下相对丰度无显著变化。另外,片段54 bp在T2、T3、T4、T5和T6处理下相对丰度明显上升,为优势菌种;片段91 bp在T5处理下相对丰度显著增加,也成为该处理下的优势菌种;片段111 bp在T1、T3和T4处理下相对丰度显著增加,成为该处理下的优势菌种;片段117 bp在除T2和T6处理外的其他处理中相对丰度均较高,为其他处理的优势菌种,不同施肥处理下0~20 cm土层nosZ型反硝化菌群落结构发生了变化。
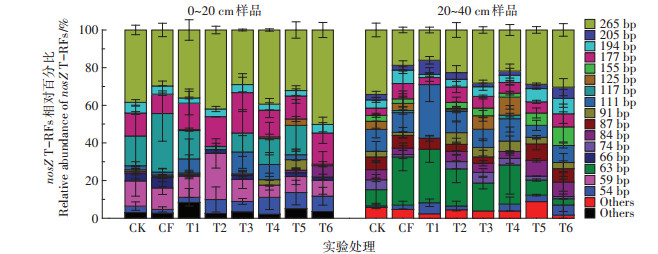
|
图 4 不同处理条件下nosZ型反硝化菌的T-RFLP图谱 Figure 4 T-RFLP fingerprints of nosZ genes from soils under different regimes |
20~40 cm土层中nosZ基因经HhaI酶切产生了20个T-RFs片段,种类较0~20 cm土层丰富。片段111、265 bp在所有处理中均存在且相对丰度较高,是共有优势菌种。片段63 bp在CF、T1、T2、T3和T4处理下相对丰度明显增加,成为这些处理下的优势菌种;片段111 bp在T1处理下相对丰度显著增加;片段125 bp在T3和T4处理下相对丰度显著增加,成为优势菌种;片段84、155 bp和194 bp在T5处理下相对丰度明显增加,成为该处理下的优势菌种。片段63 bp在T6处理下相对丰度明显减少,而片段155 bp和194 bp相对丰度相对增加,成为优势菌种。综上可知,不同施肥处理下20~40 cm土层nosZ型反硝化菌群落结构发生了变化。另外,比较两土层发现20~40 cm土层中出现0~20 cm土层中没有的片段63、111、84、87、125 bp和205 bp,且相对丰度较高,表明相同施肥条件下土壤0~20 cm与20~40 cm土层nosZ型反硝化细菌群落结构存在一定差异。
对不同处理下两土层中nosZ型反硝化菌群落多样性指数分析(表 6)发现,0~20 cm土层中T5处理下的Shannon-Wiener H′指数显著高于T2和T6处理,Simpson D指数T5也高于T6,Pielou E指数均无显著的差别;而20~40 cm土层T5处理下的Shannon-Wiener H′指数也高于T2和T6处理,但差异不显著。比较T5(含氮量为317 kg·hm-2)、T2(含氮量为210 kg· hm-2)和T6(含氮量为126 kg·hm-2)处理,3个处理均为越冬期和拔节期灌溉,表明相同灌溉次数,高浓度肥水灌溉有利于两土层中nosZ型反硝化菌群落多样性发展。另外发现在20~40 cm土层中,T1处理Shannon-Wiener H′指数显著低于其他处理,而T5处理Shannon-Wiener H′指数显著高于CK、CF、T1和T4处理,表明T1处理能显著降低nosZ型反硝化菌群落多样性,T5处理相对其他处理更有助于增加nosZ群落多样性。
|
|
表 6 不同施肥水平处理下两土层nosZ基因多样性指数 Table 6 The diversity index of nosZ denitrifying bacteria in soils under different fertilizer treatments |
根据nosZ基因T-RFs数据进行主成分分析(图 5)发现,在0~20 cm土层中,第一主成分(PC1)解释了59.5%的群落结构变化,第二主成分(PC2)解释了16.1%的群落结构变化。CF、T2和T6处理在PC1上距离CK较远,而T1、T3、T4和T5处理则在PC1上较为接近CK,说明T2、T3、T4和T5处理使0~20 cm土层中nosZ群落结构变化较小,而T2和T6处理nosZ群落结构变化较大,nosZ群落结构展示出对肥水浓度及灌溉次数的敏感性。在20~40 cm土层中,第一主成分(PC1)解释了68.1%的物种组成变化,第二主成分(PC2)解释了16.2%的物种组成变化。T5在PC1和PC2上最接近CK,说明T5处理对20~40 cm土层nosZ群落结构影响较小;T2、T3、T4与CF聚成一簇,说明这几个处理nosZ群落结构相似。

|
图 5 不同施肥处理下两土层中 nosZ型反硝化菌群落的 PCA分析 Figure 5 The PCA analysis of nosZ denitrifying bacteria under different fertilizer treatments in soils |
基于T-RFLP图谱中处理的片段长度的数量和比例的均匀性,选取0~20 cm和20~40 cm土层nosZ基因建立克隆文库,在两土层nosZ基因文库中均选取约100个克隆子测序,通过登陆NCBI网站进行BLAST分析,剔出假阳性克隆,与Genbank数据库中已存在的序列进行同源性比对,最终获得了35个有代表性的操作分类单元(OTUs),进行系统发育分析,结果如图 6所示,测序获得的nosZ序列与其相似菌株的相似性在85%~99%之间。
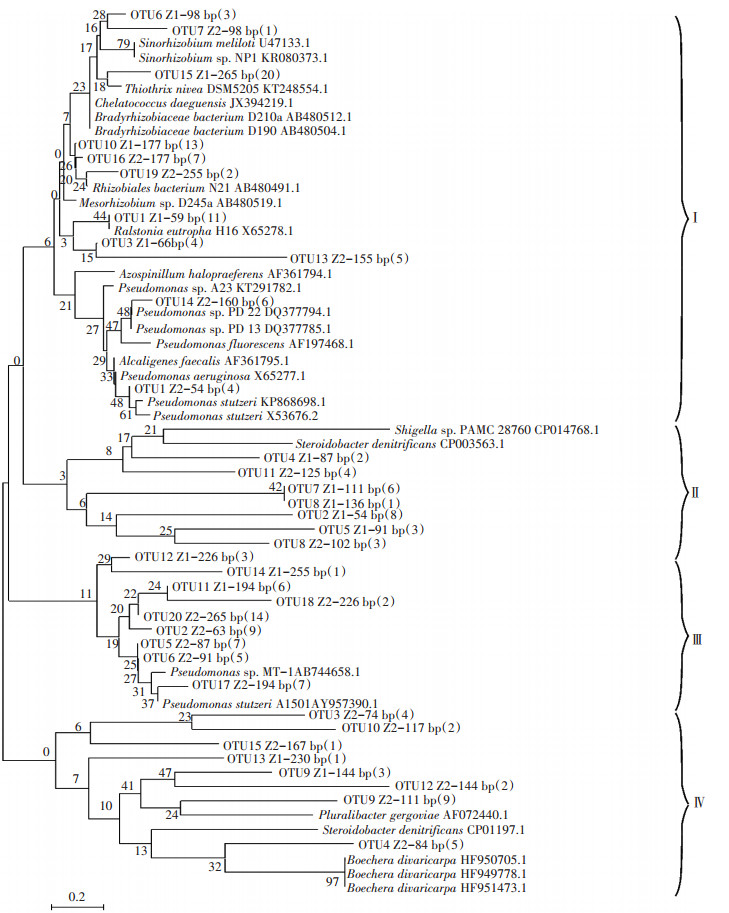
|
Z1、Z2分别表示0~20 cm和20~40 cm土层;bp表示酶切片段;括号中的数表示克隆子数 图 6 nosZ型反硝化菌系统发育树(邻接法) Figure 6 Neighbour-joining phylogenetic tree of nosZ gene sequences |
根据序列在进化树上的分布,将nosZ基因系统发育树分为4个类群:其中,总克隆的39.7%分布在第Ⅰ个类群,主要包括0~20 cm土层的优势菌群54 bp(8)、59 bp(11)、177 bp(13)、265 bp(20)和20~40 cm土层的优势菌群177 bp(7),另外包括两个土层的次优势种群(如:0~20 cm土层的66、98 bp;20~40 cm土层的54、98、155、160 bp和255 bp等),在系统进化树中,这些序列与α-变形菌纲的根瘤菌属(Rhizobium)和固氮螺菌属(Azospirillum)、β-变形菌纲的贪铜菌属(Cupriavidus)和γ -变形菌纲的假单胞菌属(Pseudomonas)、丝硫菌属(Thiothrix)聚类在一起,有较近的亲缘关系。15.1%的克隆分布在第Ⅱ个类群,主要包括0~20 cm土层的54 bp(8)、87 bp(2)、91 bp(3)、111 bp(6)、136 bp(1)和20~40 cm土层的102 bp(3)、125 bp(4),这些序列与α-变形菌纲的中华根瘤菌属(Sinorhizobium)和γ -变形菌纲的志贺氏菌属(Shigella)聚类在一起,有较近的亲缘关系。30.2%的克隆分布在第Ⅲ个类群,主要包括20~40 cm土层的优势菌群63 bp(9)、265 bp(14),此外还包括有Z1- 226 bp(3)、Z1-255 bp(1)、Z1-194 bp(6)片段,Z2- 226 bp(2)、Z2-265 bp(14)、Z2-63 bp(9)、Z2-87 bp(7)、Z2-91 bp(5)、Z2-194 bp(7)、Z2-74 bp(4)等片段,与此类群相近的微生物为γ-变形菌纲的假单胞菌属(Pseudomonas)。15.1%的克隆分布在第Ⅳ个类群,主要包括20~40 cm土层的优势菌群111 bp(9),此外还包括有Z1-230 bp(1)、Z1-144 bp(3)、Z2-144 bp(2)、Z2-74 bp(4)、Z2-117 bp(2)、Z2-167 bp(1)、Z2-84 bp(5)等片段,与此类群相近的微生物为γ-变形菌纲的Steroidobacter菌属和Pluralibacter菌属。
3 讨论反硝化作用是在微生物参与下的土壤氮循环中的一个重要过程,反硝化作用强弱直接影响着氮素的利用[14]。杜会英等[15]研究发现,牛场肥水灌溉使冬小麦-夏玉米轮作体系作物累计氮利用率逐年升高,且显著高于常规化肥处理。然而农田施用氮肥会导致温室气体N2O的排放,通过管理氮肥的种类及用量则可在一定程度上控制N2O的排放[16]。沈仕洲等[17]研究发现,采用不同浓度奶牛场沼液灌溉都使N2O排放较清水增加,但不高于常规化肥排放值。可见,牛场肥水灌溉可能通过调节土壤反硝化过程提高了氮素利用率。
温度、pH、水分、含氧量、碳氮类型和碳氮比以及土壤质地、土壤利用和耕作方式等都对反硝化作用产生影响[14, 18]。本研究各试验田的光照、土壤、作物、种植方式、灌溉次数与灌溉水量均相同,不同施肥方式主要通过pH、碳氮类型和碳氮量对反硝化微生物产生影响。因采用了长期定位试验田,施肥模式从2012年10月持续到2015年6月,土壤性质因长期的施肥措施产生了一定差异(表 1),施肥处理组(CF,T1~T6)的NO3--N和NH4+-N普遍高于不施肥处理CK,这种差异反映了施肥措施的长期影响;同样,反硝化微生物群落结构的改变也反映了在施肥措施影响下的长期适应结果。本研究中nirS型反硝化细菌主要与假单胞菌属(Pseudomonas)、副球菌属(Paracoccus)、贪铜菌属(Cupriavidus)具有较近的亲缘关系;而nosZ型反硝化细菌与假单胞菌属(Pseudomonas)、根瘤菌属(Rhizobium)、固氮螺菌属(Azospirillum)、贪铜菌属(Cupriavidus)等具有较近的亲缘关系,这些菌属与已知报道吻合[14, 18]。
现有报道nirS型反硝化细菌群落结构对施肥条件的响应存在矛盾,如宋亚娜等[19]研究发现施用氮肥对稻田土壤的nirS型反硝化细菌群落结构没有明显影响;Yin等[20]发现,我国南方水稻土中nirS型反硝化菌的群落结构对施加无机肥和有机肥处理均没有显著的响应;另一些研究则表明nirS对施肥有明显响应,如莫旭华等[21]发现,不同类型的反硝化细菌对无机氮肥的反应不同而导致了nirS型反硝化细菌群落结构改变,但未改变nirS型反硝化细菌的多样性;王婷等[22]研究发现牛场肥水施用改变了0~5 cm表层土中nirS型反硝化细菌的多样性。本研究发现施肥种类及施肥方式对0~20 cm土层nirS型反硝化细菌群落组成无显著影响,但对20~40 cm土层nirS型反硝化细菌有一定影响,这与前述两种报道都有吻合之处。这可能是由于nirS型反硝化细菌群落结构更易受土壤异质性(土层深度)的影响。本研究还发现施肥措施的不同对两土层nosZ型反硝化细菌群落结构有明显的影响,nosZ型反硝化细菌群落结构对施肥种类及施肥方式更为敏感。有研究显示nirS基因的分布与样品所处的环境类型有关,nirS基因序列在不同的环境类型中存在的差异非常大,即使在土壤环境相同的情况下,nirS基因的序列也存在很大的不同[23];另外,Ishii等[24]研究亦发现对土壤环境变化响应的反硝化菌种群具有土壤的特异性,这在一定程度上解释了两土层中nirS型反硝化菌对不同施肥处理的不同响应。两土层nirS型反硝化细菌群落结构对施肥条件的响应是综合了0~20 cm和20~40 cm土层不同环境因素影响的结果,如水分、NO3--N含量和氧分压等因素,本实验中施肥条件下两土层土壤水分含量相近,可见水分不是导致两土层中施肥条件下nirS型反硝化细菌群落结构特征变化不同的原因。土壤中反硝化细菌的分布与土壤环境中NO3--N浓度及氧分压密切相关[25-26],NO3--N是反硝化作用的底物,本实验中施肥条件下两土层的NO3--N含量变化差异明显,另外20~40 cm土层较之0~20 cm氧分压明显较低,施肥处理下土壤低氧含量的20~40 cm土层环境可能更适合nirS型反硝化细菌的生存,使得适合在该环境条件下生存的反硝化细菌的种类及丰度增加,逐渐取代了数量较少且不适宜在该环境下生存的反硝化细菌,从而成为优势种群并发挥重要作用[27],所以NO3--N含量和氧分压等综合因素的影响可能是导致两土层中施肥条件下nirS、nosZ型反硝化细菌群落结构特征变化不同的原因。
对nirS、nosZ基因的多样性指数的研究显示实验条件下施肥种类及方式对nirS、nosZ基因多样性指数影响不同。不同施肥处理条件下两土层nosZ型反硝化细菌群落组成受肥水灌溉及灌溉次数等的环境变化影响显著,说明土壤nosZ型反硝化微生物群落多样性指数受到施肥的影响,这与之前研究相一致,如Enwall等[28-29]研究发现nosZ型反硝化细菌的群落对有机肥敏感,氮肥可导致nosZ型反硝化细菌多样性降低,Chen等[30]研究发现长期单施氮肥可提高nosZ基因型反硝化细菌群落多样性,Patrick等[31]研究发现nosZ型反硝化细菌的多样性和丰度随着氮输入的增加而增加。本研究还发现,尽管不同施肥处理改变了20~40 cm土层nirS型反硝化细菌群落结构,但并未对多样性造成显著改变,说明这种改变主要是优势菌群与劣势菌群位置的互换,并未显著增加菌群的种类,因此整体多样性改变不大。另外不同施肥处理条件下土壤nosZ型反硝化微生物群落多样性指数影响也存在差异,相同施氮量(约300 kg·hm-2)的CF、T5和T3处理下相比常规施肥处理较高浓度肥水灌溉(T5)能更好地促进两土nosZ基因多样性发展。本研究还发现0~20 cm土层中的nirS、nosZ基因的3个多样性指数低于20~40 cm土层,原因可能在于施肥条件下0~20 cm土层中nirS、nosZ型反硝化细菌片段的种类不及20~40 cm土层丰富,反硝化菌可以在厌氧环境下更好地生存的结果。
4 结论nirS型反硝化细菌群落结构在0~20、20~40 cm土层中存在一定的垂直分布差异,且0~20 cm土层中nirS群落结构对施肥种类、肥水浓度及灌溉次数不敏感,而20~40 cm土层中nirS群落结构对施肥条件敏感性提高。两土层nosZ型反硝化细菌群落均对施肥条件较为敏感。
相同灌溉次数,较高肥水浓度灌溉(T5处理:含氮为317 kg·hm-2,清水:沼液=1:1)有利于两土层中nosZ型反硝化菌群落多样性发展,而较低浓度肥水灌溉1次(T1处理:含氮为105 kg·hm-2)显著降低20~40 cm土层中nosZ型反硝化菌群落多样性。不同施肥条件不改变两土层nirS群落多样性。
本实验所得到的nirS、nosZ序列均来自细菌中的变形菌纲,主要与α、β、γ-变形菌纲相近,且分布广泛。
| [1] |
王风, 高尚宾, 张克强, 等. 废水灌溉农田研究进展与展望[J]. 土壤通报, 2009, 40(6): 1485-1488. WANG Feng, GAO Shang-bin, ZHANG Ke-qiang, et al. Development and expectation of research on wastewater irrigation in farmland[J]. Chinese Journal of Soil Science, 2009, 40(6): 1485-1488. |
| [2] |
陶晓婷, 朱正杰, 高威, 等. 规模化猪场处理废水与化肥配施对小麦氮素吸收利用的影响[J]. 农业环境科学学报, 2014, 33(3): 555-561. TAO Xiao-ting, ZHU Zheng-jie, GAO Wei, et al. Nitrogen uptake and utilization in wheat as influenced by pig slurry from large-scale pig farm[J]. Journal of Agro-Environment Science, 2014, 33(3): 555-561. |
| [3] |
Mohammad M J, Mazahreh N. Changes in soil fertility parameters in response to irrigation of forage crops with secondary treated wastewater[J]. Communications in Soil Science and Plant Analysis, 2003, 34(9/10): 1281-1294. |
| [4] |
王玮, 孙岩斌, 周祺, 等. 国内畜禽厌氧消化沼液还田研究进展[J]. 中国沼气, 2015, 33(2): 51-57. WANG Wei, SUN Yan-bin, ZHOU Qi, et al. A review on irrigation of biogas slurry from livestock manure anaerobic fermentation in China[J]. China Biogas, 2015, 33(2): 51-57. DOI:10.3969/j.issn.1000-1166.2015.02.009 |
| [5] |
曹汝坤, 陈灏, 赵玉柱. 沼液资源化利用现状与新技术展望[J]. 中国沼气, 2015, 33(2): 42-50. CAO Ru-kun, CHEN Hao, ZHAO Yu-zhu. Resource utilization of biogas slurry:Current status and future prospects[J]. China Biogas, 2015, 33(2): 42-50. DOI:10.3969/j.issn.1000-1166.2015.02.008 |
| [6] |
方晶晶, 马传明, 刘存富. 反硝化细菌研究进展[J]. 环境科学与技术, 2010(增刊1): 206-210. FANG Jing-jing, MA Chuan-ming, LIU Cun-fu. The advance of study on denitrifying bacteria[J]. Environmental Science & Technology, 2010(Suppl 1): 206-210. |
| [7] |
Mosier A R, Duxbury J M, Freney J R, et al. Assessing and mitigating N2O emissions from agricultural soils[J]. Climatic Change, 1998, 40(Suppl 1): 7-38. |
| [8] |
Firestone. Biological denitrification[C]//Stevenson E J. Agronomy Monoyraph 22, American Society of Agronomy, 1982: 289-326.
|
| [9] |
Rösch C, Mergel A, Bothe H. Biodiversity of denitrifying and dinitrogen-fixing bacteria in an acid forest soil[J]. Applied & Environmental Microbiology, 2002, 68(8): 3818-3829. |
| [10] |
Li F, Li M, Shi W, et al. Distinct distribution patterns of proteobacterial nirK-and nirS-type denitrifiers in the Yellow River estuary, China[J]. Canadian Journal of Microbiology, 2017, 63(8): 708-718. DOI:10.1139/cjm-2017-0053 |
| [11] |
Braker G, Zhou J Z, Wu L Y, et al. Nitrite reductase genes(nirK and nirS)as functional markers to investigate diversity of denitrifying bacteria in Pacific northwest marine sediment communities[J]. Applied and Environmental Microbiology, 2000, 66(5): 2096-2104. DOI:10.1128/AEM.66.5.2096-2104.2000 |
| [12] |
郭丽芸, 时飞, 杨柳燕, 等. 反硝化菌功能基因及其分子生态学研究进展[J]. 微生物学通报, 2011, 38(4): 583-590. GUO Li-yun, SHI Fei, YANG Liu-yan, et al. Advances in functional genes and molecular ecology in denitrifiers[J]. Microbiology China, 2011, 38(4): 583-590. |
| [13] |
Barrett M, Jahangir M M, Lee C, et al. Abundance of denitrification genes under different peizometer depths in four Irish agricultural groundwater sites[J]. Environmental Science and Pollution Research, 2013, 20(9): 6646-6657. DOI:10.1007/s11356-013-1729-3 |
| [14] |
王海涛, 郑天凌, 杨小茹. 土壤反硝化的分子生态学研究进展及其影响因素[J]. 农业环境科学学报, 2013, 32(10): 1915-1924. WANG Hai-tao, ZHENG Tian-ling, YANG Xiao-ru. Molecular ecology research progress for soil denitrification and research status for its influencing factors[J]. Journal of Agro-Environment Science, 2013, 32(10): 1915-1924. DOI:10.11654/jaes.2013.10.003 |
| [15] |
杜会英, 冯洁, 郭海刚, 等. 麦季牛场肥水灌溉对冬小麦-夏玉米轮作土壤氮素平衡的影响[J]. 农业工程学报, 2015, 31(3): 159-165. DU Hui-ying, FENG Jie, GUO Hai-gang, et al. Effects of dairy effluents irrigation on N balance in soil under winter wheat-summer maize rotation system[J]. Transactions of the CSAE, 2015, 31(3): 159-165. DOI:10.3969/j.issn.1002-6819.2015.03.022 |
| [16] |
Lazcano C, Tsang A, Doane T A, et al. Soil nitrous oxide emissions in forage systems fertilized with liquid dairy manure and inorganic fertilizers[J]. Agriculture Ecosystems & Environment, 2016, 225: 160-172. |
| [17] |
沈仕洲, 王风, 薛长亮, 等. 沼液灌溉对冬麦田CO2、N2O排放及土壤因子的影响[J]. 节水灌溉, 2015(7): 25-31. SHEN Shi-zhou, WANG Feng, XUE Chang-liang, et al. Effects of irrigating biogas slurry on CO2 and N2O emission and soil parameters in winter-wheat field[J]. Water Saving Irrigation, 2015(7): 25-31. DOI:10.3969/j.issn.1007-4929.2015.07.007 |
| [18] |
刘秋丽, 马娟娟, 孙西欢, 等. 土壤的硝化-反硝化作用因素研究进展[J]. 农业工程, 2011, 1(4): 79-83. LIU Qiu-li, MA Juan-juan, SUN Xi-huan, et al. Research advancement on soil nitrification-denitrification and its influencing fators[J]. Agricultural Engineering, 2011, 1(4): 79-83. |
| [19] |
宋亚娜, 林智敏, 林艳. 氮肥对稻田土壤反硝化细菌群落结构和丰度的影响[J]. 中国生态农业学报, 2012, 20(1): 7-12. SONG Ya-na, LIN Zhi-min, LIN Yan. Response of denitrifying bacteria community structure and abundance to nitrogen in paddy fields[J]. Chinese Journal of Eco-Agriculture, 2012, 20(1): 7-12. |
| [20] |
Yin C, Fan F, Song A, et al. Different denitrification potential of aquic brown soil in Northeast China under inorganic and organic fertilization accompanied by distinct changes of nirS-and nirK -denitrifying bacterial community[J]. European Journal of Soil Biology, 2014, 65: 47-56. DOI:10.1016/j.ejsobi.2014.09.003 |
| [21] |
莫旭华, 麻威, 史荣久, 等. 氮肥对小麦田土壤nirS型反硝化细菌多样性的影响[J]. 微生物学报, 2009, 49(9): 1203-1208. MO Xu-hua, MA Wei, SHI Rong-jiu, et al. Diversity of nirS-type denitrifying bacteria under different nitrogen fertilizer management in wheat soil[J]. Acta Microbiologica Sinica, 2009, 49(9): 1203-1208. DOI:10.3321/j.issn:0001-6209.2009.09.012 |
| [22] |
王婷, 刘丽丽, 张克强, 等. 牛场肥水灌溉对土壤nirK、nirS型反硝化微生物群落结构的影响[J]. 生态学报, 2017, 37(11): 3655-3664. WANG Ting, LIU Li-li, ZHANG Ke-qiang, et al. Effects of fertilizer on community structure of nirK and nirS type denitrifying bacteria in soil[J]. Acta Ecologica Sinica, 2017, 37(11): 3655-3664. |
| [23] |
Priemé A, Braker G, Tiedje M James. Diversity of nitrite reductase (nirK and nirS)gene fragments in forested upland and wetland soils[J]. Applied and Environmental Microbiology, 2002, 68(4): 1893-1900. DOI:10.1128/AEM.68.4.1893-1900.2002 |
| [24] |
Ishii S, Yamamoto M, Tago K, et al. Microbial populations invarious paddy soils respond differently to denitrification-inducing conditions, albeit background bacterial populations are similar[J]. Soil Science & Plant Nutrition, 2010, 56(2): 220-224. |
| [25] |
Cavigelli M A, Robertson G P. The functional significance of denitrifier community composition in a terrestrial ecosystem[J]. Ecology, 2000, 81(5): 1402-1414. DOI:10.1890/0012-9658(2000)081[1402:TFSODC]2.0.CO;2 |
| [26] |
Alyson E S, Alexandria B B, Christopher A F. Denitrifier community composition along a nitrate and salinity gradient in a coastal aquifer[J]. Applied and Environmental Microbiology, 2006, 72(3): 2102-2109. DOI:10.1128/AEM.72.3.2102-2109.2006 |
| [27] |
曾希柏, 王亚男, 王玉忠, 等. 施肥对设施菜地nirK型反硝化细菌群落结构和丰度的影响[J]. 应用生态学报, 2014, 25(2): 505-514. ZENG Xi -bai, WANG Ya -nan, WANG Yu-zhong, et al. Effects of different fertilization regimes on abundance and community structure of the nirK type denitrifying bacteria in greenhouse vegetable soils[J]. Chinese Journal of Applied Ecology, 2014, 25(2): 505-514. |
| [28] |
Enwall K, Philippot L, Hallin S. Activity and composition of the denitrifying bacterial community respond differently to long-term fertilization[J]. Applied and Environmental Microbiology, 2009, 71(11): 8335-8343. |
| [29] |
Enwall K, Hallin S. Comparison of T-RFLP and DGGE techniques to assess denitrifier community composition in soil[J]. Letters in Applied Microbiology, 2009, 48(1): 145-148. |
| [30] |
Chen Z, Yuan H Z, Wu J S, et al. Activity and composition of the denitrifying bacterial community respond differently to long-term fertilization[J]. Acta Ecologica Sinica, 2009, 29(11): 5923-5929. |
| [31] |
Patrick J K, John H A, et al. Long-term nutrient addition differentially alters community composition and diversity of genes that control nitrous oxide flux from salt marsh sediments[J]. Estuarine, Coastal and Shelf Science, 2015, 154: 39-47. DOI:10.1016/j.ecss.2014.12.014 |
 2019, Vol. 38
2019, Vol. 38