2. 华南农业大学国家生猪种业工程技术研究中心, 广州 510642;
3. 华南农业大学农业部华南热带农业环境重点实验室, 广州 510642
2. National Engineering Research Center for Breeding Swine Industry, South China Agricultural University, Guangzhou 510642, China;
3. Key Laboratory of Tropical Agriculture Environment of South China, Ministry of Agriculture, Guangzhou 510642, China
猪场废水处理系统是抗生素抗性基因的重要储存库,其最高绝对丰度达到108 copies·mL-1,相对丰度达到10-1 copies·16S rRNA-1 [1-2]。猪场废水中的抗性基因由不同耐药类型的抗性基因亚类组成。Jia等[3]在猪场废水中检测到196种抗性基因,包括常见四环素类、氨基糖苷类、磺胺类、多重耐药类、大环内酯类、β-内酰胺类、喹诺酮类、甲氧苄啶、链霉素、多黏菌素和万古霉素等的14大类抗性基因。废水中不同类型的抗性基因存在于菌群中,赋予菌群抵御不同类型抗生素的能力,甚至存在于作物病原菌和人类致病菌中,增加耐药性的污染风险。然而,研究表明猪场废水处理系统并不能完全去除废水中的抗性基因,其系统出水中仍能检测到102~106 copies· mL-1的抗性基因[2, 4]。系统出水中残留的抗性基因及耐药菌会随着出水被重复利用或外排进入到环境中,对猪场及周边环境造成更广泛的污染,威胁养殖生产及公共健康。
废水中的总抗性基因包括细菌胞内抗性基因和胞外抗性基因[5-6]。目前大部分的研究侧重细菌胞内抗性基因,缺少对胞外抗性基因的研究。胞外抗性基因包括废水中游离的抗性基因及噬菌体所携带的抗性基因。水体中游离的抗性基因是指游离的核酸片段,存在形态不稳定,容易受pH、温度和重金属等环境因子的影响[7],并且只有融入到细菌中才能发挥其耐药功能。噬菌体是细菌之间基因水平转移的主要载体,可从感染细菌中摄取抗性基因,是废水中抗性基因的小型储存库[8-9]。在噬菌体感染下一个细菌时,其携带的抗性基因可通过转导的方式传播到其他细菌中,实现抗性基因在不同细菌间的水平转移,增加环境抗性基因污染的复杂性。因此,环境中噬菌体携带抗性基因的污染现状受到广泛关注,在畜禽粪便、农田土壤和城市污水等环境均检测到高丰度的噬菌体携带抗性基因。猪场废水处理系统出水是周边环境重要的潜在污染源之一。然而,目前鲜有关于猪场废水处理系统出水及周边环境中噬菌体携带抗性基因的报道。
基于此,本研究选择5个典型猪场废水处理系统为研究对象,采集其出水样及排入河流上下游水样,检测9种抗性基因及2种整合子基因的丰度,分析其在不同猪场出水及周边河流中的污染特征,初步探讨猪场废水处理系统出水中噬菌体携带抗性基因对周边河流环境的影响,为评估养猪场抗性基因污染风险提供参考。
1 材料与方法 1.1 采样猪场选择位于江西省境内的5个规模化猪场的废水处理系统,采集其系统出水及排入河流上、下游水样。共采集5个种猪场,编号分别为A、B、C、D、E,其养殖规模分别为5 500、3 500、6 000、9 000、16 000头(存栏母猪),均采用厌养+两级A/O(缺氧、好氧工艺)+加药混凝、消毒废水处理系统。
1.2 主要仪器及试剂主要仪器:普通PCR仪(BIO-RAD,T100,美国)、凝胶成像系统(Tanon,2500,中国)、实时荧光定量PCR仪(BIO-RAD,CFX96,美国)、循环水真空泵[瑞德,SHZ-D(Ⅲ),中国]、台式高速冷冻离心机(Micro 17R,Thermo Scientific,美国)、微量紫外分光光度计(Nano Drop,2000,Thermo Scientific,美国)和-80 ℃超低温冰箱(Haier,DW-86L626,中国)。
主要试剂:SYBR Green荧光定量检测试剂盒(TIANGEN,FP209,中国)、pMD18-T Vector(Takara,日本)、DH5α感受态细胞(TIANGEN,CB101,中国)、噬菌体DNA提取试剂盒(TIANGEN,TIANamp Virus DNA / RNA Kit,中国)、质粒提取试剂盒(OMEGA,Plasmid Mini Kit,美国)、超强高保真PCR试剂盒(TIANGEN,KP203,中国)。
1.3 试验方法 1.3.1 水样采集及预处理采集5个典型猪场废水处理系统的出水样、排入河流的上游200 m处水样和下游200 m处水样,每一个采样点同时采集3个样品作为3个重复;每个重复样采集相隔1 m,采集水面10 cm处水样,每个样4 L。所有样品用冰覆盖运输,当日到达实验室进行预处理。用0.22 μm孔径低蛋白结合滤膜去除水样中的细菌,获得含有噬菌体的滤液,并用100 kDaAmicon Ultra离心过滤器(Millipore)滤去游离的核酸,并将滤液浓缩至1 mL(浓缩500倍)。含噬菌体的浓缩样于- 80 ℃保存备用。
1.3.2 检测指标及方法提取样品噬菌体中的DNA,检测3种四环素类抗性基因(tetG、tetX和tetW)、2种磺胺类抗性基因(sul1和sul2)、2种大环内酯类抗性基因(ermA和ermB)、1种β-内酰胺类抗性基因(blaTEM)、1种氯霉素类抗性基因(cmlA)和2个整合子基因(intl1和intl2)的绝对丰度。
(1)噬菌体DNA提取:按照噬菌体DNA提取试剂盒(TIANGEN,TIANamp Virus DNA/RNA Kit,中国)提供的说明书提取样品中的噬菌体DNA,用超微量分光光度计测定所提取DNA样品的浓度及D260/280,经1%凝胶电泳分析所提DNA样品的质量,并用16S rRNA通用引物对所提DNA样品进行定性,确保所提噬菌体DNA样品不含细菌DNA。
(2)建立荧光定量PCR标准曲线:参考本团队之前研究构建的qPCR标准曲线[10]。主要步骤如下:利用抗性基因特异性引物(表 1)对样品进行普通PCR,通过凝胶电泳获得特异性条带。将确认后的特异性条带进行回收纯化,构建于pMD180T载体中,并转化到DH5α感受态细胞。菌液扩繁,提取菌液质粒进行10倍稀释,获得以质粒为模板的荧光定量标准曲线。标准曲线的拷贝数(copies)=L×C/(N×M×109),其中L为阿弗加德罗常数(6.02×1023),C是质粒DNA浓度,N为目的DNA模板长度(目的基因长度+2 992 bp),M为每对DNA的平均分子量(660)。
|
|
表 1 抗性基因及整合子PCR引物信息 Table 1 PCR primer information of resistance genes and integrants |
每个稀释梯度设3个重复进行扩增反应。反应体系为25 μL:SYBR Mix 12.5 μL,上下游引物(10 μmol·L-1)各0.75 μL,ddH2O 10 mL,模板1 μL。反应程序为:95 ℃预变性3 min,95 ℃变性5 s,60 ℃退火15 s,40个循环,溶解曲线按默认程序。所有基因的标准曲线扩增效率在95%~105%,R2大于0.99。
(3)样品中抗性基因及可移动遗传元件的检测:采用建立好的标准曲线,以样品噬菌体DNA为模板,使用(2)的反应体系和反应程序,每个样品3个重复,检测样品噬菌体中抗性基因及可移动遗传元件的拷贝数。抗性基因和可移动遗传元件拷贝数(copies· mL-1)=标曲算出的拷贝数(copies·μL-1)×DNA洗脱体积(μL)/样品体积(mL),基因绝对丰度使用基因拷贝数(N)的以10为底的对数(lgN)表示。
1.4 数据分析检测数据经Excel进行初步处理,GraphPad Prism 8.0软件作图,SPSS 22.0软件进行单因素方差分析和相关性分析(Spearman),结果以“平均数±标准误”表示,P < 0.05表示差异显著。
2 结果与分析 2.1 噬菌体携带抗性基因的检出情况对5个典型猪场废水处理系统出水及周边河流上下游水样中的9种噬菌体携带抗性基因和2种整合子基因进行普通PCR定性检测,结果如表 2所示。所有样品均检测到抗性基因blaTEM、sul2、tetW和tetX;在猪场废水处理系统出水中只有1/5(C场)的采样点检测到抗性基因cmlA,2/5(B场和C场)的采样点检测到抗性基因sul1,而所有上下游样品均检测到cmlA和sul1;在系统出水中只有1/5(E场)的采样点检测到抗性基因ermA,而有4/5的上游河流采样点和3/5的下游河流采样点检测到ermA,表明猪场废水处理系统出水中噬菌体携带抗性基因检测率低于周边的河流样,这也说明本研究中的猪场废水处理系统出水不是周边河流噬菌体携带抗性基因的主要污染来源。所有上游河流样都检测到抗性基因ermB和tetG,而在下游河流中只有4/5(除了B场)的采样点检测到ermB和tetG;上游河流样中有4/5的采样点检测到抗性基因ermA,而下游只有3/5的采样点检测到ermA,表明下游河流样中的噬菌体携带抗性基因检出率低于上游河流样,也说明河流中的抗性基因等污染物可能通过河流自净功能被消除[19]。此外,所有样品噬菌体中都检测到与基因水平转移相关的整合子基因intl1和intl2,这增加了系统出水和周边河流中噬菌体携带抗性基因污染的复杂性。
|
|
表 2 不同采样点中抗性基因及整合子基因的检出情况 Table 2 Detection of resistance genes and integron genes in different sampling sites |
对所有样品中的噬菌体携带抗性基因进行检测,结果发现5个猪场废水处理系统出水和周边河流中含有较高丰度的噬菌体携带抗性基因(表 3),其中磺胺类抗性基因sul2的平均丰度最高,高达4.09±0.16,显著高于四环素类抗性基因tetX(3.24±0.19)和tetG(3.22±0.29)(P < 0.05);tetX和tetG的绝对丰度显著高于磺胺类抗性基因sul1(2.46±0.39)和四环素类抗性基因tetW(2.38±0.18)(P < 0.05);sul1和tetW的绝对丰度与blaTEM(2.18±0.22)的丰度之间没有显著差异,显著高于大环内酯类抗性基因ermB(2.04±0.18)和氯霉素类抗性基因cmlA(1.93±0.34)(P < 0.05);大环内酯类抗性基因ermA的平均绝对丰度最低,为1.05±0.28,显著低于其他抗性基因(P < 0.05)。整合子基因intl1和intl2也被检测含有较高的丰度,其平均丰度对数值分别为3.70±0.15和2.28±0.18。
|
|
表 3 样品中抗性基因及整合基因绝对丰度 Table 3 The logarithm of the copy number of resistance genes and integron genes in samples |
对样品中9种噬菌体携带抗性基因和2种整合子基因的丰度进行双变量相关性分析,采用Spearman相关系数,双尾检验,结果如图 1所示。结果发现,除基因tetW和blaTEM、intl2和ermB,以及intl2和tetW之间外,其余抗性基因之间均呈显著正相关(P < 0.05),其中氯霉素抗性基因cmlA分别与β-内酰胺类抗性基因blaTEM和磺胺类抗性基因sul1之间存在强正相关(P < 0.05);整合子基因intl1分别与抗性基因blaTEM、cmlA和sul1之间呈强正相关(P < 0.05)。以上结果表明猪场废水处理系统出水及周边河流中的噬菌体携带抗性基因及整合子基因之间存在共现模式。

|
|r|≥0.80表示强相关,0.50≤|r| < 0.8表示中等相关,0<|r| < 0.50表示弱相关,r=0表示不存在相关性,“*”表示相关性显著(P < 0.05) |r|≥0.80 means strong correlation, 0.50≤|r| < 0.8 means medium correlation, 0 < |r| < 0.50 means weak correlation, r=0 means there is no correlation, "*"means significant correlation(P < 0.05) 图 1 不同基因丰度之间相关性分析热图 Figure 1 Heatmap of correlation analysis between different gene abundances |
对不同猪场废水处理系统出水样和上下河流水样中的噬菌体携带抗性基因丰度进行分析(图 2),结果发现不同猪场样品中噬菌体携带抗性基因的丰度有较大差异。氯霉素抗性基因cmlA和磺胺类抗性基因sul1的丰度在各猪场之间没有显著差异(P > 0.05);而β-内酰胺类抗性基因blaTEM在猪场E的绝对丰度最高,为2.69±0.29,显著高于其他猪场(P < 0.05);磺胺类抗性基因sul2在猪场B(4.58±0.21)和C(4.50± 0.16)中的丰度显著高于其他猪场(P < 0.05);大环内酯类抗性基因ermB在猪场C(2.62±0.16)中的丰度显著高于其他猪场(P < 0.05);大环内酯类抗性基因ermA在猪场E中的丰度最高,为1.43±0.4;四环素类抗性基因tetG在猪场A中的丰度最高,为3.62±0.34;tetW在猪场B(2.88±0.35)中的丰度最高;tetX在猪场E中的丰度最高,为3.68±0.25。
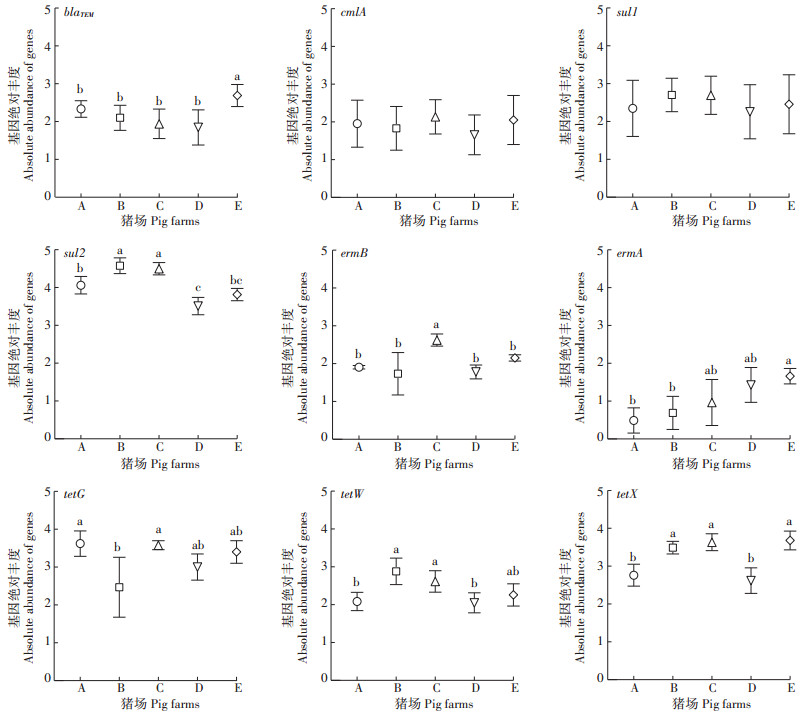
|
A~E表示不同猪场对应的所有样品(系统出水和周边河流样品),不同小写字母代表猪场间差异显著 A~E represent all samples from different farms(effluent and surrounding river samples). Different lowercase letters indicate significant differences among pig farms 图 2 不同猪场样品噬菌体携带抗性基因的绝对丰度 Figure 2 Absolute abundance of phage carrying resistance genes in samples from different farms |
菌体携带抗性基因的绝对丰度对猪场废水处理系统出水、河流上游和下游水样中噬菌体携带抗性基因的丰度进行分析,结果发现系统出水中9种抗性基因的总丰度(2.01±0.21)显著低于河流上游(3.03±0.13)和下游(2.88±0.16)中总抗性基因的丰度(P < 0.05)(图 3),河流上游的总抗性基因丰度高于河流下游,但差异不显著(P > 0.05)。其中,β-内酰胺类抗性基因blaTEM、氯霉素类抗性基因cmlA、磺胺类抗性基因sul1和大环内酯类抗性基因ermA在系统出水中的绝对丰度分别为1.38±0.34、0.10±0.10、0.50±0.31和0.26±0.26,显著低于其在河流上下游中的丰度(P < 0.05)(图 4);抗性基因sul2在河流上游中的丰度高于系统出水和河流下游中的丰度,但差异不显著(P > 0.05);其他噬菌体携带抗性基因在系统出水、河流上游和下游中的丰度没有显著差异。此外,整合子基因intl1和intl2基因在系统出水中的丰度显著低于河流上游和下游中的丰度(P < 0.05)。综上结果表明,猪场废水系统出水中抗性基因的丰度显著低于河流中抗性基因的丰度,这也进一步说明猪场废水处理系统出水中噬菌体携带的抗性基因未对周边河流造成显著影响。
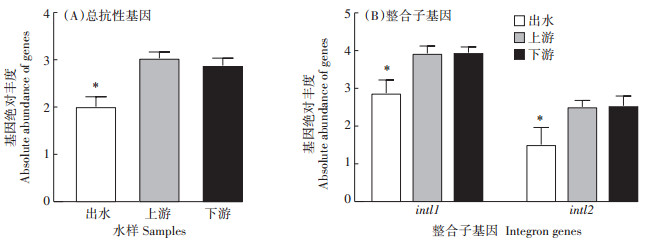
|
“*”表示差异显著(P < 0.05)。下同 "*" indicates significant difference among different samples(P < 0.05). The same below 图 3 系统出水及河流上下游水样中总抗性基因及整合子基因的丰度 Figure 3 Abundance of total resistance genes and integron genes in the effluent of the system and the water samples of the upstream and downstream |

|
图 4 系统出水及河流上下游样品中不同噬菌体携带抗性基因的丰度 Figure 4 Abundance of resistance genes carried by different bacteriophages in samples from the effluent, upstream and downstream |
猪场废水处理系统是连接废水和外界环境的重要桥梁,其出水中残留的抗性基因直接影响周边环境的安全。然而目前大部分的报道仅对系统出水中的细菌抗性基因进行了研究,缺少关于系统出水中噬菌体携带抗性基因的报道。研究表明,猪场废水处理系统出水中仍能检测到高丰度的细菌抗性基因,其检出丰度高达105~106 copies·mL-1 [20-21]。本研究对5个典型猪场废水处理系统出水中的9种噬菌体携带抗性基因进行检测,结果发现系统出水中噬菌体携带抗性基因的平均丰度高达2.01±0.21,其中sul2和tetX平均丰度分别高达3.85±0.38和3.24±0.28,说明猪场废水处理系统不能完全去除废水中的噬菌体携带抗性基因。此外,还检测到高丰度的整合子基因intl1和intl2。整合子基因与细菌基因水平转移相关,可把抗性基因等遗传物质整合到转座子和质粒等可移动遗传因子上,实现抗性基因在不同细菌间的传播[22],进一步促进系统出水中抗性基因的水平转移。系统出水中残留的噬菌体携带抗性基因和整合子基因增加了抗性基因防控的复杂性,也说明在关注细菌抗性基因污染的同时,也应重视环境中噬菌体携带抗性基因污染问题。
猪场废水处理系统出水中残留的抗性基因会随着出水外排进入周边河流中,对周边环境安全造成威胁。本研究在5个猪场周边河流的上下游样品中均检测到9种噬菌体携带抗性基因,其中blaTEM、cmlA、sul1、sul2、tetW和tetX的检出率是100%。在河流上游中噬菌体携带抗性基因的平均检出丰度高于下游的检测丰度,但差异不显著(P > 0.05)。这说明河流本身有一定的自净功能[19],能一定程度上降低水体中残留的噬菌体携带抗性基因。意外的是,研究发现猪场废水处理系统出水中噬菌体携带抗性基因的平均丰度远低于河流上下游的平均丰度(P < 0.05),达到1个数量级的差异,表明猪场废水处理系统出水不是周边河流噬菌体携带抗性基因的主要污染源。前期研究也表明,猪场废水处理系统出水中残留的细菌抗性基因丰度显著低于周边河流中的丰度(P < 0.05),未对周边河流造成显著影响[10]。目前,我国典型的猪场废水处理系统末端都有深度处理工艺,系统出水在外排到外界环境之前会经过化学物理等深度处理,虽然不能完全去除出水中残留的抗性基因,但能杀死大部分的细菌及噬菌体等微生物,从而能有效减低出水中细菌及噬菌体携带抗性基因的丰度,降低了其对周边环境安全的污染风险[23-24]。但是个别噬菌体携带抗性基因,如ermB和tetW,在废水处理系统出水中的丰度高于周边河流中的丰度,对周边环境的安全仍存在较大的威胁,需要加以重视。
本研究结果发现不同猪场废水处理系统及周边河流中噬菌体携带抗性基因的分布特征具有显著差异,如猪场E废水处理系统的出水及周边河流中噬菌体携带抗性基因blaTEM的丰度显著高于其他处理系统(P < 0.05),猪场C的出水及周边河流中ermB的丰度显著高于其他系统(P < 0.05),而猪场D的出水及周边河流中sul2的丰度显著低于其他系统(P < 0.05)。影响抗性基因分布的因子很多,如抗生素、重金属和消毒水等污染物[1, 25]。不同猪场废水处理系统及周边河流中环境因子的不同导致其抗性基因分布的差异。在此需要特别注意的是,规模化猪场和中小型猪场所使用的废水处理设施与管理水平有一定的差异,可能会造成其出水的水质及抗性基因残留水平差异。本研究仅评估了规模化猪场废水处理系统出水对周边河流的影响,中小型猪场废水处理系统出水中残留的噬菌体携带抗性基因及对周边环境的污染风险需要进一步评估。以上结果说明,噬菌体携带抗性基因在不同处理系统及环境之间的污染差异增加了耐药性防控的复杂性,需要针对不同抗性基因污染的环境设计不同的防控方案。
4 结论(1)典型猪场废水处理系统出水中常见噬菌体携带抗性基因污染普遍存在,表明猪场废水处理系统未能完全去除废水中残留的噬菌体携带抗性基因,进一步凸显了猪场及周边环境中抗性基因污染问题的严重性。
(2)猪场废水处理系统周边河流中噬菌体携带抗性基因的检测率及丰度显著高于其废水处理系统出水中的检出率及丰度(P < 0.05),表明典型猪场废水处理系统出水中残留的噬菌体携带抗性基因未对周边河流造成显著影响。
| [1] |
Cheng D, Hao N H, Guo W, et al. Contribution of antibiotics to the fate of antibiotic resistance genes in anaerobic treatment processes of swine wastewater:A review[J]. Bioresource Technology, 2020, 299: 122654. |
| [2] |
Yang Y, Liu Z, Xing S, et al. The correlation between antibiotic resistance gene abundance and microbial community resistance in pig farm wastewater and surrounding rivers[J]. Ecotoxicology and Environmental Safety, 2019, 182: 109452. |
| [3] |
Jia S, Zhang X, Miao Y, et al. Fate of antibiotic resistance genes and their associations with bacterial community in livestock breeding wastewater and its receiving river water[J]. Water Research, 2017, 124: 259-268. DOI:10.1016/j.watres.2017.07.061 |
| [4] |
Rodríguez-Chueca J, Varella Della Giustina S, Rocha J, et al. Assessment of full-scale tertiary wastewater treatment by UV-C basedAOPs:Removal or persistence of antibiotics and antibiotic resistance genes?[J]. Science of the Total Environment, 2019, 652: 1051-1061. DOI:10.1016/j.scitotenv.2018.10.223 |
| [5] |
Sui Q, Chen Y, Yu D, et al. Fates of intracellular and extracellular antibiotic resistance genes and microbial community structures in typical swine wastewater treatment processes[J]. Environment International, 2019, 133: 105183. DOI:10.1016/j.envint.2019.105183 |
| [6] |
Zhang Y, Snow D D, Parker D, et al. Intracellular and extracellular antimicrobial resistance genes in the sludge of livestock waste management structures[J]. Environmental Science & Technology, 2013, 2070610513. |
| [7] |
Hao H, Shi D Y, Yang D, et al. Profiling of intracellular and extracellular antibiotic resistance genes in tap water[J]. Journal of Hazardous Materials, 2019, 365: 340-345. |
| [8] |
Brown-Jaque M, Calero-Cáceres W, Muniesa M. Transfer of antibiotic-resistance genes via phage-related mobile elements[J]. Plasmid, 2015, 79: 1-7. DOI:10.1016/j.plasmid.2015.01.001 |
| [9] |
Marti E, Variatza E, Balcazar J L. Bacteriophages as a reservoir of extended-spectrum beta-lactamase and fluoroquinolone resistance genes in the environment[J]. Clinical Microbiology and Infection, 2014, 20(7): 456-459. DOI:10.1111/1469-0691.12446 |
| [10] |
杨亦文, 米见对, 邹永德, 等. 广东省规模化猪场猪粪中典型耐药基因污染分析[J]. 中国兽医学报, 2018, 38(8): 1559-1567. YANG Yi-wen, MI Jian-dui, ZOU Yong-de, et al. Analysis of typical antibiotic resistance genes in the manure of large scale swine farms in Guangdong Province[J]. Chinese Journal of Veterinary Science, 2018, 38(8): 1559-1567. |
| [11] |
Aminov R I, Chee-Sanford J C, Garrigues N, et al. Development, validation, and application of PCR primers for detection of tetracycline efflux genes of gram-negative bacteria[J]. Applied & Environmental Microbiology, 2002, 68(4): 1786. |
| [12] |
Huang M, Zhang W, Liu C, et al. Fate of trace tetracycline with resistant bacteria and resistance genes in an improved AAO wastewater treatment plant[J]. Process Safety and Environmental Protection, 2015, 93: 68-74. DOI:10.1016/j.psep.2014.04.004 |
| [13] |
Aminov R I, Garriguesjeanjean N, Mackie R I. Molecular ecology of tetracycline resistance:Development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins[J]. Applied & Environmental Microbiology, 2001, 67(1): 22-32. |
| [14] |
Pei R, Kim S, Carlson K H, et al. Effect of river landscape on the sediment concentrations of antibiotics and corresponding antibiotic resistance genes (ARG)[J]. Water Research, 2006, 40(12): 2427-2435. DOI:10.1016/j.watres.2006.04.017 |
| [15] |
Erik K, Jerker F, Anders J, et al. Pyrosequencing of antibiotic-contaminated river sediments reveals high levels of resistance and gene transfer elements[J]. Plos One, 2011, 6(2): e17038. |
| [16] |
Xi C, Zhang Y, Marrs C F, et al. Prevalence of antibiotic resistance in drinking water treatment and distribution systems[J]. Applied & Environmental Microbiology, 2009, 75(17): 5714-5718. |
| [17] |
Juan L, Bing S, Jianzhong S, et al. Occurrence of chloramphenicol-resistance genes as environmental pollutants from swine feedlots[J]. Environmental Science & Technology, 2013, 47(6): 2892-2897. |
| [18] |
何良英.典型畜禽养殖环境中抗生素耐药基因的污染特征与扩散机理研究[D].广州: 中国科学院研究生院(广州地球化学研究所), 2016. HE Liang-ying. Contamination and spread of antibiotic resistance genes in the environment of representative livestock feedlots[D]. Guangzhou: Graduate School of the Chinese Academy of Sciences (Guangzhou Institute of Geochemistry), 2016. |
| [19] |
王庆鹤.典型自然河道形态结构差异对水体自净作用的关系[D].贵阳: 贵州大学, 2016. WANG Qing-he. The influence of typical structures of a natural river on water purification[D]. Guiyang: Guizhou University, 2016. |
| [20] |
Ma Z, Wu H, Zhang K, et al. Long-term low dissolved oxygen accelerates the removal of antibiotics and antibiotic resistance genes in swine wastewater treatment[J]. Chemical Engineering Journal, 2018, 334: 630-637. DOI:10.1016/j.cej.2017.10.051 |
| [21] |
Tong J, Tang A, Wang H, et al. Microbial community evolution and fate of antibiotic resistance genes along six different full-scale municipal wastewater treatment processes[J]. Bioresource Technology, 2019, 272: 489-500. DOI:10.1016/j.biortech.2018.10.079 |
| [22] |
Ravi A, Avershina E, et al. Integrons in the intestinal microbiota as reservoirs for transmission of antibiotic resistance genes[J]. Pathogens, 2014, 3(2): 238-248. |
| [23] |
Jeong J, Song W, Cooper W J, et al. Degradation of tetracycline antibiotics:Mechanisms and kinetic studies for advanced oxidation/reduction processes[J]. Chemosphere, 2010, 78(5): 533-540. DOI:10.1016/j.chemosphere.2009.11.024 |
| [24] |
Hou J, Chen Z, Gao J, et al. Simultaneous removal of antibiotics and antibiotic resistance genes from pharmaceutical wastewater using the combinations of up-flow anaerobic sludge bed, anoxic-oxic tank, and advanced oxidation technologies[J]. Water Research, 2019, 159: 511-520. DOI:10.1016/j.watres.2019.05.034 |
| [25] |
Zhao Y, Cocerva T, Cox S, et al. Evidence for co-selection of antibiotic resistance genes and mobile genetic elements in metal polluted urban soils[J]. Science of the Total Environment, 2019, 656: 512-520. DOI:10.1016/j.scitotenv.2018.11.372 |
 2020, Vol. 39
2020, Vol. 39





