2. 农业农村部环境保护科研监测所, 天津 300191
2. Agro-Environmental Protection Institute, Ministry of Agriculture and Rural Affairs, Tianjin 300191, China
在畜禽养殖过程中,抗生素因具有预防疾病、提高饲料转化率、促进动物生长等功效,在畜禽养殖业被广泛应用[1]。据统计,2013年我国抗生素使用总量约16.2万t,其中兽用抗生素占一半以上,高达52%[2]。兽用抗生素滥用会导致动物肠道内产生大量含有抗生素耐药基因(ARGs)的耐药性细菌,进而增加疾病的治疗难度[3]。现如今我国推行畜禽粪污的还田利用,而大量的耐药性细菌和耐药基因随着粪污排出动物体外[4],并以粪肥、肥水的形式进入农田土壤,最终危害人类健康[5]。
抗生素在畜禽养殖业中的长期、大量使用,导致我国畜禽环境中ARGs污染形势十分严峻。Ji等[6]在规模化猪场和牛场粪污中检测到磺胺类耐药基因(sulⅠ、sulⅡ、sulⅢ和sulA)和四环素类耐药基因(tetO、tetW、tetM和tetB),其中sulA、sul1和tetW在两种粪污中均显示较高丰度;Mu等[7]在规模化鸡场粪污中发现喹诺酮类耐药基因oqxB和大环内酯类耐药基因ermB,其绝对浓度分别达3.5×1010、2×109 copies·g-1;此外,在大规模养殖的猪场和鸡场粪污中也有研究[8]调查了氨基糖苷类、氯霉素类和万古霉素类等耐药基因,结果显示各类耐药基因在所测样本中均有检出。值得关注的是,在规模化养殖的畜禽环境中发现了与人类健康密切相关的高风险耐药基因。如有学者在部分鸡场粪污中检出了绝对丰度高达106~107 copies· g-1的blaTEM[9];同时Yang等[10-11]在规模化猪场粪污中还发现了blaOXA-1、blaTEM-1、blaGES-1等十余种β-内酰胺类耐药基因,并证实猪场粪污是高风险耐药基因blaNDM和mcr-1的重要储库。另外,近年来畜禽养殖环境中具有多重耐药性的“超级细菌”的出现[12],进一步凸显了养殖环境中细菌耐药问题的严重性。
目前,畜禽粪污中ARGs的污染问题已引起学者们的广泛关注,相关报道亦陆续增多。然而现有报道主要集中在规模化畜禽养殖环境中ARGs的污染研究,而有关家庭农场畜禽养殖环境中ARGs的污染规律及其对农田环境的影响尚不清楚,仍缺乏相关的研究数据。现如今,家庭农场畜禽养殖是我国乡村地区的主要养殖方式,具有养殖场数量多、养殖地点分散、抗生素使用不规范、粪污收集困难且缺乏后续处理设施等特点[13]。其产生的粪污未经处理或者处理不当而随意排放给农村生态环境造成了巨大压力,由此引起的环境污染日益严重,对公共健康的危害不容忽视。因此,解析家庭农场养殖粪污中ARGs污染情况及其潜在的环境风险,对于全面了解和控制我国畜禽养殖环境中ARGs的污染亦十分必要。
天津市蓟州区家庭农场具有分布广、数量多(600~700家)、养殖畜种丰富等特点,是研究家庭农场的典型区域。因此,本研究选取天津市蓟州区22家粪污还田达2年以上的具有代表性的家庭农场(8家猪场、8家牛场和6家鸡场)作为考察对象,采用实时荧光定量PCR方法对养殖粪污及施用粪肥的农田土壤中ARGs进行了全面调查。比较天津市蓟州区家庭农场不同畜种间常见ARGs的污染特征及差异性;详细分析不同畜种及其周边环境中常见ARGs的多样性及污染水平;特别针对与人类健康密切相关的高风险β-内酰胺类ARGs和多黏菌素耐药基因mcr-1,评估其在家庭农场中的分布及污染状况;探讨家庭农场畜禽粪肥的施用对农田土壤中ARGs的影响及存在的环境风险。本研究提供了家庭农场畜禽养殖环境中各类抗生素耐药基因的综合概况,为家庭农场畜禽养殖过程中兽用抗生素的管理和使用提供参考,以期减轻这些高风险耐药决定因子的传播。
1 材料与方法 1.1 试验材料试验样品于2018年冬季在天津市蓟州区(北纬39°45′~40°15′,东经117°05′~117°47′)22家典型的家庭农场内采集,包括8家猪场、8家牛场和6家鸡场,共38个粪便样品和44个土壤样品。粪便样品主要采自舍内新鲜粪便,试验土壤为施用粪肥2年以上的农田土壤,对照土壤则是在同区域未施用粪肥或少有人活动的地方采集。养殖场分布的详细信息见图 1。
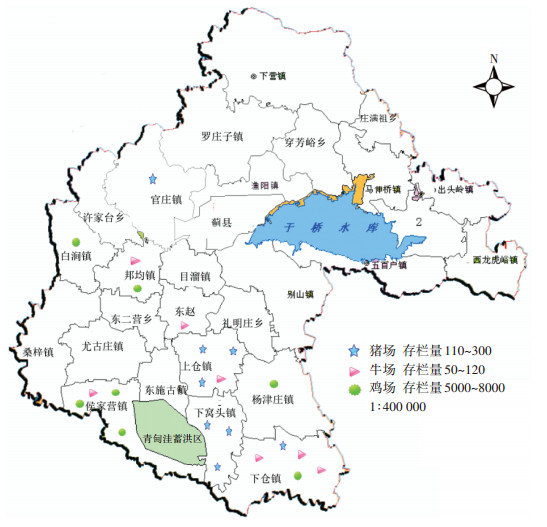
|
图 1 天津市蓟州区养殖场分布图 Figure 1 Distribution map of livestock farms in Jizhou area of Tianjin |
根据粪堆的大小分别收集3~5个粪便样本,采样方法按照《畜禽粪便监测技术规范》(GB/T 25169—2010)操作。土壤样品采用梅花采样法采集0~15 cm耕层土壤,采样方法按照《农田土壤环境质量监测技术规范》(NY/T 39—2012)操作。将粪便和土壤样品使用冷冻干燥机进行处理后,将等量的离散子样品混合,并通过2 mm的网筛制备复合样品。样品预处理完成后于-20 ℃保存至实验室,等待DNA提取。
1.3 DNA提取及PCR反应采用Fast DNA SPIN Kit for soil试剂盒(MP Biomedicals,LLC,Santa Ana,CA,美国)按操作手册对0.5 g粪便和0.5 g土壤进行DNA提取,采用超微量紫外可见光分光光度计检测DNA样品纯度。对每个DNA模板进行普通PCR检测,PCR反应体系为25 μL,包括12.5 μL 2×EasyTaq PCR SuperMix(TransGen Biotech,Beijing,China)、上下游引物各0.5 μL(10 μmol·L-1)、0.5 μL DNA模板和11 μL ddH2O。反应条件为95 ℃预变性5 min,35个循环包括95 ℃变性30 s,退火30 s(退火温度详见表 1),72 ℃延伸30 s至1 min。特异性引物详细信息见表 1。最后采用1%琼脂糖凝胶电泳检测,确定扩增产物是否含有目的基因,详细步骤见参考文献[14]。
|
|
表 1 PCR引物的详细信息 Table 1 The detail information of PCR primer |
本研究对21个ARGs进行qPCR检测,使用仪器7500实时荧光定量PCR仪(Applied Biosystems美国)进行实时qPCR分析。将提取的DNA样品稀释10倍,qPCR反应体系为20 μL,包括10.0 μL TB Green Premix Ex Taq(Tli RNase H Plus,Takara)、上下游引物各0.4 μL、0.4 μL ROX Reference Dye Ⅱ、6.8 μL ddH2O和2 μL DNA模板。两步法qPCR扩增过程如下:预变性95 ℃持续30 s,随后是40个循环包括95 ℃变性5 s,60 ℃退火/延伸持续34 s,熔点曲线分析在60~95 ℃之间进行。每个DNA模板设置3个平行,无菌水为阴性对照,qPCR反应程序和标准曲线的制作参照文献[10]。最终换算为样品中耐药基因的相对浓度(ARGs copies/16S rRNA gene copies)。
1.5 数据分析采用SPSS 22.0对数据进行ANOVA方差分析,以P < 0.05作为差异显著水平。采用R软件的pheatmap制作热图,RStudio软件的Boxplot制作箱线图,利用Origin 2017绘制了土壤分析柱状图。数据的相对丰度、平均值和标准差的计算均使用Excel 2007。
2 结果与讨论 2.1 家庭农场养殖粪污中常见耐药基因的总体污染特征及差异性为了研究家庭农场畜禽粪污中ARGs的污染特征及分布规律,本文在蓟州区选取22家典型的养殖场(包括牛场、鸡场和猪场),针对常见的5类典型抗生素的15种ARGs进行了系统的调查。从耐药基因的赋存来看(图 2),磺胺类耐药基因(sul1和sul2)、四环素类耐药基因(tetO、tetQ、tetW、tetX和tetL)、大环内酯类耐药基因(ermB和ermC)和链霉素类耐药基因(aadA、strA和strB)遍及所有样品,检出率为100%(38/38);而喹诺酮类耐药基因qnrS、qnrB和oqxB的检出率为73%(28/38)、94%(36/38)和92%(35/38),由此可见,磺胺类、四环素类、大环内酯类和链霉素类耐药基因在家庭农场畜禽粪污中存在范围最广。这主要是因为这几类抗生素具有抗菌广、见效快和价格低廉等优点,被广泛用于养殖业,导致其相应的ARGs在畜禽粪污中蓄积[20]。

|
1F~8F:育肥猪粪;1P~8P:仔猪粪;1S~8S:母猪粪;1C~8C:牛粪;1J~8J:鸡粪;PS:施用猪粪肥的土壤;CS:施用牛粪肥的土壤;JS:施用鸡粪肥的土壤;PCK:猪场对照土;CCK:牛场对照土;JCK:鸡场对照土;NULL:未检出 1F~8F: Fattening pig feces; 1P~8P: Piglet feces; 1S~8S: Sow feces; 1C~8C: Cattle feces; 1J~8J: Chicken feces; PS: Soil of pig manure; CS: Soil of cattle manure; JS: Soil of chicken manure; PCK: Control soil of pig farms; CCK: Control soil of cow farms; JCK: Control soil of chicken farms; NULL: No detected 图 2 家庭农场畜禽养殖粪污及其周边土壤中ARGs的相对丰度 Figure 2 The relative abundance of ARGs in livestock waste and surrounding soils from family farms |
值得注意的是,喹诺酮类耐药基因(oqxB、qnrS和qnrB)在猪场粪污中不仅检出率为100%,其污染水平(10-6~10-3)也显著高于鸡场(10-7~10-4)和牛场(10-8~10-6)(P < 0.05),其原因可能是动物饲料中该类抗生素的添加方式不同,以及不同畜种间肠道菌群存在差异[21]。然而,喹诺酮类以外的其他类耐药基因在鸡场粪污中的相对丰度(10-4~10-2)却比猪场(10-5~10-2)和牛场(10-6~10-3)更为严重,这与规模化养殖场不同畜禽粪污中ARGs的赋存特征一致[8]。鸡的养殖密度大且销售周期短,使用的抗生素剂量远高于猪场和牛场;另外,与哺乳动物猪和牛相比,鸡的消化功能较差,容易造成肠道内抗生素的残留,为耐药细菌的产生提供了有利条件[22],由此导致鸡场中ARGs污染严重。此外,调查还发现在同一养殖场不同猪群的发育阶段中ARGs的多样性与丰度也有所不同,例如母猪粪中ARGs的污染水平(10-3~100)显著高于育肥猪(10-3~10-5)和仔猪(10-4~10-2)(P < 0.05),这可能与其饲料配比、使用剂量和频率的差异有关[6]。
2.2 不同家庭农场养殖粪污中常见抗生素耐药基因的污染分析不同畜禽粪污中耐药基因的相对含量如图 3所示。在鸡场粪污中(图 3A),除了喹诺酮类ARGs,其他类耐药基因均以高浓度普遍存在。其中,sul1、sul2、tetO、tetQ、tetW、tetL、tetX、strA、strB、aadA、ermB和ermC赋存水平相对较高,数量级高达10-3~100;而喹诺酮类耐药基因qnrB、qnrS和oqxB的相对丰度仅为10-7~10-4。特别值得注意是,磺胺类耐药基因sul1和大环内酯类耐药基因ermB在鸡场粪污中的丰度显著高出其他类耐药基因(P < 0.05),达到10-2~100,其污染最为严重的原因可能是sul1位于可移动遗传元件int1和int2上,可以促进sul1在微生物群落中的持久性[23];以及类似Tn1545-like转座子的移动基因元件可促进ermB在不同介质间转移,提高了其在粪污环境中的污染水平[24]。此外,关于四环素类ARGs的研究发现,表达核糖体保护蛋白机制的基因含量高于其他表达机制[25],而在本研究中表达核糖体保护蛋白机制的基因tetO、tetQ、tetW与编码外排泵基因tetL和酶修饰基因tetX的相对丰度却在同一污染水平(10-3~10-1);tetL和tetX浓度偏高的原因可能是蓟州区鸡场粪污环境与其传播机制相互作用的结果。另外,从污染水平来看,还发现家庭农场的鸡场粪污中耐药基因的丰富度高于规模化鸡场粪污,如Mu等[7]在天津市规模化鸡场中的调查结果显示四环素类耐药基因tetW和tetX丰度水平为10-2~10-1,而本研究中相应的四环素类耐药基因的最高含量却达到了100;同时sul1和ermB的污染水平也比规模化鸡场中相应耐药基因的水平高出1~2个数量级[26]。
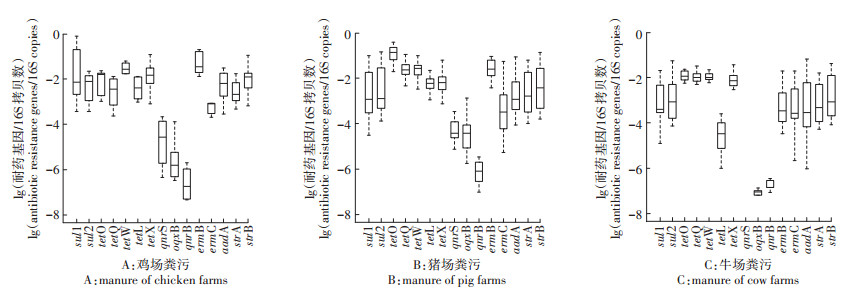
|
图 3 家庭农场中不同畜禽粪污中常见ARGs的相对丰度 Figure 3 Relative abundance of common ARGs in different livestock wastes from family farms |
与鸡场粪污中ARGs不同的是,猪场粪污中所有耐药基因包括喹诺酮类ARGs普遍存在(图 3B),且tetO、tetQ、tetW、tetX、tetL和ermB的污染相对较严重,其水平高达10-3~100。从图 2B还可以明显看出,四环素类耐药基因tetO和大环内酯类耐药基因ermB在猪场粪污中的相对水平显著高出其他类耐药基因,均达到10-2~100。其中tetO普遍存在偶联质粒上,增加了其水平转移的风险[27];ermB的宿主菌如肠球菌等在粪污中广泛分布[24],由此导致tetO和ermB含量偏高。从四环素的抗性机制角度分析,发现猪粪中编码核糖体保护蛋白基因tetO、tetQ和tetW的相对含量(10-2~100)显著高于编码外排泵基因tetL(10-3~10-2)和酶修饰基因tetX(10-3~10-1)(P < 0.05),这可能由于核糖体保护蛋白机制是动物胃肠道中四环素抗性的主导机制,且与接合质粒和整合子等密切相关造成的[27],同时该规律与Chen等[23]研究结论一致。此外,虽然喹诺酮类ARGs在猪场粪污中均有检出,但其污染水平不高,仅为10-7~10-4;qnrS和qnrB同为质粒介导的喹诺酮类耐药基因[28],而前者的污染水平(10-5~10-3)显著高于后者(10-7~10-5)(P < 0.05),其原因可能是本试验所测的区域环境不利于相应菌落的生长繁殖。有学者对规模化猪场ARGs的分析中发现,ermB、strA、sul1、tetO、tetQ、tetW和tetX的污染较严重,相对含量在10-6~10-1范围内[8, 29],而上述基因在家庭猪场中的污染水平均较高,最高达100。蓟州区家庭农户主要依靠经验养殖,养殖技术不完善、不规范和不标准,这是其污染较重的主要原因[30]。
相比于鸡场和猪场,牛场粪污中各类耐药基因的污染水平稍低,其相对丰度范围为10-6~10-(1图 3C)。四环素类耐药基因tetO、tetQ、tetW和tetX在牛场粪污中的相对丰度最高,可达10-2~10-1;磺胺类基因(sul1和sul2)、大环内酯类基因(ermB和ermC)和链霉素类基因(aadA、strA和strB)的污染水平次之,相对浓度为10-4~10-2。tetL(10-6~10-4)的相对含量显著低于其他四环素类耐药基因,原因可能是tetL在牛场粪污中的相关菌属内分布有限。此外,喹诺酮类耐药基因在牛场粪污中的污染水平相比于其他类的耐药基因亦是最低,仅为10-8~10-6,这与鸡场和猪场结果相似。原因可能是牛本身对喹诺酮类抗生素摄入较少导致粪污中的含量最低。
2.3 高风险耐药基因在家庭农场养殖粪污中的污染情况及赋存规律本研究针对家庭农场畜禽粪污中高风险的β-内酰胺类耐药基因(bla基因)和多黏菌素耐药基因mcr- 1进行了详细调查。高风险耐药基因在家庭农场畜禽粪污中的分布情况见图 2。从图中可知,blaampC、blaTEM-1和blaOXA-1在家庭农场(猪、鸡和牛)中存在普遍,检出率均为100%(38/38);此外,mcr-1和blaNDM也有较高的检出率,分别达84%(32/38)和76%(29/38);而blaGES-1的检出数量仅占总样品的39%(15/38)。以上结果表明高风险bla基因中blaampC、blaTEM-1和blaOXA-1在家庭农场畜禽粪污中较为常见,意味着这些基因及其宿主细菌在目标养殖场存在高普遍性。
图 4表示养殖粪污中高风险耐药基因的相对含量。如图所示,除blaGES-1外,3种粪污中高风险耐药基因的相对丰度大小一致为blaampC > blaTEM-1 > blaOXA-1 > blaNDM > mcr-1,与本课题组Yang等[10-11]在规模化养殖场报道的结果类似。其中blaampC、blaTEM-1和blaOXA-1在猪场、鸡场和牛场粪污中均为主导基因,且相对丰度较高,分别达10-5~10-2、10-4~10-1和10-6~10-3,与各养殖场中常见的大环内酯类耐药基因ermC的污染水平相似。牛场中blaampC、blaTEM-1和blaOXA-1的污染水平较低,其与不同畜禽的代谢能力及养殖方式的不同有着密切关系。图 2可看出,仔猪和母猪粪污中blaampC、blaTEM-1和blaOXA-1的相对含量显著高出育肥猪约1~2个数量级(P < 0.05),喂食的抗生素剂量和频率的差异导致了该结果的发生。然而,与优势基因相比较,blaNDM和mcr-1在猪场、鸡场和牛场粪污中的污染水平显著偏低(P < 0.05),分别为10-8~10-5、10-9~10-6和10-9~10-5。blaGES-1在牛场中未检出,而在猪场和鸡场的小部分样品中检出,这可能是由于相关的抗生素使用较少或牛场粪污环境不利于相应菌群生长导致。同时,Yang等[10-11]在天津市规模化猪场的研究中显示,bla基因和mcr-1的相对含量范围为10-9~10-3,而本研究中相应的高风险基因的最高含量却达到了10-1。暗示着家庭农场畜禽环境中高风险耐药基因污染问题的严重性,同时也反映出加强畜禽粪污合理化处置的必要性。

|
图 4 不同畜禽粪污中高风险ARGs的相对丰度 Figure 4 The relative abundance of high risk ARGs in different livestock manures |
因污染的影响现如今我国推行畜禽粪污的还田利用,以改良土壤和提高土壤肥力。为了确定家庭农场的畜禽粪肥对农田土壤中耐药基因的影响,本研究还对长期施用粪肥的农田土壤展开了系统的调查。结果如图 2所示,在施用有机肥的土壤中抗生素耐药基因普遍存在,其中磺胺类(sul1和sul2)、四环素类(tetO、tetQ、tetW、tetL和tetX)、大环内酯类(ermB和ermC)、链霉素类(strA、strB和aadA)和高风险的β -内酰胺类(blaampC、blaTEM-1和blaOXA-1)耐药基因在所用样品中均有检出,其中相对含量最高的3种耐药基因为sul1、sul2和tetO,均在10-4~10-2范围内;另外,虽然高风险基因中blaNDM和mcr-1相对水平较低,为10-7~10-5,但其在土壤中亦分布广泛,检出率分别达78%和75%。土壤中ARGs的高检出表明它们在土壤环境中存在高稳定性,应引起广泛注意。喹诺酮类耐药基因qnrS、qnrB和oqxB检出率最低,仅分别为27%、34%和52%。该类耐药基因在畜禽粪污中含量低是导致这一结果的原因之一,其二也可能是因为粪污的堆肥过程削减了部分喹诺酮类耐药基因,但该想法需要进一步验证。这说明粪肥施用对农田土壤中喹诺酮类耐药基因的赋存影响不大。
不同畜禽粪污的处理对农田土壤中ARGs的影响不同。通过对比未施用粪肥的对照土壤,分析粪肥施用的农田土壤中耐药基因的富集情况,结果如图 5所示。施用鸡粪肥后的土壤中ARGs的相对丰度增加了18倍,而施用猪粪肥和牛粪肥则分别增加了14倍和8倍。由上述可得,3种粪肥的施入对土壤中ARGs影响依次为鸡粪肥 > 猪粪肥 > 牛粪肥。该结果与不同动物粪污中ARGs的污染规律相一致,表明家庭农场粪污中ARGs的差异是造成不同粪肥土壤中ARGs污染差异的主要原因;同时不同粪肥土壤中寄主细菌的多样性和生存能力的差异,也可导致ARGs的持久性不同[8, 31]。显然,畜禽粪肥的施加可显著增加土壤环境中抗生素耐药基因的丰度,这与以往的研究结果相一致[32-33]。此外,不同施肥土壤中相同基因的含量也有所不同(图 2),如鸡粪肥土壤中sul1、emrB和blaOXA-1的相对丰度(10-2~100)显著高于施用猪粪肥(10-3~10-2)和牛粪肥土壤(10-4~10-3)(P < 0.05),这表明同种耐药基因在不同土壤中的持久性也有所不同。综上分析,除了喹诺酮类ARGs在3种不同土壤中很少检出,且mcr-1和blaNDM浓度较低外,其他基因在施肥后的土壤中均以高浓度普遍存在,由此可充分证明家庭农场的畜禽粪污是农田土壤环境中抗生素耐药基因的一个重要污染源。因此,采用有效的粪污处理技术来控制家庭农场粪肥中耐药基因的污染问题势在必行。
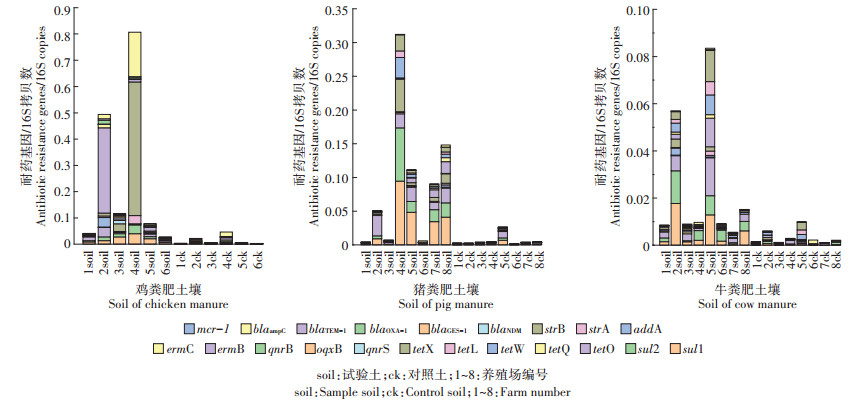
|
图 5 施用不同粪肥的土壤中ARGs的相对丰度 Figure 5 Relative abundance of ARGs in soil applied different animal manures |
(1)家庭农场畜禽养殖粪污中常见耐药基因污染较为普遍且存在多种耐药机制并存的污染特点;高风险耐药基因在畜禽粪污中的广泛分布,进一步凸显了家庭农场畜禽养殖环境中耐药基因污染问题的严重性。
(2)不同种类畜禽粪污中抗生素耐药基因的污染程度存在差异,且不同生长发育阶段对猪粪中的耐药基因污染存在影响。
(3)家庭农场畜禽粪肥的施用不仅显著增加土壤环境中耐药基因的丰度,也增大了耐药基因在农业系统中的扩散风险,同时揭示出家庭农场的畜禽粪污是农业环境中耐药基因的一个重要污染源。
(4)应加强对家庭畜禽养殖场的管理,并针对养殖密集区域集中设置粪污存储和处置设施,如粪污处理厂和有机肥加工厂,遵循源头减量、过程控制和末端利用的原则,实现粪水肥处理技术的一体化,进而降低环境中耐药基因的传播风险。
| [1] |
邓雯文, 杨盛智, 何雪萍, 等. 牛粪发酵过程中抗生素耐药基因及相关菌群组成变化规律[J]. 生态毒理学报, 2019, 14(2): 153-163. DENG Wen-wen, YANG Sheng-zhi, HE Xue-ping, et al. Change of antibiotic resistance genes and bacterial communities during dairy manure composting process[J]. Asian Journal of Ecotoxicology, 2019, 14(2): 153-163. |
| [2] |
Zhang Q Q, Ying G G, Pan C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China:Source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49: 6772-6782. |
| [3] |
Bergeron S, Raj B, Nathaniel R, et al. Presence of antibiotic resistance genes in raw source water of a drinking water treatment plant in a rural community of USA[J]. International Biodeterioration & Biodegradation, 2017, 124: 3-9. |
| [4] |
Wang F H, Qiao M, Chen Z, et al. Antibiotic resistance genes in manure-amended soil and vegetables at harvest[J]. Journal of Hazardous Materials, 2015, 299: 215-221. |
| [5] |
Chen Q, An X, Li H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92: 1-10. |
| [6] |
Ji X, Shen Q, Liu F, et al. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai, China[J]. Journal of Hazardous Materials, 2012, 235: 178-185. |
| [7] |
Mu Q, Li J, Sun Y, et al. Occurrence of sulfonamide-, tetracycline-, plasmid-mediated quinolone- and macrolide-resistance genes in livestock feedlots in Northern, China[J]. Environmental Science and Pollution Researsh, 2015, 22(9): 6932-6940. |
| [8] |
Qian X, Gu J, Sun W, et al. Diversity, abundance, and persistence of antibiotic resistance genes in various types of animal manure following industrial composting[J]. Journal of Hazardous Materials, 2018, 344: 716-722. |
| [9] |
Gómez-Gómez C, Blanco-Picazo P, Brown-Jaque M, et al. Infectious phage particles packaging antibiotic resistance genes found in meat products and chicken feces[J]. Scientific Reports, 2019, 9(1): 1-11. |
| [10] |
Yang F, Zhang K, Zhi S, et al. High prevalence and dissemination of β-lactamase genes in swine farms in northern China[J]. Science of the Total Environment, 2019, 651: 2507-2513. |
| [11] |
Yang F, Gu Y, Zhou J, et al. Swine waste:A reservoir of high-risk blaNDM and mcr-1[J]. Science of the Total Environment, 2019, 683: 308-316. |
| [12] |
刘丹华, 张晓伟, 张翀. 抗生素滥用与超级细菌[J]. 国外医药(抗生素分册), 2019, 40(1): 1-4. LIU Dan-hua, ZHANG Xiao-wei, ZHANG Chong. Antibiotic overuse and superbugs[J]. World Notes on Antibiotic, 2019, 40(1): 1-4. |
| [13] |
周琳, 杨祯妮, 程广燕. 发展畜禽养殖家庭农场的制约因素及分析——基于上海、浙江两地的调研[J]. 中国畜牧杂志, 2014, 50(20): 13-17. ZHOU Lin, YANG Zhen-ni, CHENG Guang-yan. Constraints and analysis of developing livestock and poultry family farms:Based on research in Shanghai and Zhejiang[J]. Chinese Journal of Animal Science, 2014, 50(20): 13-17. |
| [14] |
王晓慧, 张明, 任雪丽, 等. 四环素耐药基因在猪粪便、堆肥和土壤中的归趋[J]. 安徽农学通报, 2017, 23(9): 22-23, 76. WANG Xiao-hui, ZHANG Ming, REN Xue-li, et al. Transport and fate of tetracycline resistance genes in pig manure, compost and soil[J]. Anhui Agricultural Science Bulletin, 2017, 23(9): 22-23, 76. |
| [15] |
Marti E, Balcázar J L. Real-time PCR assays for quantification of qnr genes in environmental water samples and chicken feces[J]. Applied and Environmental Microbiology, 2013, 79(5): 1743-1745. |
| [16] |
Johnson E A. Monitoring foreign gene incorporation into the plastome of Chlamydomonas reinhardtii by multiplex qPCR[J]. Photosynthesis Research, 2013, 115(1): 81-87. |
| [17] |
Walsh F, Ingenfeld A, Zampicolli M, et al. Real-time PCR methods for quantitative monitoring of streptomycin and tetracycline resistance genes in agricultural ecosystems[J]. Journal of Microbiological Methods, 2011, 86(2): 150-155. |
| [18] |
You Y, Hilpert M, Ward M J. Detection of a common and persistent tet (L)-carrying plasmid in chicken-waste-impacted farm soil[J]. Applied and Environmental Microbiology, 2012, 78(9): 3203-3213. |
| [19] |
Ghosh S, Ramsden S J, LaPara T M. The role of anaerobic digestion in controlling the release of tetracycline resistance genes and class 1 integrons from municipal wastewater treatment plants[J]. Applied Microbiology and Biotechnology, 2009, 84(4): 791-796. |
| [20] |
闫书海.畜禽养殖废水/粪便中典型抗药基因的调查研究[D].杭州: 浙江大学, 2013. YAN Shu-hai. Research on representative antibiotic resistance genes in livestock lagoons and manures[D]. Hangzhou: Zhejiang University, 2013. http://cdmd.cnki.com.cn/Article/CDMD-10335-1013153363.htm |
| [21] |
Warburton P J, Amodeo N, Roberts A P. Mosaic tetracycline resistance genes encoding ribosomal protection proteins[J]. Journal of Antimicrobial Chemotherapy, 2016, 71: 3333-3339. |
| [22] |
张昊, 王盼亮, 杨清香, 等. 畜禽粪便中多重耐药细菌及耐药基因的分布特征[J]. 环境科学, 2018, 39(1): 460-466. ZHANG Hao, WANG Pan-liang, YANG Qing-xiang, et al. Distribution of multidrug-resistant bacteria and antibiotic-resistant genes in livestock manures[J]. Environmental Science, 2018, 39(1): 460-466. |
| [23] |
Chen B, Hao L, Guo X, et al. Prevalence of antibiotic resistance genes of wastewater and surface water in livestock farms of Jiangsu Province, China[J]. Environmental Science and Pollution Research International, 2015, 22(18): 13950-13959. |
| [24] |
Montanari M P, Cochetti I, Mingoia M, et al. Phenotypic and molecular characterization of tetracycline- and erythromycin-resistant strains of Streptococcus pneumoniae[J]. Antimicrobial Agents and Chemotherapy, 2013, 47: 2236-2241. |
| [25] |
Li C, Jiang C, Wu Z, et al. Diversity of antibiotic resistance genes and encoding ribosomal protection proteins gene in livestock waste polluted environment[J]. Journal of Environmental Science and Health, Part. B, 2018, 53(7): 423-433. |
| [26] |
邓雯文, 陈姝娟, 何雪萍, 等. 鸡粪-堆肥中重金属残留、抗生素耐药基因及细菌群落变化研究[J]. 农业环境科学学报, 2019, 38(2): 439-450. DENG Wen-wen, CHEN Shu-juan, HE Xue-ping, et al. Dynamics of heavy metal residues, antibiotic resistance genes, and bacterial communities during chicken manure composting[J]. Journal of Agro-Environment Science, 2019, 38(2): 439-450. |
| [27] |
Liu L, Liu Y H, Wang Z, et al. Behavior of tetracycline and sulfamethazine with corresponding resistance genes from swine wastewater in pilot-scale constructed wetlands[J]. Journal of Hazardous Materials, 2014, 278: 304-310. |
| [28] |
Cheng W, Chen H, Su C, et al. Abundance and persistence of antibiotic resistance genes in livestock farms:A comprehensive investigation in eastern China[J]. Environment International, 2013, 61: 1-7. |
| [29] |
杨亦文, 米见对, 邹永德, 等. 广东省规模化猪场猪粪中典型耐药基因污染分析[J]. 中国兽医学报, 2018, 38(8): 1559-1567. YANG Yi-wen, MI Jian-dui, ZOU Yong-de, et al. Analysis of typical antibiotic resistance genes in the manure of large scale swine farms in Guangdong Province[J]. Chinese Journal of Veterinary Science, 2018, 38(8): 1559-1567. |
| [30] |
周德强. 找差距补短板, 提升家庭农场畜禽养殖生产效益[J]. 中国畜禽种业, 2019, 15(8): 34-35. ZHOU De-qiang. Find gaps to fill short boards and improve the efficiency of livestock farming in family farms[J]. The Chinese Livestock and Poultry Breeding, 2019, 15(8): 34-35. |
| [31] |
Zhou X, Qiao M, Wang F H, et al. Use of commercial organic fertilizer increases the abundance of antibiotic resistance genes and antibiotics in soil[J]. Environmental Science and Pollution Research International, 2017, 24(1): 701-710. |
| [32] |
Xie S, Wu N, Tian J, et al. Review on the removal of antibiotic resistance genes from livestock manure by composting[C]//IOP Conference Series: Earth and Environmental Science, IOP Publishing, 2019, 237(5): 052010.
|
| [33] |
田甜甜, 王瑞飞, 杨清香. 抗生素耐药基因在畜禽粪便-土壤系统中的分布、扩散及检测方法[J]. 微生物学通报, 2016, 43(8): 1844-1853. TIAN Tian-tian, WANG Rui-fei, YANG Qing-xiang. Distribution, spread and detection methods of antibiotic resistance genes in livestock manure and soil system[J]. Microbiology China, 2016, 43(8): 1844-1853. |
 2020, Vol. 39
2020, Vol. 39



