2. 国家农业环境大理观测实验站, 云南 大理 671004
2. Dali Observation and Experimental Station of National Agricultural Environment, Dali 671004, China
随着规模化养殖业的快速发展,抗生素滥用导致的抗生素耐药基因(Antibiotic resistance genes,ARGs)污染问题日益突出[1-2]。目前,粪污还田实现种养循环是各级农业部门主推的粪污处理模式,因此ARGs不可避免地发生水平转移而随施肥进入农田,进而对土壤环境造成严重污染[3-4]。研究表明,粪肥施用可增加农田土壤中耐药基因的丰度,如在施用鸡粪肥的土壤中,喹诺酮类ARGs(qnrA、qnrD和oqxB)和大环内酯类ARGs(ermA、ermB)比不施肥的对照土中同类ARGs的丰度最高可增加4个数量级[5]。LIU等[6]在猪粪、鸡粪和牛粪重复连续施用的农田土壤中发现ARGs丰度及多样性呈上升趋势。土壤环境中含有大量ARGs的耐药菌可能会通过水平转移由内生菌进入植物内部,从而将外源ARGs转移至植物的内生系统。现已在新鲜果蔬中发现的大量ARGs及多种相关整合子,增加了其通过食物链向人类传播的风险[7]。
不同施肥模式可显著影响ARGs在土壤中的积累和传播。研究表明,施加化肥、粪肥可使ARGs水平相较不施肥土壤增加[8-9]。尤其是粪肥施用能显著增加土壤中ARGs的丰度水平,引起土壤环境微生物抗性水平的提高。科学施用有机肥可能是限制ARGs在农田传播的有效途径。然而,目前对不同施肥模式下农田土壤ARGs的分布模式及传播研究较少,且长期施用粪肥对不同土壤深度ARGs垂直迁移的影响尚未得到重视,即缺乏不同施肥方式下不同土层中ARGs迁移富集的研究。因此,解析不同施肥方式下,农田土壤不同深度中ARGs的积累分布规律及其潜在环境风险,对分析施肥方式对土壤中ARGs的污染及传播十分必要,同时也可进一步认识不同施肥处理模式对农田生态系统的影响。本研究选取云南大理市洱海流域某水稻田作为研究对象,在减氮条件下设置施用化肥、施用有机肥、施用缓控释肥处理,通过田间小区试验,探究土壤中各类ARGs在长期定量不同施肥方式下的组成及丰度变化特征,以及在受纳土壤不同土层中的迁移富集规律,并统计ARGs与可移动遗传元件(MGEs)的相关关系,分析土壤中ARGs多样性程度与丰度水平差异的影响因素及环境风险。本研究从ARGs防控角度为水稻肥料施用配比及用量提供科学依据和应用参考。
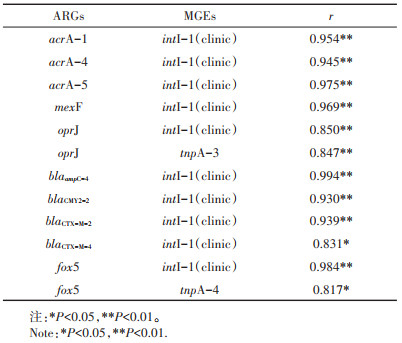
1 材料与方法 1.1 样品采集及处理长期定位试验田位于云南省大理市农业农村部环境保护科研监测所大理综合实验站内(25°53′ 34″ N,100°10′ 27″ E)。水稻试验采用完全随机区组设计,共8个处理,即(1)CK:不施肥;(2)CF:常规施肥,尿素和普钙用量为424 kg·hm-2和450 kg·hm-2;(3)T1:常规施肥减量20%,尿素和普钙用量分别为339 kg·hm-2和375 kg·hm-2;(4)T2:粪肥等氮替代T1,粪肥用量为6 500 kg·hm-2;(5)T3:粪肥等磷替代T1,尿素补齐氮肥,粪肥和尿素用量分别为2 609 kg·hm-2和203 kg·hm-2;(6)T4:考虑粪肥矿化率25%,以氮计粪肥替代T1,粪肥用量为26 000 kg·hm-2;(7)T5:考虑粪肥矿化率25%,以磷计有机肥替代T1,尿素补齐氮肥,粪肥和尿素用量分别为10 435 kg·hm-2和203 kg· hm-2;(8)T6:缓控释肥,缓控释肥用量为678 kg·hm-2。每个处理3次重复,共24个小区,种植方式和农田管理方式相同。2019年10月水稻收获后采集土壤样品,分别按0~20、20~50 cm和50~80 cm不同剖层采集。每个小区采取梅花采样法,现场均匀混合,去除水稻根和可见有机物残体后,采取四分法,留样1 kg左右,并将土样保存于-20 ℃冰箱,待DNA提取。
1.2 土壤样品DNA的提取称取约0.5 g土壤样品,所有样品采用Fast DNA SPIN Kit for soil试剂盒(MP Bio-medicals,LLC,Santa Ana,CA,美国)并按生产商提供的操作手册进行总DNA提取,每个土壤样品3个重复。提取后用1.0% 琼脂糖凝胶电泳验证DNA的质量。提取的DNA样品浓度使用超微量核酸蛋白检测仪(Nanodrop 2000,德国)进行测定,所提取的DNA样品的A260/A280值在1.8左右。提取后的DNA样品置于-20 ℃的冰箱中保存,用于后续的检测分析。
1.3 高通量荧光定量PCR与实时荧光定量检测方法使用WafergenSmartchip超高通量荧光定量PCR系统检测ARGs及MGEs,共设置144对引物,其中包括1对16S rRNA内参引物。反应体系为1×LightCy- cler 480 SYBR Gree I Master;引物浓度为500 nmol· L-1;DNA模板2 ng·µL-1,扩增反应体系为100 nL。反应程序:95 ℃预变性10 min;95 ℃变性30 s,60 ℃退火处理,30 s,40个循环;最后系统自动进行熔解曲线分析。使用7500实时荧光定量PCR仪(Applied Biosystems,美国)对16S rRNA进行实时qPCR分析。反应体系为20 µL,包括16S rRNA上下游引物各0.4 µL、ddH2O 6.8 µL、TB Green Premix Ex Taq(Tli RNase H Plus,Takara)10.0 µL、ROX Reference Dye Ⅱ 0.4 µL以及DNA模板2 µL。qPCR扩增过程:95 ℃预变性30 s;然后为40个循环,包括95 ℃变性5 s和60 ℃退火持续34 s,熔解曲线分析在60~95 ℃之间进行。其中无菌水为阴性对照,每个DNA模板设置3个平行,qPCR反应程序和标准曲线的制作参照文献[10]。
1.4 数据分析使用Excel 2007计算数据的绝对丰度、相对丰度、平均值和标准差等。采用SPSS 22.0对数据进行相关分析和ANOVA方差分析,P < 0.05表示差异显著。采用Origin 2021软件绘制柱状图、热图和维恩图。
利用标准曲线计算16S rRNA基因的绝对拷贝数,经线性拟合分析可知其与高通量定量PCR检测出的16S rRNA的相对拷贝数有较好的线性关系(r= 0.956),且极显著相关(P < 0.01),故可以使用公式(1)[11]和公式(2)计算ARGs的绝对丰度,进一步ARGs的相对丰度通过公式(3)可计算得到。
基因的相对拷贝数=10(31-CT)/(10/3)(1)
ARGs的绝对丰度=ARGs的相对拷贝数/16S rRNA基因的相对拷贝数×16S rRNA基因的绝对拷贝数(2)
ARGs的相对丰度=基因的相对拷贝数/16S rRNA基因的相对拷贝数(3)
式中:ARGs是所要测定的144种抗生素抗性基因;CT是高通量荧光定量所测定的循环阈值。
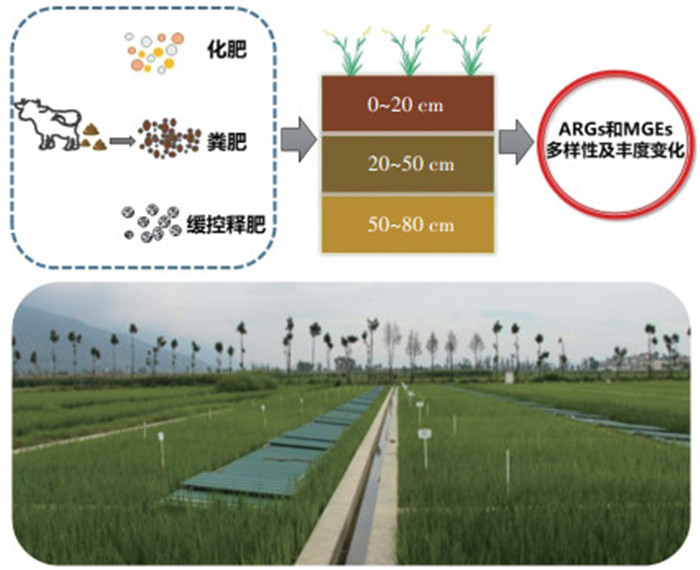
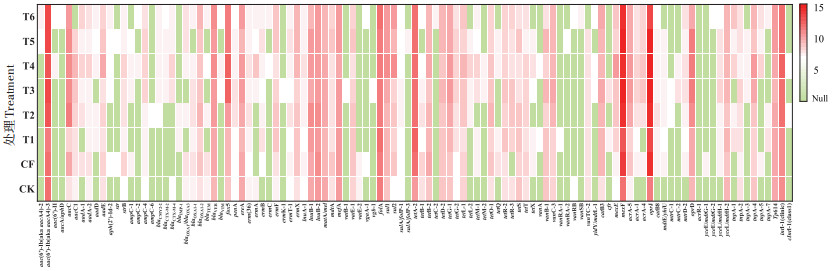
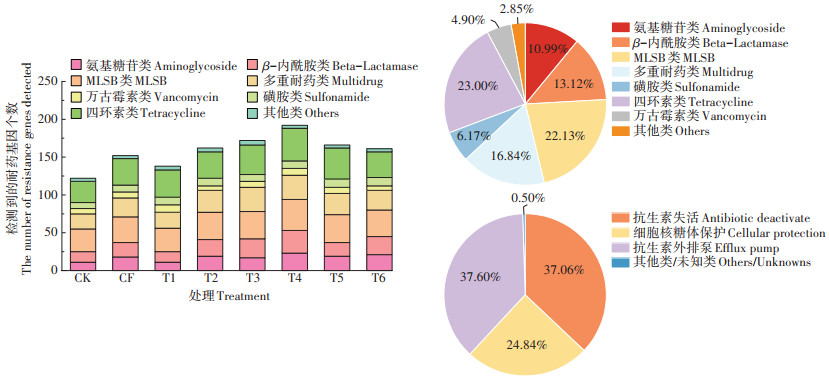
2 结果与讨论 2.1 长期施用粪肥稻田土壤中抗生素耐药基因的赋存多样性特征选取7大类144种常见的ARGs及10种与ARGs传播密切相关的MGEs,对其在长期施用粪肥的稻田土壤中的存在情况进行了分析检测,结果如图 1所示。在长期施肥土壤中,共检出100种ARGs亚型和9种MGEs编码基因(7种转座子基因和2种整合子基因)。从不同类别与不同作用机制来看,水稻土中检测到的ARGs包含了目前已知且常见的ARGs类型,主要包括氨基糖苷类aac-ARGs(13种)、β-内酰胺类bla-ARGs(16种)、磺胺类sul-ARGs(5种)、大环内脂类林肯酰胺类链阳性菌素MLSB-ARGs(20种)、多重耐药类MDR-ARGs(9种)、四环素类tet-ARGs(18种)和万古霉素类van-ARGs(8种)等7大类耐药基因,另外还有11种其他类的ARGs。其中,四环素类ARGs和大环内脂类林肯酰胺类链阳性菌素B(MLSB)的ARGs在水稻土中检出较多,分别达23.00% 和22.13%);其次是与人类危害息息相关的多重耐药类和β-内酰胺类抗生素ARGs[12-15],占比达16.84% 和13.12%,值得引起注意。氨基糖苷类ARGs检出占比为10.99%;磺胺类和万古霉素类的数量较少,占比仅为6.17% 和4.90%。所有水稻土处理中检测到ARGs的抗生素抗性机制主要是抗生素失活(Antibiotic deactivation)、抗生素外排泵(Efflux pump)和细胞核糖保护(Cellular protection),这3类抗生素抗性机制在整体中的比例分别为37.06%、37.60% 和24.84%,还有0.50%的其他或未知抗性机制(图 2)。
|
图 1 稻田土壤中ARGs的绝对丰度 Figure 1 The absolute abundance of ARGs in paddy soil |
|
图 2 稻田土中ARGs的检出种类 Figure 2 Profiles of detected ARGs in paddy soil |
值得注意的是,不同施肥模式下,稻田土壤中ARGs的赋存多样性存在差异,其中施用粪肥土壤(T2~T5处理)中ARGs的多样性最为丰富,其检出种类可达88种;其次为长期施用缓控释肥的T6处理土壤(76种)和施用化肥(CF和T1处理)的稻田土壤(74种);而在CK土壤中也存在多种ARGs,种类亦达63种。同ARGs污染多样性特征相似,在施用粪肥量最大的T4处理中检测到的MGEs种类最多,达7种,显著高于其他施肥类型的土壤。由维恩图(图 3)可以看出,有55种(50.5%)ARGs亚型为6种不同施肥模式稻田土壤中的核心ARGs(即共现ARGs)。其中,CK处理中ARGs的总检出数量最少,基因多样性最低;另外3种施用肥料的处理中ARGs的总检出数量均有增多。施用粪肥的处理中ARGs的总检出数量最多,其次是施用缓控释肥和施用化肥的处理,分别是CK处理的1.3、1.2倍和1.1倍,均显著高于CK水稻土(P < 0.05)。施用化肥的处理中,T1处理的ARGs数量与CF处理相比有所降低。施用粪肥水稻土中独有的ARGs有7个(blaampC-6、vatB-1、sulA/folP-3、tetL-2、tetM-2、vanRA-2、mtrC-2)。从ARGs的检出数量来看,在4种不同用量的粪肥替代处理组中,T4处理田块中ARGs的总检出量最高,所有样品中总共有210个ARGs;其次是T3处理,检出185个ARGs;T2处理和T5处理的检出量相差较少,分别为176个和179个ARGs。施用粪肥土壤中的ARGs相对于CK土壤具有更复杂的多样性,施用粪肥显著改变了稻田土壤中的ARGs种类组成(P < 0.05)。
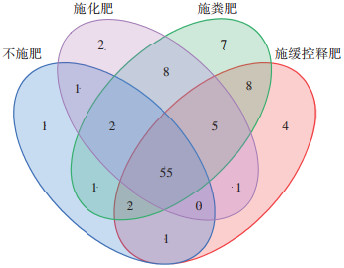
|
图 3 不同施肥模式ARGs的维恩图 Figure 3 Venn diagram of ARGs in different fertilization patterns |
不同施肥模式下,稻田土壤中ARGs的污染水平见图 4。结果显示,不同处理模式下的稻田土壤ARGs污染水平在1.0×107~1.3×1010 copies·g-1的范围内,且不同施肥模式的稻田土壤中ARGs的存在水平不同,整体呈现出施用粪肥>施用缓控释肥>施用化肥>不施肥的趋势,这与之前的研究结果一致[16-17]。在4种施用粪肥的处理模式中,施用粪肥较多的T3处理中ARGs的丰度水平最高,ARGs的总丰度达9.6×1010 copies·g-1;其次为T4和T5处理,ARGs总丰度分别达7.6×1010 copies·g-1和7.3×1010 copies·g-1;施用粪肥量较少的T2处理中ARGs的总绝对丰度最低,是T3处理的60%,其相应的ARGs种类数也较低。从ARGs的类别来看,不同施肥模式下稻田土壤中的优势基因主要包括多重耐药类和氨基糖苷类,且各模式稻田中污染水平最高的类型均为多重耐药类,占ARGs总丰度的62.9%~77.4%,为1.1 × 107~1.3 × 1010 copies ·g-1;其次为氨基糖苷类,占ARGs总丰度的8.2%~12.3%,为1.0×107~3.3×109 copies·g-1。而四环素类和MLSB类ARGs在各处理稻田土壤中总丰度的占比较低,分别为4.0%~9.2% 和3.0%~8.2%。此外,虽然与人类健康密切相关的β-内酰胺类和万古霉素类ARGs在所有稻田处理中的丰度水平较低,但其检出率较高,分布较为广泛,亦应引起重视。
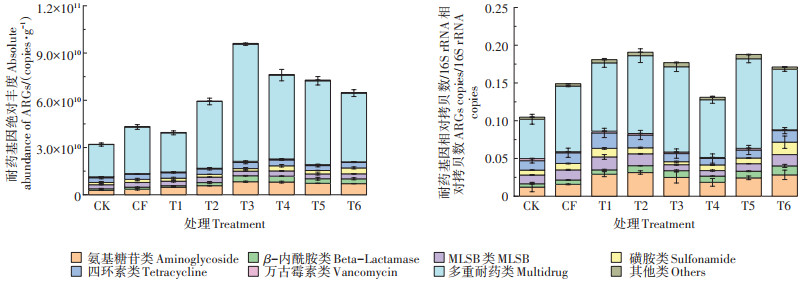
|
图 4 不同施肥模式稻田土中各类ARGs的污染水平 Figure 4 Pollution levels of different ARGs types in paddy soils under different fertilization modes |
为了规避不同处理中微生物量对ARGs的影响,又进一步从ARGs的相对丰度分析了不同模式下ARGs的污染水平,结果见图 4。当不同施肥土壤中ARGs的丰度水平统一平均到每个细胞中ARGs的拷贝数,即将ARGs的丰度归一到单个细菌水平时,所有处理稻田土壤中ARGs的相对丰度水平在1.7× 10-5~3.6×10-2范围内。在施用粪肥的4个处理模式中,T4处理稻田土壤中的ARGs总相对丰度最低,为0.13,这可能是由于T4处理较大的粪肥施肥量显著增加了土壤中的微生物总量,从而使得ARGs的相对丰度低于其他粪肥施用量较少的处理;而T3处理总相对丰度水平稍高,是T4处理的1.4倍;而T2和T5处理中的ARGs总相对丰度水平相似,分别为0.19和0.18。综上可见,施用粪肥处理的相对丰度水平与绝对丰度的赋存规律不一致,这主要是因为不同施肥模式下,稻田土壤中微生物量的不同所致。从相对丰度的角度来看,稻田土的主导ARGs类型与绝对丰度处理的规律一致,亦为多重耐药类、氨基糖苷类和四环素类。丰度最高的多重耐药类ARGs在T5处理中相对丰度最高(7.4×10-5~2.5×10-2),而氨基糖苷类和四环素类ARGs则分别在施用粪肥的T2处理和减量施用化肥的T1处理中丰度最高,分别为6.2×10-5~2.1× 10-2和8.9×10-5~8.3×10-3。MLSB类和磺胺类ARGs在所有处理中分别占总丰度的4.5%~11.1% 和2.3%~ 9.6%,分别在T1和T6处理中相对丰度最高;与人类关系密切的β-内酰胺类在各处理水稻田中也有检出,但总丰度占比较低,为3.2%~6.9%;万古霉素类的相对丰度在各处理中差异不显著(P>0.05),且丰度水平最低。
相比于CK处理的稻田土壤,施用化肥、粪肥以及缓控释肥均明显增加了稻田土壤中ARGs的多样性与丰度。其中,在施用粪肥处理中,ARGs的丰度水平较施用化肥处理也有较大程度的增加,多重耐药类、氨基糖苷类以及β-内酰胺类ARGs水平增加较为明显。不同施用粪肥处理中ARGs含量的高低水平在绝对丰度与相对丰度中存在一定差异,绝对丰度较高的T4处理,其相对丰度水平最低,这可能是由于T4处理施用粪肥量较大,土壤中微生物量(16S rRNA)显著增加,从而使得归一后的相对丰度水平降低。与WANG等[8]的研究结果相似,施用化肥或粪肥的土壤与不施肥土壤相比,能够不同程度地提高ARGs的多样性及丰度。
2.3 长期不同施肥模式下稻田土壤中耐药基因的垂直积累分布规律为了明确长期施肥稻田土壤中ARGs的垂直积累特征,本研究又进一步调查了各施肥模式下不同土层中ARGs的积累富集规律。调查结果显示,在不同施肥模式各土层中含量较高的ARGs依然是多重耐药类、氨基糖苷类以及四环素类,但不同模式各土层中ARGs的迁移富集规律不同。如在长期施用粪肥的4个处理模式中,稻田土壤中ARGs的绝对丰度总体呈现由表层向深层土壤递减的趋势,这与PAN等在污水灌溉农田不同土层中ARGs的丰度变化一致[18]。且β-内酰胺类、磺胺类、多重耐药类和MLSB类ARGs在耕层(0~20 cm)中的丰度分别是底层50~ 80 cm的2.3、1.8、1.7倍和1.5倍。此外,在施用粪肥量最大的T4处理中,各类ARGs的绝对丰度从稻田土表层到底层逐渐减少(图 5)。而在CK稻田土中,多重耐药类ARGs在20~50 cm的丰度最高,高达1.3×1010 copies·g-1,分别是50~80 cm和0~20 cm土壤的1.7倍和48.9倍;而四环素类、磺胺类以及MLSB类ARGs在0~20 cm的丰度最高;万古霉素类、氨基糖苷类以及β-内酰胺类ARGs的绝对丰度则出现向下累积的趋势。施用化肥后的处理中丰度较高的多重耐药类、氨基糖苷类和四环素类ARGs在不同土层中的累积情况不同,氨基糖苷类在0~20 cm的丰度最高,多重耐药类则是在较深层的50~80 cm丰度最高,而四环素类在各土层中无明显丰度变化差异(P>0.05)。T6处理稻田土壤中,丰度最高的多重耐药类在20~50 cm的绝对丰度最高,与CK处理规律一致,达1.0×1010 copies·g-1,不同深度土层中的各类ARGs,大部分都在施用缓控释肥后丰度增加。
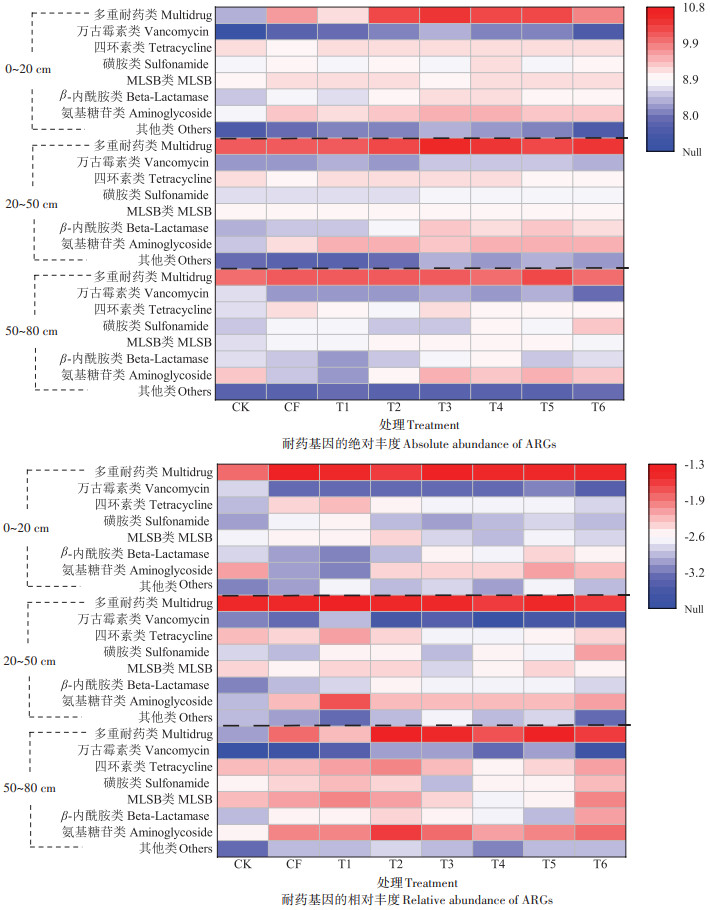
|
图 5 不同土层中各类ARGs的绝对丰度与相对丰度的分布热图 Figure 5 Heatmaps of absolute abundance and the relative abundance of ARGs in different soil layers |
而从相对丰度来看,CK、CF、T1和T6处理的土壤中,大多数ARGs在50~80 cm的深层土壤中的丰度相比0~20 cm和20~50 cm土层显著增高(P < 0.05),其与绝对丰度中20~50 cm和50~80 cm的ARGs赋存规律不一致。ARGs的相对丰度在深层土中更高的原因主要是因为深层土壤中微生物量含量较少,从而导致归一后相对丰度水平较高。施用粪肥的稻田土壤中,ARGs在50~80 cm土层中的相对丰度水平比0~20 cm高(是0~20 cm的1.2倍)。施用粪肥量较少的T2处理中ARGs的丰度相较其他施肥处理在深层50~80 cm土层中累积显著(P < 0.05)。在6个不同施肥处理模式中,心层土(30~50 cm)和底层土(60~80 cm)中ARGs的相对丰度与耕层土(0~20 cm)相比均较高,不同类型ARGs从耕层(0~20 cm)经20~50 cm土层向50~80 cm的累积速度有一定差异。不同深度的稻田土中,相对丰度呈现明显垂直向下层累积的现象,这与之前的研究结果相同[19]。可能是由于本研究于水稻收获后采集稻田土壤,已超过施用肥料1个月的时间,且经过了较长时间水稻种植的淹水时期,携带ARGs的细菌由水分的渗透作用加快了向下层土迁移的速度[20]。同时,粪肥的施用年限、土壤理化性质和相关MGEs等因素的不确定性[21],可能导致不同土层中不同种类ARGs的分布累积具有差异,部分在20~50 cm富集,部分则主要在50~80 cm。综上,施肥方式会对ARGs在不同深度土层的分布产生一定的影响,但具体的迁移机制有待进一步研究。
2.4 不同施肥模式下主导ARGs的谱图特征及其与MGEs的关系在水稻土中多重耐药类、氨基糖苷类、四环素类和β-内酰胺类ARGs为主导基因类型,占比较高;多重耐药类不仅丰度高,且其和β-内酰胺类ARGs均与人类健康密切相关,因此风险性较高。在多重耐药类ARGs的不同亚型中,mexF和oprJ的丰度在各处理中检出量均较高,其绝对丰度分别为7.3×109~2.4×1010 copies·g-1和5.6×109~2.4×1010 copies·g-1,是稻田土中流行的ARGs,很多关于农田土壤的研究结果也表明多种耐药类的丰度较高[22-24];acrA-1、acrA-4、acrA-5、catB3和mexE等基因在各处理中均有检出且丰度也较高,平均丰度达108 copies·g-1;cfr及yidY/mdtL-1的丰度较低,在施肥处理后才有检出。氨基糖苷类ARGs中,aac(6′)-Ib(aka aacA4)-3的丰度最高,其次为aacC,绝对丰度水平分别为2.5×109~5.5×109 cop- ies·g-1和8.7×107~2.4×109 copies·g-1;aacC、aacC1、aadA-1、aadA-2、aadD、aadE和strB也在水稻土中普遍存在。四环素类ARGs中,丰度最高的是tetA-2,所有处理的平均丰度均达到109 copies·g-1;其次是tetG-1和tetB-2,分别为tetA-2的19%和14%。β-内酰胺类ARGs在稻田土中丰度最高的是fox5,为2.8×108~2.6× 109 copies·g-1,blaVEB和blaOXA1-2丰度略低于fox5,平均丰度在107 copies·g-1水平;blaOXA-1/blaOXA-3、blaOXA1-1、blaOXA1-2、blaTEM和penA也在各处理中普遍存在。多重耐药类、氨基糖苷类、四环素类以及β-内酰胺类ARGs的总绝对丰度,施用化肥、粪肥以及缓控释肥的3种处理相对于未施肥土壤中均有增多,分别增加了0.4、1.6倍和1.1倍,其中施用粪肥和缓控释肥的水稻土显著富集了ARGs。
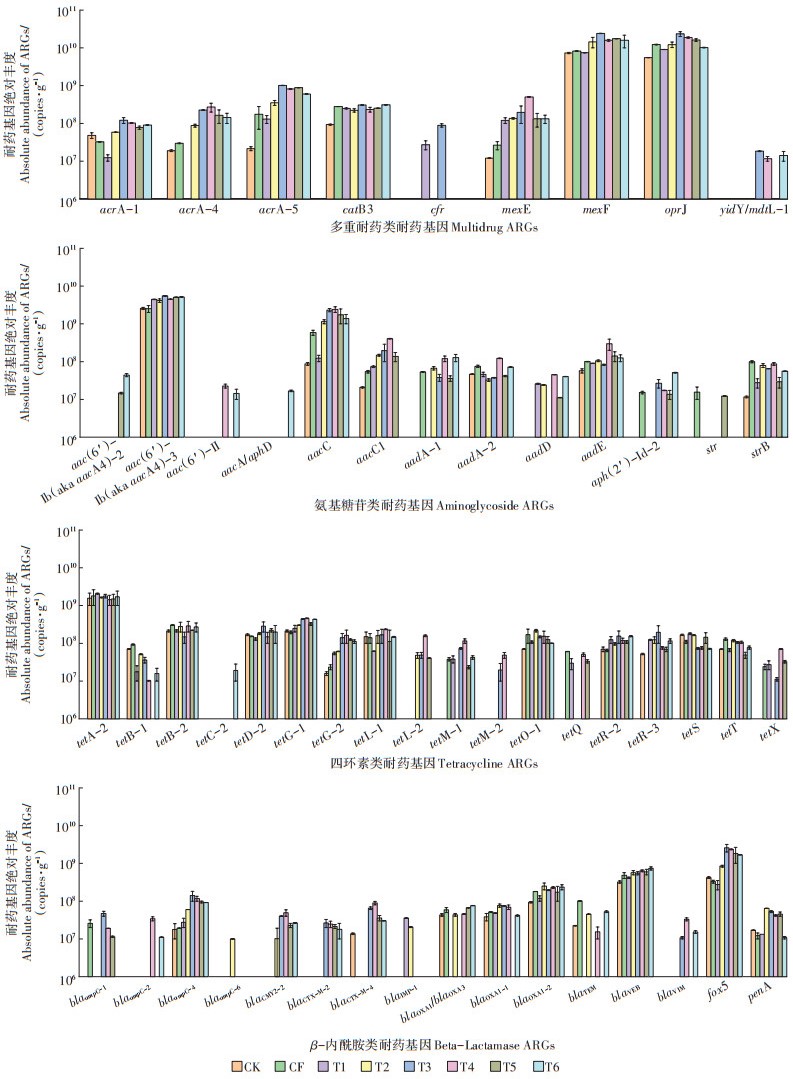
|
图 6 不同施肥处理稻田土壤中主导ARGs的分布规律 Figure 6 Distribution of dominant ARGs in paddy soils under different fertilization treatments |
研究表明,ARGs在环境微生物间的传播和转移与MEGs紧密相关[25-26],因此对ARGs的绝对丰度与MGEs之间的相关性进行分析,结果显示(表 1),部分ARGs与转座子基因、整合子基因存在显著相关性,表明MGEs可能促进了ARGs在稻田土壤中的迁移和传播。有研究发现环境微生物中的intI1(clinic)与许多ARGs有关[27],aadA、tetG、acrA-1、mexE等均与intI1(clinic)显著相关。本研究中intI-1(clinic)与多重耐药类ARGs(acrA-1、acrA-4、acrA-5、mexF和oprJ)以及β-内酰胺类(blaampC-4、blaCMY2-2、blaCTX-M-2、blaCTX-M-4和fox5)有显著正相关,进一步证明稻田土壤中intI1整合子基因对ARGs的传播转移起着重要作用。此外,转座子基因也与高丰度的基因显著相关,如tnpA-3与多重耐药类ARGs中的oprJ以及tnpA-3、tnpA-4与β-内酰胺类ARGs中的fox5。在施用粪肥及施用缓控释肥的处理中,检测到MGEs的丰度分别是CK处理稻田土的2.0倍和2.1倍,表明MGEs在施用粪肥及缓控释肥的水稻土中富集。在intI-1(clinic)、tnpA-3和tnpA-4等主要MGEs的丰度较高的情况下,可能导致ARGs的基因水平转移速度加快,进而促进了其在稻田土中的进一步传播、富集和演化。ARGs通过MGEs水平转移作为基因的重要转移方式应在施肥土壤环境中得到重视,并应加强农田土壤中ARGs的水平转移机制的研究。
|
|
表 1 耐药基因与可移动遗传元件的相关性 Table 1 Correlation analysis between ARGs and MGEs |
(1)不同施肥模式下,稻田土壤中ARGs具有不同的分布特征,其中ARGs绝对丰度呈现施用粪肥土壤>施用缓控释肥土壤>施用化肥土壤>不施肥对照土壤,但不同施肥处理模式下的稻田土中优势基因基本一致,多为多重耐药类、氨基糖苷类和四环素类ARGs。
(2)不同施肥模式下,稻田土壤中ARGs和MGEs的赋存多样性存在显著差异,其中施加粪肥稻田土壤中的ARGs的赋存多样性和MGEs的种类显著高于不施肥、施加化肥和施加缓控释肥的田块,不施肥模式则显著低于其他处理模式。
(3)不同深度土层中稻田土壤ARGs的相对丰度呈现向下富集的趋势,转移传播的速度可能受水稻生长条件、可移动遗传元件、施肥方式等自然环境和人为因素影响,其在不同土层中的传播机制仍需进一步探究。
(4)长期施肥模式下,不同土层土壤中的ARGs水平与MGEs丰度显著相关,其中intI-1(clinic)与丰度较高的多重耐药类和β-内酰胺类中的多种ARGs有极显著正相关性(P < 0.01),表明土壤中MGEs可能促进了ARGs的传播扩增,加剧了农田土壤的ARGs污染。
| [1] |
周启星, 罗义, 王美娥. 抗生素的环境残留、生态毒性及抗性基因污染[J]. 生态毒理学报, 2007, 2(3): 243-251. ZHOU Q X, LUO Y, WANG M E. Environmental residues and ecotoxicity of antibiotics and their resistance gene pollution: A review[J]. Asian Journal of Ecotoxicology, 2007, 2(3): 243-251. |
| [2] |
KNAPP C W, DOLFING J, EHLERT P A, et al. Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940[J]. Environmental Science & Technology, 2009, 44(2): 580-587. |
| [3] |
TIEN Y C, LI B, ZHANG T, et al. Impact of dairy manure pre-application treatment on manure composition, soil dynamics of antibiotic resistance genes, and abundance of antibiotic-resistance genes on vegetables at harvest[J]. Science of the Total Environment, 2017, 581/582: 32-39. DOI:10.1016/j.scitotenv.2016.12.138 |
| [4] |
CHEE-SANFORD J C, MACKIE R I, KOIKE S, et al. Fate and transport of antibiotic residues and antibiotic resistance genes following land application of manure waste[J]. Journal of Environmental Quality, 2009, 38(3): 1086-1108. DOI:10.2134/jeq2008.0128 |
| [5] |
张昊. 抗生素及其耐药性在畜禽粪便-土壤-蔬菜中的传播和转移[D]. 新乡: 河南师范大学, 2018. ZHANG H. Spread and transfer of antibiotics and antibiotic resistance in the livestock manure-soil-vegetable endophytic system[D]. Xinxiang: Henan Normal University, 2018. |
| [6] |
LIU W, LING N, GUO J, et al. Dynamics of the antibiotic resistome in agricultural soils amended with different sources of animal manures over three consecutive years[J]. Journal of Hazardous Materials, 2021, 401: 123399. DOI:10.1016/j.jhazmat.2020.123399 |
| [7] |
JONES-DIAS D, MANAGEIRO V, FERREIRA E, et al. Architecture of class 1, 2, and 3 integrons from gram negative bacteria recovered among fruits and vegetables[J]. Frontiers in Microbiology, 2016, 7: 1400. |
| [8] |
WANG F, XU M, STEDTFELD R D, et al. Long-term effect of different fertilization and cropping systems on the soil antibiotic resistome[J]. Environmental Science & Technology, 2018, 52(22): 13037-13046. |
| [9] |
左金龙, 孙宇琪, 郭雅杰, 等. 不同施肥处理对蔬菜土壤中抗生素抗性基因多样性与丰度的影响[J]. 环境污染与防治, 2021, 43(5): 553-556, 561. ZUO J L, SUN Y Q, GUO Y J, et al. Effects of different fertilization treatments on the diversity and abundance of antibiotic resistance genes in vegetable[J]. Environmental Pollution & Control, 2021, 43(5): 553-556, 561. |
| [10] |
YANG F, ZHANG K, ZHI S, et al. High prevalence and dissemination of β-lactamase genes in swine farms in northern China[J]. Science of the Total Environment, 2019, 651: 2507-2513. DOI:10.1016/j.scitotenv.2018.10.144 |
| [11] |
LOOFT T, JOHNSON T A, ALLEN H K, et al. In-feed antibiotic effects on the swine intestinal microbiome[J]. Proceedings of the National Academy of Sciences, 2012, 109(5): 1691-1696. DOI:10.1073/pnas.1120238109 |
| [12] |
GÓMEZ-GÓMEZ C, BLANCO-PICAZO P, BROWN-JAQUE M, et al. Infectious phage particles packaging antibiotic resistance genes found in meat products and chicken feces[J]. Scientific Reports, 2019, 9(1): 1-11. |
| [13] |
LIU Y Y, WANG Y, WALSH T R, et al. Emergence of plasmid-mediated colistin resistance mechanism MCR-1 in animals and human beings in China: A microbiological and molecular biological study[J]. The Lancet Infectious Diseases, 2016, 16(2): 161-168. DOI:10.1016/S1473-3099(15)00424-7 |
| [14] |
YANG F, GU Y, ZHOU J, et al. Swine waste: A reservoir of high-risk blaNDM and mcr-1[J]. Science of the Total Environment, 2019, 683: 308-316. DOI:10.1016/j.scitotenv.2019.05.251 |
| [15] |
YANG F, ZHANG K, ZHI S, et al. High prevalence and dissemination of β-lactamase genes in swine farms in northern China[J]. Science of the Total Environment, 2019, 651: 2507-2513. DOI:10.1016/j.scitotenv.2018.10.144 |
| [16] |
WANG F, HAN W, CHEN S, et al. Fifteen-year application of manure and chemical fertilizers differently impacts soil ARGs and microbial community structure[J]. Frontiers in Microbiology, 2020, 11: 62. DOI:10.3389/fmicb.2020.00062 |
| [17] |
XU Y, LI H, SHAO Z, et al. Fate of antibiotic resistance genes in farmland soil applied with three different fertilizers during the growth cycle of pakchoi and after harvesting[J]. Journal of Environmental Management, 2021, 289: 112576. DOI:10.1016/j.jenvman.2021.112576 |
| [18] |
PAN M, CHU L M. Occurrence of antibiotics and antibiotic resistance genes in soils from wastewater irrigation areas in the Pearl River Delta region, southern China[J]. Science of the Total Environment, 2018, 624: 145-152. DOI:10.1016/j.scitotenv.2017.12.008 |
| [19] |
MCKINNEY C W, DUNGAN R S, MOORE A, et al. Occurrence and abundance of antibiotic resistance genes in agricultural soil receiving dairy manure[J]. FEMS Microbiology Ecology, 2018, 94(3). |
| [20] |
彭双, 王一明, 林先贵. 连续施用发酵猪粪对土壤中四环素抗性基因数量的影响[J]. 中国环境科学, 2015, 35(4): 1173-1180. PENG S, WANG Y M, LIN X G. Abundance of the tetracycline resistance genes in a paddy soil after continuous application of composted swine manure for 6 years[J]. China Environmental Science, 2015, 35(4): 1173-1180. |
| [21] |
何燕, 朱冬, 王东, 等. 四川省稻田土壤的抗生素抗性基因多样性研究[J]. 农业环境科学学报, 2020, 39(6): 1249-1258. HE Y, ZHU D, WANG D, et al. Diversity of antibiotic resistance genes in paddy soils in Sichuan Province, China[J]. Journal of Agro-Environment Science, 2020, 39(6): 1249-1258. |
| [22] |
CHEN Q, AN X, LI H, et al. Long-term field application of sewage sludge increases the abundance of antibiotic resistance genes in soil[J]. Environment International, 2016, 92/93: 1-10. DOI:10.1016/j.envint.2016.03.026 |
| [23] |
张毓森, 叶军, 苏建强. 粪肥与铜一次性施用对农田土壤抗生素抗性基因的长期影响[J]. 应用与环境生物学报, 2019, 25(2): 328-332. ZHANG Y S, YE J, SU J Q. Long-term effect of one-time application of manure or copper antibiotic resistance genes in agricultural soil[J]. Chinese Journal of Applied and Environmental Bioligy, 2019, 25(2): 328-332. |
| [24] |
黄福义, 李虎, 韦蓓, 等. 长期施用猪粪水稻土抗生素抗性基因污染研究[J]. 环境科学, 2014, 35(10): 3869-3873. HUANG F Y, LI H, WEI B, et al. Long-term manure application induced shift of diversity and abundance of antibiotic resistance genes in paddy soil[J]. Environmental Science, 2014, 35(10): 3869-3873. |
| [25] |
ZHU Y G, JOHNSON T A, SU J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [26] |
ZHU Y G, ZHAO Y, ZHU D, et al. Soil biota, antimicrobial resistance and planetary health[J]. Environment International, 2019, 131: 105059. DOI:10.1016/j.envint.2019.105059 |
| [27] |
PARTRIDGE S R, TSAFNAT G, COIERA E, et al. Gene cassettes and cassette arrays in mobile resistance integrons[J]. FEMS Microbiology Reviews, 2009, 33(4): 757-784. DOI:10.1111/j.1574-6976.2009.00175.x |
 2021, Vol. 40
2021, Vol. 40