2. 沈阳大学区域污染环境生态修复教育部重点实验室, 沈阳 110044;
3. 东北农业大学水利与土木工程学院, 哈尔滨 150030;
4. 四川省地质矿产勘查开发局成都水文地质工程地质中心, 成都 610081
2. Key Lab of Eco-restoration of Regional Environment Pollution, Shenyang University, Shenyang 110044, China;
3. College of Water Conservancy and Civil Engineering, Northeast Agricultural University, Harbin 150030, China;
4. Chengdu Center of Hydrogeology and Engineering Geology, SBGEEMR, Chengdu 610081, China
草甘膦[N-(膦酸甲基)甘氨酸]是一种广谱的系统性除草剂,主要作用于杂草的韧皮部,对杂草中合成蛋白质所必需的5-烯醇丙酮酸莽草酸-3-磷酸合酶(简称为EPSP合酶)具有显著的抑制作用,并会在整个植物中转移,还会通过其磷酸基团吸附在土壤矿物上[1]。作为除草剂农达(Roundup)的主要成分,草甘膦因具有低成本、高效率除杂草的优点而在全世界农业领域得到广泛使用。美国地质勘探局(USGS)在2007年对采集于全国范围内的1 262个地表水样本和193个土壤样本进行了草甘膦残留量的检测,结果表明其中489个地表水样本中有草甘膦的检出,浓度范围为0.02~427.00 μg·kg-1,119个土壤样本中有草甘膦的检出,其浓度范围为1.0~476.0 μg·kg-1,同时在地下水样本和雨水样本中也分别有不同程度的草甘膦检出[2]。草甘膦在土壤和水环境中的积累,对人类健康以及水土环境产生诸多负面影响[3-6]。草甘膦会在作物的组织中积累,当其通过污染作物被人类食用并在体内积累到一定含量后即会危害健康,导致癌症、慢性肾病、先天性缺陷等[7-9]。2009年Poulsen等[10]最先提出草甘膦能够穿透人类胎盘障碍进入胎儿室,危害胎儿正常发育。2009年Senapati等[11]首次提出草甘膦会使人食道、胃和肠道中蛋白酶、脂肪酶和淀粉酶的活性降低,从而引发消化系统紊乱,出现腹泻、过敏等症状。2017年,国际癌症研究机构(IARC)首次将草甘膦列为人类“可能致癌”物质,随后世界卫生组织根据其结论,把草甘膦列为“2A级”致癌物质[12]。同时,草甘膦的积累给土壤和水环境带来各种危害[13-15]。2009年Valeska等[16]首次对比了分别暴露于草甘膦与其合成制剂农达中的夹杂带丝蚓,同时段内两种物质都使蠕虫体内抗氧化酶活性显著提高,结果表明草甘膦对于土壤生物存在氧化应激效应。去除草甘膦在水土环境中的残留污染已成为当下急需解决的问题。
土壤中的草甘膦降解方式主要有水解、光化学降解、生物降解等[17],其中生物降解是草甘膦降解的主要途径。自然界中微生物广泛存在,尤其在长期施用草甘膦的土壤中会存在耐受或利用草甘膦的微生物菌群[18]。因此,利用具有草甘膦降解能力的本土微生物进行生物修复是最适合的策略,且生物降解技术具有低成本、高效、无环境负面影响的优点[19-21]。近年来利用微生物降解草甘膦的研究得到了越来越多的关注。Moore等[22]于1983年首次筛选出利用草甘膦作为唯一磷源的假单胞菌Pseudomonas sp. PG2982,这为筛选草甘膦降解菌的研究奠定了基础。Fan等[23]首次分离出具有草甘膦降解能力的蜡样芽孢杆菌,该菌能利用草甘膦为唯一碳源生长。Hadi等[24]分离筛选出草甘膦降解菌Ochrobactrum sp. GDOS,这是首次报道的能在60 h内将507 mg·L-1草甘膦完全降解的细菌,其对于污染水土修复具有重要意义。Zhao等[25]在中国某除草剂生产厂附近污染土壤中分离得到3株具有草甘膦降解能力的假单胞菌属细菌,接种菌的污染土壤相较于未接种土壤的草甘膦去除率提高了2~3倍,同时利用一级反应动力学方程对草甘膦降解过程进行拟合,但一级反应动力学方程并不能反映微生物在生长代谢中受到抑制的过程。利用微生物降解草甘膦(细菌、真菌、放线菌等)时,细菌最有效,并起到最关键的作用,诸多已知的具备降解草甘膦能力的菌种都属于细菌,例如Pseudomonas sp.、Burkholderia sp.、Arthrobacter sp.、Ochrobactrum sp.等[26-30]。
在对降解菌的生物降解动力学研究中,草甘膦可以被认为是一种生长抑制化合物,Haldane方程是表征底物对微生物生长抑制的方程,适用于拟合草甘膦菌株的生长动力学[31]。Nourouzi等[32]利用Haldane方程拟合不同初始浓度、pH、初始接种量下草甘膦在溶液中的生物降解,结果显示当草甘膦浓度大于500 mg·L-1时,Haldane方程拟合的R2值较高(0.942),表明在较高草甘膦初始浓度下,Haldane模型能够表征底物的抑制作用。Yan等[33]在活性污泥中筛选得到草甘膦降解菌Agrobacterium tumefaciens BZ8,并对其降解过程进行了Haldane动力学方程拟合,得到菌株BZ8的动力学参数μmax为1.28 h-1、Ks为84.82 mg·L-1、Ksi为227.59 mg·L-1(R2=0.99),确定其菌株相应的降解动力学参数,同时证明Haldane方程与实验数据具有很强的相关性。
在以往对草甘膦微生物修复的研究报道中,利用剑菌降解草甘膦同时优化其最佳培养条件的研究尚未出现,并且目前还没有报道使用Haldane方程拟合剑菌菌株降解草甘膦的动力学参数。本研究通过分离筛选实验获取了一株具有草甘膦降解能力的剑菌菌株Ensifer sp. BRY,并利用16s rDNA序列分析手段对其进行了菌种鉴定。通过探究该细菌最佳降解条件(pH、草甘膦初始浓度、接种量、外加碳源)以获得最大的生物降解性能,并利用Haldane降解动力学模型首次拟合剑菌菌株对草甘膦的降解过程,确定其降解动力学参数,优化降解菌的培养条件,为剑菌菌株用于含草甘膦农药的生物降解提供数据参考。
1 材料与方法 1.1 试验材料98.2%草甘膦原药购买于百灵威科技有限公司,琼脂购于索莱宝生化试剂公司,其他化学试剂为国产分析纯。
以草甘膦为唯一碳源的基础盐培养基,每1 L蒸馏水中组分构成:MgSO4·7H2O 0.500 g、K2HPO4·3H2O 1.310 g、FeSO4 · 7H2O 0.018 g、NaNO3 3.000 g。外加200 mg·L-1草甘膦用于培养和分离草甘膦降解菌株。
主要仪器包括高压蒸汽灭菌锅(日本TOMY/SX-500),离子色谱仪(瑞士万通/930 Compact),紫外分光光度计(Hitachi/U-2910),恒温培养振荡器(上海智城/ZWYR-2102C),生化培养箱(上海智城/ZXSD-B1090),超净工作台(安泰/SW-CJ-2FD),Milli-Q超纯水系统(默克)。
1.2 草甘膦降解菌的富集与分离供试土壤取自辽宁省沈阳市沈北新区前进农场土壤(123°31′ 29″ ~123°31′ 55″ E,41°04′ 50″ ~42°05′ 10″ N),该区长期施用多种复合农药。土样200 g取自0~15 cm土层,自然阴干后储存备用。
取10 g土样和100 mL基础盐培养基于250 mL锥形瓶中,外加50 mg·L-1草甘膦,在30 ℃、150 r·min-1的条件下振荡培养3 d,取10 mL上层悬浮液加入到100 mL新鲜基础盐培养基中,外加100 mg·L-1草甘膦,在同样条件下继续培养,第3个周期以后固定外加200 mg·L-1草甘膦,并在相同条件下培养。自第4个周期起,在液体培养基中蘸取适量培养液涂布于基础盐固体培养基和牛肉膏蛋白胨固体培养基平板上,其中基础盐培养基以草甘膦为唯一碳源,在30 ℃恒温培养箱中培养3 d,待菌落长出后,用接种环在分离较好的单一菌落上沾取少量菌体涂抹于以草甘膦为唯一碳源的基础盐培养基平板上,倒置于30 ℃恒温培养箱中培养,如此反复,经过13个周期,直到平板上长出的所有菌落形状和大小相近。
1.3 细菌的鉴定和系统发育分析利用高通量测序对筛选得到的菌株进行菌种分类学鉴定,通过PCR通用引物对菌株的16S rDNA进行基因序列扩增,正向引物为27F:5′-AGAGTTTGATCCTGGCTCAG-3′,反向引物为1492R:5′-AAGGAGGTGATCCAGCCGCA-3′,扩增反应体积50 μL,反应条件为94 ℃预变性3 min,94 ℃变性1 min,55 ℃退火1 min,72 ℃延伸2 min,共进行35个循环。产物经1% 琼脂糖凝胶电泳,经染色剂染色后凝胶成像,将产物于-80 ℃保存。在The EzBioCloud database(www.ezbiocloud.net)对BRY进行序列比对分析,将所得结果利用MEGA10.0软件中的Neighbor-Joining(NJ)方法构建系统发育树。
1.4 菌株BRY的降解特性挑取适量BRY菌落接种于200 mg·L-1草甘膦基础盐培养基中,在30 ℃、150 r·min-1条件下培养至对数生长期后离心(10 000 r·min-1,10 min),去上清,使用磷酸盐缓冲溶液对沉淀菌体进行反复冲洗两次后重悬,调节菌悬液OD600值为0.2,采用稀释涂布平板法测得此时菌液细胞浓度约为1.4×107 cfu·mL-1。通过每次改变一种培养条件,来探究菌株BRY的生长特性。
以10% 的接种量接种BRY至不同pH(5.0、6.0、7.0、8.0、9.0,pH调节使用1 mol·L-1 HCl溶液和1 mol· L-1 NaOH溶液)的基础盐培养基中,并设置草甘膦浓度为100 mg·L-1,放置于30 ℃、150 r·min-1的摇床中,自0 h起,每隔4 h取8 mL样品,经0.22 μm滤膜过滤后,使用离子色谱法[34]测定草甘膦浓度,仪器参数设置:淋洗液为8 mmol · L-1的Na2CO3;抑制系统为0.5%H2SO4+超纯水;流速为0.7 mL·min-1;进样体积为20 μL;柱温308 K;泵压9.9 MPa;色谱运行时间31.0min;保留时间25.0~26.0 min,该方法检测范围为0.1~ 100.0 mg·L-1。同时利用紫外分光光度计测定菌液OD600值,以考察菌株在不同pH条件下的生长情况。草甘膦降解率计算公式:
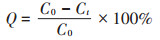
|
式中:Q为草甘膦降解率,%;C0为草甘膦的初始浓度,mg·L-1;Ct为培养结束后培养基中残留的草甘膦浓度,mg·L-1。
为研究BRY对污染物草甘膦的耐受能力,以10% 的接种量接种BRY至不同浓度(50、100、200、300、400 mg·L-1)草甘膦基础盐培养基中,放置于30 ℃、150 r· min-1的摇床中,自0 h起,每隔4 h取8 mL样品,经0.22 μm滤膜过滤后,利用离子色谱测定草甘膦浓度,同时利用紫外分光光度计测定菌液OD600值。
为研究接种量对降解速率的影响,以1%、5%、10%、20%、30%的接种量接种BRY至200 mg·L-1草甘膦基础盐培养基中,放置于30 ℃、150 r·min-1的摇床中,自0 h起,每隔4 h取8 mL样品,经0.22 μm滤膜过滤后,利用离子色谱测定草甘膦浓度,同时利用紫外分光光度计测定菌液OD600值。
为研究BRY在有其他碳源的影响下对草甘膦的利用能力,选取单糖(葡萄糖0.5 g·L-1)和双糖(蔗糖0.5 g·L-1)加入到200 mg·L-1草甘膦基础盐培养基中,另设不外加碳源的草甘膦基础盐培养基作为对照。其中BRY接种量为10%,在30 ℃、150 r·min-1的摇床中培养,自0 h起,每隔4 h取8 mL样品,经0.22 μm滤膜过滤后,测定溶液pH值,利用离子色谱测定草甘膦浓度,同时利用紫外分光光度计测定菌液OD600值。
1.5 菌株对草甘膦的降解动力学实验Monod动力学模型是最常用来描述微生物生长的动力学模型,但当体系含有抑制性底物时Monod模型并不适用。Haldane模型在Monod模型基础上开发,目前已经被广泛用于表征底物对微生物的生长抑制[35]。因此本研究采用Haldane方程来描述菌株BRY的生长动力学。对于不同初始浓度草甘膦的反应体系,菌株比生长速率用如下公式计算:
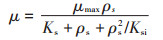
|
式中:μ为菌体的比生长速率,h-1;μmax为最大比细胞生长速率,h-1;ρs为草甘膦初始浓度,mg·L-1;Ks为半饱和常数,mg·L-1;Ksi为抑制常数,mg·L-1。
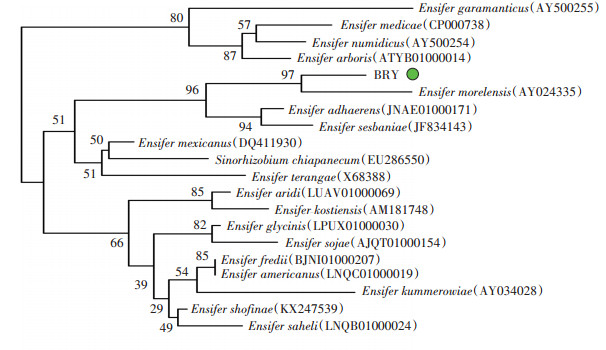
2 结果与讨论 2.1 菌株BRY的分子生物学鉴定以BRY的总DNA为模板,PCR扩增16s rDNA序列,16S rDNA长度为1 363 bp。将BRY的16S rDNA扩增序列在The EzBioCloud database上做序列比对分析,结果表明其与Ensifer morelensis模式菌株Lc04(GenBank登录号AY024335)的16S rDNA基因序列相似性达到99.48%。根据BRY的16S rDNA序列与其相关种属16S rDNA序列构建系统进化发育树(图 1)可知,菌株BRY与Ensifer morelensis进化关系最近,并与多株Ensifer sp.型菌株聚在一个支持良好的分支上,因此确定菌株BRY归属于剑菌属(Ensifer sp.)。模式菌株Ensifer morelensis Lc04最早由Wang等[36]在2002年提出,该菌株为革兰氏阴性菌,无芽孢,杆状,好氧,能以广泛的碳水化合物和氨基酸作为唯一碳源生长,并且除dl-丙氨酸和尿素外,大多数氨基酸可以作为其生长的唯一氮源,同时对羧苄青霉素、卡那霉素、红霉素等抗生素具有多重耐药性。
|
图 1 草甘膦降解菌株BRY基于16S rDNA的系统发育树 Figure 1 Neighbour-joining tree based on 16S rRNA sequences for isolat strain BRY |
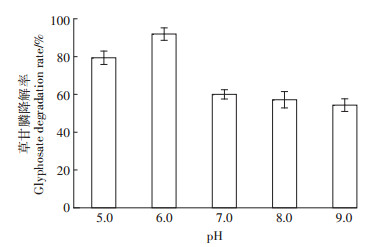
微生物生长体系中pH的变化会通过影响细胞膜所带电荷而改变细胞对外界物质的吸收,偏酸性或偏碱性的环境条件均会影响生物酶的活性,主要表现在细胞膜电荷的变化以及蛋白质、核酸等生物大分子所带电荷的变化[37]。细菌的生长状况会直接影响对污染物的降解率[38]。pH对菌株BRY降解草甘膦能力的影响如图 2所示,可以看出pH对菌株BRY的降解率影响较大,在50 h培养时间内,菌株BRY在pH 5.0时的草甘膦降解率为79.36%,在pH 6.0时达到最高降解率91.93%,在pH为7.0、8.0、9.0时的降解率逐渐下降,分别为60.02%、57.18%、54.32%。草甘膦属于酸性除草剂,长期过量使用会导致土壤酸化,影响土壤中的生物转化[39]。一些草甘膦降解菌从长期施用草甘膦的农田土壤中分离筛选出来后,都在偏酸性的条件下展现出更强的降解能力。Fan等[23]对降解菌Bacillus cereus CB4降解草甘膦的pH条件的研究结果表明,在pH 6.0时草甘膦降解率最高,并且随着pH的增大降解率逐渐下降。张慧芳等[40]分离筛选得到的菌株Burkholderia vietnamiensis B-1和假单胞菌属Pseudomonas sp. Y-1均在pH 6.0条件下达到最佳降解能力,认为弱酸性条件下降解酶活性较高。Sun等[41]对固氮细菌Ensifer meliloti及其腈水合酶(NHase)降解新烟碱类杀虫剂的特性研究中表明,NHase在pH 6.0~9.0条件下保存12 h后,活性仍保留在93% 以上,其中在pH 6.0条件下,NHase活性保持最高。因此推测pH 6.0更有助于提高菌株BRY的草甘膦降解酶的活性。总体而言,Ensifer sp. BRY在pH 6.0时的降解效果最佳。
|
图 2 不同pH条件下菌株BRY的草甘膦降解率 Figure 2 Degradation rate of glyphosate by strain BRY with different pH |
当微生物培养体系中草甘膦浓度发生变化时,细菌的生长状况也会受到影响,草甘膦的生物毒性随着其浓度的增大而增大,研究细菌对不同浓度草甘膦的降解程度是评价其降解能力的主要标准之一[42]。菌株BRY对不同浓度草甘膦的降解能力见图 3。总体上,同时间内菌株的生长量随着草甘膦浓度的增大而有所减少,当草甘膦初始浓度为100、200、300 mg·L-1时,同时间内菌株OD600值相差较小,说明在这一浓度范围内草甘膦对菌株BRY的生长影响较小。50 h内,在草甘膦浓度为50、100、200、300、400 mg·L-1时的降解率分别为53.39%、60.64%、62.52%、69.60%、40.01%,表明Ensifer sp. BRY能够在以草甘膦为唯一碳源的培养基中生长,可以耐受最高400 mg·L-1的草甘膦,并在此浓度下具有一定降解能力。在不同初始浓度草甘膦溶液中,同时间内菌株BRY在300 mg·L-1草甘膦培养基中降解率最高。降解过程在50 h左右趋于平稳,因此在实际场地修复过程中,菌株BRY应至少在土壤中停留50 h以上才能达到较佳修复效果。

|
图 3 不同初始草甘膦浓度下菌株BRY的草甘膦降解情况 Figure 3 Degradation of glyphosate by strain BRY under different initial glyphosate concentrations |
初始接种量是直接影响草甘膦降解菌降解的影响因素,一定程度上增加细菌初始接种量可以提高降解速率、减少生长延迟时间[43]。研究不同初始接种量对菌株BRY降解草甘膦速率的影响,以此确定菌株BRY对草甘膦的最佳降解条件。不同初始接种量对菌株BRY降解草甘膦能力的影响见图 4。24 h内,在初始接种量为1%、5%、10%、20% 和30% 时的草甘膦降解率分别为7.52%、15.57%、14.89%、40.06% 和26.80%。总体上,从经济成本以及修复效率考虑,20% 接种量是草甘膦降解的最佳接种量,在8 h内草甘膦降解菌的降解率可达到40.06%,并在72 h达到完全降解。

|
图 4 不同初始接种量时菌株BRY的草甘膦降解率 Figure 4 Degradation rate of glyphosate by strain BRY at different initial inoculum |
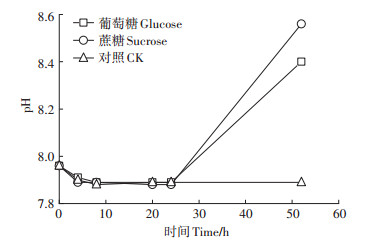
蔗糖和葡萄糖均属于容易被微生物利用的外加碳源,葡萄糖属单糖,蔗糖是由果糖和葡萄糖构成的双糖。在微生物降解过程中,如培养体系中含有多种碳源时,会优先消耗葡萄糖和蔗糖这类速效碳源,因此参与利用的其他碳源的酶系合成会受到抑制,这一现象称为碳源阻遏效应[44]。因此分别用葡萄糖和果糖作为外加碳源加入培养体系中,以探究外加碳源对菌株BRY降解草甘膦能力的影响,其结果如图 5所示。由结果可知,外加葡萄糖和蔗糖都明显促进了菌株BRY的生长,在30 ℃、起始pH 7.96、150 r·min-1时,对照中草甘膦降解率较高,加入葡萄糖和果糖都会使草甘膦降解率下降,因为在碳源阻遏效应的影响下,菌株BRY会优先代谢消耗葡萄糖和蔗糖。在培养过程中实时监测溶液pH变化,结果如图 6,显示外加碳源的两种体系pH均升高,由于降解过程中pH的升高间接导致了草甘膦降解率的降低。

|
图 5 外加碳源(葡萄糖和蔗糖)对BRY草甘膦降解率的影响 Figure 5 The effect of adding carbon sources(glucose and sucrose)on the degradation of BRY glyphosate |
|
图 6 外加葡萄糖、蔗糖以及对照体系中pH的变化 Figure 6 pH changes in systems with added glucose, sucrose and no treatment |
微生物在不同浓度污染物质中的比生长速率(μ)由指数生长期决定,是根据指数生长期的菌体细胞干质量和时间的半对数图绘制线性最小二乘拟合得到,在指数生长期可以认为比生长速率为一个常数[45]。菌体比生长速率的实验数据与Haldane模型拟合结果如图 7所示。结果表明,初始浓度由0 mg·L-1增加到100 mg·L-1时,菌体的比生长速率随着初始草甘膦浓度的增加而增大;初始浓度由100 mg·L-1增加到400 mg·L-1时,比生长速率随草甘膦浓度的增加而显著下降,呈现出由于底物浓度增加导致的菌体生长受到抑制现象。通过实验数据确定Haldane方程参数,并且利用Origin 2018软件进行非线性曲线拟合,得到以草甘膦为唯一碳源的培养基培养的菌株BRY的Haldane参数,μmax为1.68 h-1,Ks为167.80 mg·L-1,Ksi为50.55 mg·L-1,Ksi/Ks=0.30,相关系数R2=0.99,拟合情况良好。Ksi为50.55 mg·L-1,说明该菌株的草甘膦抑制浓度为50.55 mg·L-1,并且用Haldane方程描述菌株BRY降解过程是合理的。通过表 1中生长动力学拟合结果对比可以看出BRY具有较高的降解率和μmax值,说明将Ensifer sp. BRY应用于实际修复草甘膦污染具有较高的可行性和使用前景。

|
图 7 菌株比生长速率的实验数据与Haldane模型拟合结果 Figure 7 Experimental and predicted specific growth rates of the culture due to Haldane′s model |
|
|
表 1 不同草甘膦降解菌生长动力学参数对比 Table 1 Comparative list of glyphosate-degrading bacteria growth due to Haldane ′s model |
(1)通过富集培养法在辽宁省沈阳市沈北新区某农田土壤中分离得到一株草甘膦高效降解菌株Ensifer sp. BRY,其能够在以草甘膦为唯一碳源的培养基中生长。
(2)微生物修复的研究结果表明,草甘膦降解菌Ensifer sp. BRY对草甘膦的耐受能力最高达400 mg· L-1,其对草甘膦的去除率与初始草甘膦浓度有较大关系。30 ℃、pH 6.0、150 r·min-1、初始接种量为20%为最佳降解条件,在体系中外加碳源(葡萄糖、蔗糖,0.5 g·L-1)会促进Ensifer sp. BRY的生长,但会降低其对草甘膦的利用率。Ensifer sp. BRY在含100 mg·L-1草甘膦的基础盐培养基中50 h内降解率为91.93%,降解过程较短,在一定程度上提高了修复效率。
(3)Ensifer sp. BRY降解过程符合Haldane方程,μmax为1.68 h-1,Ks为167.80 mg·L-1,Ksi为50.55 mg·L-1,由拟合结果可知当浓度高于50.55 mg·L-1时降解速率开始下降,出现抑制模式。
| [1] |
Franz J E. Glyphosate: A Unique Global Herbicide[M]. Sikorski J A, Mao M K. Washington: American Chemical Society, 1997: 28.
|
| [2] |
Scribner E A, Battaglin W A, Gilliom R J, et al. Concentrations of glyphosate, its degradation product, aminomethylphosphonic acid, and glufosinate in ground - and surface-Water, rainfall, and soil samples collected in the United States, 2001-06[R]. U. S. Geological Survey Scientific Investigations Report, 2007.
|
| [3] |
Battaglin W A, Meyer M T, Kuivila K M, et al. Glyphosate and its degradation product AMPA occur frequently and widely in U. S. soils, surface water, groundwater, and precipitation[J]. Journal of the American Water Resources Association, 2014, 50(2): 275-290. DOI:10.1111/jawr.12159 |
| [4] |
Borggaard O K, Gimsing A L. Fate of glyphosate in soil and the possibility of leaching to ground and surface waters: A review[J]. Pest Management Science, 2010, 64(4): 441-456. |
| [5] |
Muskus A M, Krauss M, Miltner A, et al. Degradation of glyphosate in a colombian soil is influenced by temperature, total organic carbon content and pH[J]. Environmental Pollution, 2020, 259: 113767. DOI:10.1016/j.envpol.2019.113767 |
| [6] |
Okada E, Allinson M, Barral M P, et al. Glyphosate and aminomethylphosphonic acid (AMPA) are commonly found in urban streams and wetlands of Melbourne, Australia[J]. Water Research, 2020, 168: 115-139. |
| [7] |
Romano R, Souza P, Nunes M, et al. Perinatal exposure to a commercial formulation of glyphosate reduces the mRNA expression and increases the protein content of Beta TSH in the pituitary of male offspring[J]. Endocrine Abstracts, 2012, 29: 753. |
| [8] |
Marc J, Mulner-lorillon O, Bellé R. Glyphosate-based pesticides affect cell cycle regulation[J]. Biology of the Cell, 2004, 96(3): 245-249. DOI:10.1016/j.biolcel.2003.11.010 |
| [9] |
Jayasumana C, Gunatilake S, Senanayake P. Glyphosate, hard water and nephrotoxic metals: Are they the culprits behind the epidemic of chronic kidney disease of unknown etiology in Sri Lanka?[J]. Int J Environ Res Public Health, 2014, 11(2): 2125-2147. DOI:10.3390/ijerph110202125 |
| [10] |
Poulsen M S, Rytting E, Mose T, et al. Modeling placental transport: Correlation of in vitro BeWo cell permeability and ex vivo human placental perfusion[J]. Toxicology in Vitro, 2009, 23(7): 1380-1386. DOI:10.1016/j.tiv.2009.07.028 |
| [11] |
Senapati T, Mukerjee A, Ghosh A. Observations on the effect of glyphosate based herbicide on ultra structure(SEM)and enzymatic activity in different regions of alimentary canal and gill of Channa punctatus (Bloch)[J]. Journal of Crop and Weed, 2009, 5: 236-245. |
| [12] |
Peillex C, Pelletier M. The impact and toxicity of glyphosate and glyphosate-based herbicides on health and immunity[J]. Journal of Immunotoxicology, 2020, 17(1): 163-174. DOI:10.1080/1547691X.2020.1804492 |
| [13] |
Eberbach P. Applying non-steady-state compartmental analysis to investigate the simultaneous degradation of soluble and sorbed glyphosate(N-(phosphonomethyl) glycine)in four soils[J]. Pesticide Science, 1998, 52(3): 229-240. DOI:10.1002/(SICI)1096-9063(199803)52:3<229::AID-PS684>3.0.CO;2-O |
| [14] |
Kogevinas M. Probable carcinogenicity of glyphosate[J]. BMJ, 2019, 365: 13-16. |
| [15] |
Jensen P K, Wujcik C E, Mcguire M K, et al. Validation of reliable and selective methods for direct determination of glyphosate and aminomethylphosphonic acid in milk and urine using LC-MS/MS[J]. Journal of Environmental Science and Health, Part B, 2016, 51(4): 254-259. DOI:10.1080/03601234.2015.1120619 |
| [16] |
Valeska C, Eva K, Claudia W. Bioaccumulation of glyphosate and its formulation roundup Ultra in Lumbriculus variegatus and its effects on biotransformation and antioxidant enzymes[J]. Environmental Pollution, 2009, 157(1): 57-63. DOI:10.1016/j.envpol.2008.07.027 |
| [17] |
Lafi W K, Al-qodah Z. Combined advanced oxidation and biological treatment processes for the removal of pesticides from aqueous solutions[J]. Journal of Hazardous Materials, 2006, 137(1): 489-497. DOI:10.1016/j.jhazmat.2006.02.027 |
| [18] |
Mohsen N M, Chuah T G, Choong T S Y, et al. Glyphosate utilization as the source of carbon: Isolation and identification of new bacteria[J]. Journal of Chemistry, 2011, 8(4): 1582-1587. |
| [19] |
Khayat M E, Rahman M F A, Shukor M S, et al. Characterization of a molybdenum-reducing Bacillus sp. strain shayat with the ability to grow on SDS and diesel[J]. Rendiconti Linc, 2016, 27(3): 1-10. DOI:10.1007/s12210-016-0519-5 |
| [20] |
Yaacob N S, Mohamad R, Ahmad S A, et al. The influence of different modes of bioreactor operation on the efficiency of phenol degradation by Rhodococcus UKMP-5M[J]. Rendiconti Lincei, 2016, 27(4): 749-760. DOI:10.1007/s12210-016-0567-x |
| [21] |
Stefano F, Stefano A, Barbara C, et al. Arsenic removal from naturally contaminated waters: A review of methods combining chemical and biological treatments[J]. Rendiconti Lincei, 2016, 27(1): 51-58. DOI:10.1007/s12210-015-0461-y |
| [22] |
Moore J K, Braymer H D, Larson A D. Isolation of a Pseudomonas sp. which utilizes the phosphonate herbicide glyphosate[J]. Applied & Environmental Microbiology, 1983, 46(2): 316-320. |
| [23] |
Fan J Y, Yang G X, Zhao H Y, et al. Isolation, identification and characterization of a glyphosate-degrading bacterium, Bacillus cereus CB4, from soil[J]. The Journal of General and Applied Microbiology, 2012, 58(4): 263-271. DOI:10.2323/jgam.58.263 |
| [24] |
Hadi F, Mousavi A, Noghabi K A, et al. New bacterial strain of the genus Ochrobactrum with glyphosate-degrading activity[J]. Journal of Environmental Science and Health, 2013, 48(3): 208-213. DOI:10.1080/03601234.2013.730319 |
| [25] |
Zhao H Y, Tao K, Zhu J Y. Bioremediation potential of glyphosate-degrading Pseudomonas spp. strains isolated from contaminated soil[J]. The Journal of General and Applied Microbiology, 2015, 61(5): 165-170. DOI:10.2323/jgam.61.165 |
| [26] |
Zboinska E, Barbara L, Pawel K. Organophosphonate utilization by the wild-type strain of Pseudomonas fluorescens[J]. Applied and Environmental Microbiology, 1992, 58(9): 2993-2999. DOI:10.1128/AEM.58.9.2993-2999.1992 |
| [27] |
Jacob G S, Garbow J R, Hallas L E, et al. Metabolism of glyphosate in Pseudomonas sp. strain LBr[J]. Applied and Environmental Microbiology, 1988, 54(12): 2953-2958. DOI:10.1128/AEM.54.12.2953-2958.1988 |
| [28] |
李冠喜, 吴小芹, 叶建仁. 多噬伯克霍尔德氏菌WS-FJ9对草甘膦的降解特性[J]. 生态学报, 2013, 33(21): 6885-6894. LI Guan-xi, WU Xiao-qin, YE Jian-ren. The characterization of glyphosate degradation by Burkholderia multivorans WS-FJ9[J]. Acta Ecologica Sinica, 2013, 33(21): 6885-6894. |
| [29] |
Ermakova I T, Kiseleva N I, Shushkova T, et al. Bioremediation of glyphosate-contaminated soils[J]. Applied Microbiology and Biotechnology, 2010, 88(2): 585-594. DOI:10.1007/s00253-010-2775-0 |
| [30] |
Firdous S, Iqbal S, Anwar S, et al. Identification and analysis of 5-enolpyruvylshikimate-3-phosphate synthase (EPSPS) gene from glyphosate-resistant Ochrobactrum intermedium Sq20[J]. Pest management science, 2018, 74(5): 1184-1196. DOI:10.1002/ps.4624 |
| [31] |
Salihu I, Khalilah A K, Khadijah N M Z, et al. Biosurfactant production and growth kinetics studies of the waste canola oil-degrading bacterium Rhodococcus erythropolis AQ5-07 from Antarctica[J]. Molecules (Basel, Switzerland), 2020, 25(17): 38-78. |
| [32] |
Nourouzi M M, Chuah T G, Choong T S Y, et al. Modeling biodegradation and kinetics of glyphosate by artificial neural network[J]. Journal of Environmental Science & Health, 2012, 47(5): 455-465. |
| [33] |
Yan S B, Zhao W, Shi C E, et al. Isolation and characterization of a high salt-tolerant and glyphosate-degrading strain of Agrobacterium tumefaciens BZ8[J]. Brazilian Journal of Chemical Engineering, 2017, 34(3): 739-746. DOI:10.1590/0104-6632.20170343s20150803 |
| [34] |
李亚娟. 铁基石墨烯修复草甘膦污染水体的性能机理[D]. 沈阳: 沈阳大学, 2018: 20-21. LI Ya-juan. Performance and mechanism of iron -based graphene for remediation of glyphosate contaminated water[D]. Shenyang: Shenyang University, 2018: 20-21. |
| [35] |
Li Y, Li J, Wang C, et al. Growth kinetics and phenol biodegradation of psychrotrophic Pseudomonas putida LY1[J]. Bioresource Technology, 2010, 101(17): 6740-6744. DOI:10.1016/j.biortech.2010.03.083 |
| [36] |
Wang E T, Tan Z Y, Willems A, et al. Sinorhizobium morelense sp. nov., a Leucaena leucocephala-associated bacterium that is highly resistant to multiple antibiotics[J]. International Journal of Systematic and Evolutionary Microbiology, 2002, 52: 1687-1693. |
| [37] |
Singh B K, Walker A. Microbial degradation of organophosphorus compounds[J]. Fems Microbiology Reviews, 2010, 30(3): 428-471. |
| [38] |
Chodak M, Gołębiewski M, Morawska-płoskonka J, et al. Diversity of microorganisms from forest soils differently polluted with heavy metals[J]. Applied Soil Ecology, 2013, 64: 7-14. DOI:10.1016/j.apsoil.2012.11.004 |
| [39] |
李婷, 寇向龙, 李金娟, 等. 草甘膦污染现状及检测技术研究进展[J]. 江西农业, 2019, 22: 27-29. LI Ting, KOU Xiang-long, LI Jin-juan, et al. Current status of glyphosate pollution and research progress in detection technoloy[J]. Jiangxi Agriculture, 2019, 22: 27-29. DOI:10.3969/j.issn.1674-1479.2019.02.024 |
| [40] |
张慧芳, 冉梦兰, 汪倩, 等. 草甘膦微生物降解菌株的筛选及其生物学特性[J]. 贵州农业科学, 2015, 10: 111-115, 118. ZHANG Hui-fang, RAN Meng-lan, WANG Qian, et al. Screening and biological characteristics of strains with degrading glyphosate capacity[J]. Guizhou Agricultural Sciences, 2015, 10: 111-115, 118. |
| [41] |
Sun S L, Lu T Q, Yang W L, et al. Characterization of a versatile nitrile hydratase of the neonicotinoid thiacloprid-degrading bacterium Ensifer meliloti CGMCC 7333[J]. RSC Advances, 2016, 6(19): 15501-15508. DOI:10.1039/C5RA27966F |
| [42] |
Xu B, Sun Q J, Lan J C, et al. Exploring the glyphosate-degrading characteristics of a newly isolated, highly adapted indigenous bacterial strain, Providencia rettgeri GDB 1[J]. Journal of Bioscience and Bioengineering, 2019, 128(1): 80-87. DOI:10.1016/j.jbiosc.2019.01.012 |
| [43] |
Ishii S, Suzuki S, Yamanaka Y, et al. Population dynamics of electrogenic microbial communities in microbial fuel cells started with three different inoculum sources[J]. Bioelectrochemistry, 2017, 117: 74-82. DOI:10.1016/j.bioelechem.2017.06.003 |
| [44] |
冷一非. 微生物降解四环素特性及降解机理研究[D]. 武汉: 中国地质大学, 2017: 31-33. LENG Yi-fei. Characteristics and mechanism of tetracycline degradation by microorganism[D]. Wuhan: China University of Geosciences, 2017: 31-33. |
| [45] |
温玉娟. 再生水中硝基芳香族有机物污染的SAT及其生物强化修复机理研究[D]. 长春: 吉林大学, 2016: 62-64. WEN Yu-juan. Removal mechanisims of para - nitrophenol in reclaimed water using SAT and its bio-enhancement[D]. Changchun: Jilin University, 2016: 62-64. |
| [46] |
Dan F, Laure M, Guillaume C, et al. Biodegradation capabilities of acclimated activated sludge towards glyphosate: Experimental study and kinetic modeling[J]. Biochemical Engineering Journal, 2020, 161: 107643. DOI:10.1016/j.bej.2020.107643 |
| [47] |
Tazdait D, Salah R, Grib H, et al. Kinetic study on biodegradation of glyphosate with unacclimated activated sludge[J]. International Journal of Environmental Health Research, 2018, 28(4): 448-459. DOI:10.1080/09603123.2018.1487043 |
 2021, Vol. 40
2021, Vol. 40



