大气氮沉降增加呈现全球化趋势,我国是继欧洲、北美之后的第3大氮沉降区,大气氮沉降远高于全球平均水平[1]。大气氮沉降借助其对土壤碳固定[2]、植物氮素利用等的直接或间接作用[3],极大地干预了生态系统碳蓄积和氮素重新分配过程[4-5]。高氮沉降导致土壤氮转化过程发生变化[6]、土壤可利用性氮含量增加[7]、氮循环微生物数量及组成发生变化[8],进而对生态系统产生影响。
土壤氮转化主要过程包括生物固氮作用、氨化作用、硝化作用和反硝化作用,这些过程都由土壤微生物所驱动。氮沉降进入土壤后的一系列转化离不开与氮素转化的相关微生物参与。近年来的研究表明,施氮肥显著影响固氮菌、硝化菌和反硝化菌群落结构和丰度[8]。适量氮肥添加提高固氮菌丰度,促进土壤微生物的固氮功能[9];高浓度无机氮添加抑制固氮菌生长[10],从而抑制固氮微生物生长。Ning等[11]研究发现氮相关功能基因对氮添加速率表现出不同的敏感性,氨氧化细菌AOB-amoA基因比氨氧化古菌AOAamoA基因对氮添加更加敏感。土壤氨氧化细菌丰度随氮添加量增加而增加,而氨氧化古菌丰度则无明显变化[12]。氮肥种类和氮肥施用量会影响土壤氨氧化微生物组成和丰度[13]。受氮素限制的黄土高原盐碱草地,施氮后显著地提高了微生物生物量氮转化速率[14]。在亚热带森林、温带森林中研究发现,N2O排放、CH4吸收与AOA、AOB群落丰度分别呈现正、负相关关系,氨氧化菌群落动态能够解释土壤CH4吸收和N2O排放之间的消长作用[15]。
目前,在氮沉降增加的背景下,针对干旱和半干旱草原土壤氮转化功能基因丰度对氮沉降增加的响应特征还不够明确。为此,本研究通过施氮模拟氮沉降增加,研究不同氮添加水平对涉及土壤氮循环中的固氮nifH基因、氨氧化细菌AOB-amoA 基因、氨氧化古菌AOA-amoA基因和反硝化基因nirK丰度的影响,通过分析氮转化功能基因丰度与土壤理化因子之间的相关性关系,研究氮添加对微生物介导的固氮、氨氧化和反硝化过程的影响及其反馈,为深入认识草地生态系统氮循环及其对氮沉降增加的响应机制提供参考。
1 材料与方法 1.1 研究区域概况与样地设置研究区域概况、试验样地设置及试验前植被类型和基本理化性质详见参考文献[16]。试验样地位于内蒙古贝加尔针茅草原(48°27′ ~48°35′ N,119°35′ ~ 119°41′E)。于2010年开始进行围栏试验,氮添加水平设置参考国际上同类研究试验[17-18]。氮添加处理设8个水平,依次为:0、15、30、50、100、150、200、300 kg N·hm-2·a-1,分别记为:对照N0,低氮(N15、N30和N50),高氮(N100、N150、N200和N300)。
氮素为NH4NO3,每年6月中旬和7月中旬进行氮添加。共设置8个处理水平,4次重复。小区面积8 m×8 m,小区间设置2 m隔离带,重复间设置5 m隔离带。
1.2 样品采集与处理土壤样品采集于2015年8月中旬,用土钻在各小区采集0~10 cm土壤样品,每小区采集10个点,混合均匀。去除土壤入侵物和根系,将土壤样品分为两份,一份保存于冰盒中带回实验室,存于-20 ℃冰柜中;一份土壤样品于室内自然风干。
1.3 测定方法土壤理化性质测定:土壤pH采用玻璃电极法(土水比1:2.5),土壤总有机碳测定采用重铬酸钾外加热法,土壤全氮用凯氏定氮法,土壤铵态氮和硝态氮含量采用氯化钾溶液提取-流动分析仪(QC8000)测定[19]。土壤微生物生物量氮采用氯仿熏蒸-K2SO4提取-TOC仪(Multi N/C 3000)测定[20]。土壤可溶性氮以测定土壤微生物生物量氮时未熏蒸土壤的全氮含量表示,土壤可溶性有机氮以土壤可溶性氮减去铵态氮和硝态氮的差值表示[21]。
土壤DNA提取:称取0.25 g在冰柜中保存的土壤样品,用PowerSoil® DNA Isolationkit试剂盒,按照说明书提取土壤总DNA。用Nanodrop2000超微量分光光度计和1%琼脂糖电泳检测DNA质量。将通过质量检测的DNA样品于-20 ℃冰箱保存待用。
标准品配制:用目的基因引物进行PCR扩增。PCR反应体系为50 μL,包括上下游引物各1 μL,模板1 μL,2×Taq MasterMix 25 μL,灭菌超纯水22 μL。扩增条件为94 ℃ 5 min预变性后;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 30 s,共30个循环;最后1轮循环完成后再72 ℃延伸10 min。反应完毕后,用1%琼脂糖凝胶电泳检查扩增结果,目的片段进行琼脂糖凝胶回收。将回收PCR产物连接至pMD18-T载体,转化至大肠杆菌DH5α感受态细胞中,经Amp+、IPTG和X-gal的LB平板筛选阳性克隆,送去生工生物有限公司进行序列测定分析。
标准曲线制作:通过做预实验,将4种提取测序正确的质粒标准品从101~105倍进行10倍稀释,每个梯度取2 μL做模板建立标准曲线。
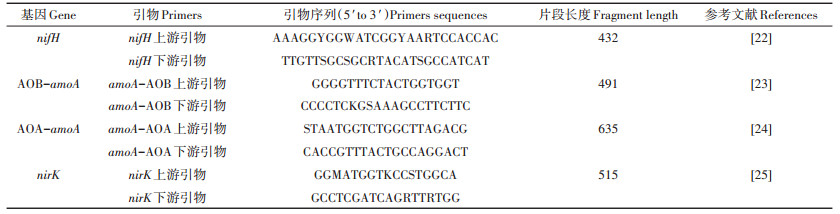
定量PCR检测:土壤固氮基因nifH、氨氧化细菌AOB、氨氧化古菌AOA和反硝化细菌定量扩增引物见表 1。扩增反应体系总体积为20 μL,包括2×GoTaq®qPCR Master Mix 10 μL,10 μmol·L-1的上下游引物各0.5 μL,DNA模板2 μL(1~10 ng),灭菌超纯水7 μL。将加好样的96-PCR板置于荧光定量PCR仪进行扩增反应,每个样品3个重复。扩增反应条件为95 ℃预变性30 s;95℃变性5 s,60 ℃退火40 s,72 ℃延伸30 s,40个循环。
|
|
表 1 定量PCR扩增引物 Table 1 Amplification primer of quantitative PCR |
应用SPSS 17.0统计软件进行单因素方差分析(One-way ANOVA)、Pearson相关性分析。
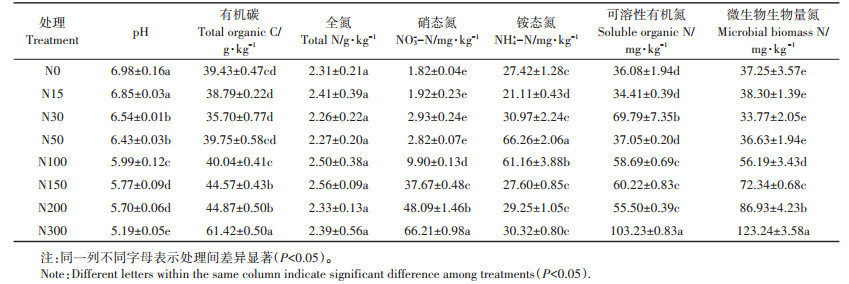
2 结果与分析 2.1 氮沉降对土壤化学性质的影响连续6年不同氮添加处理下,土壤化学性质变化见表 2。高氮添加(N100、N150、N200和N300)土壤有机碳、硝态氮和微生物生物量氮含量高于或显著高于低氮添加(N15、N30和N50)和对照N0。7个氮添加处理的土壤pH均低于或显著低于对照N0,且高氮添加pH显著低于低氮添加。土壤全氮含量在不同氮添加处理间无显著差异。
|
|
表 2 不同氮添加处理下土壤化学性质 Table 2 Soil chemical properties in different nitrogen addition treatments |
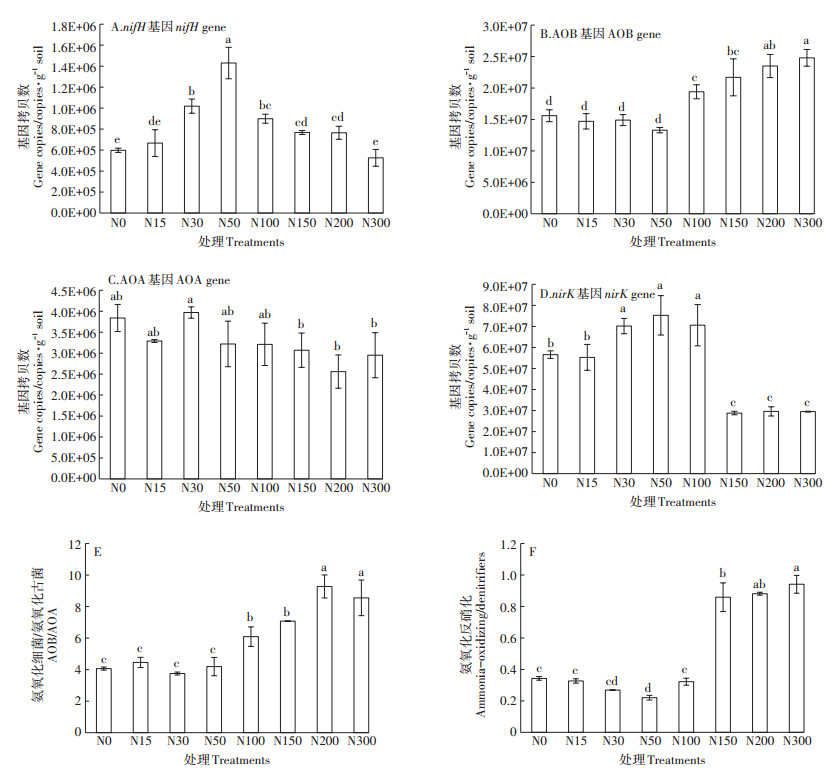
固氮微生物功能基因nifH丰度在各氮添加处理中的变化范围在5.26×105~1.43×106 copies·g-1 soil之间(图 1A)。随着氮添加水平的升高,nifH丰度表现为先升高后降低的趋势,其中N50处理是对照N0的2.39倍。N15、N30、N50、N100、N150和N200处理nifH丰度高于或显著高于对照N0。N300处理nifH丰度低于对照N0,但无显著差异(P > 0.05)。
|
图 1 不同氮处理的功能基因丰度 Figure 1 Functional genes in different nitrogen treatments |
氨氧化细菌AOB-amoA基因丰度在各氮添加处理中的变化范围为1.33×107~2.48×107 copies·g-1 soil之间(图 1B)。N15、N30和N50处理AOB-amoA 基因丰度与对照相比无显著差异(P > 0.05),高氮添加(N100、N150、N200和N300)AOB-amoA 基因丰度均显著高于对照N0(P < 0.05)。表明氮沉降增加使AOB-amoA基因丰度提高。氨氧化古菌AOA-amoA 基因丰度在各氮添加处理中的变化范围为2.56×106~ 3.97×106 copies·g-1 soil之间(图 1C)。N30处理AOAamoA基因丰度最高,但与对照相比无显著差异,其他6个氮添加处理均低于对照N0。土壤中AOB的基因拷贝数远高于AOA基因拷贝数,且高氮添加显著提高了AOB/AOA比值(图 1E)。
土壤反硝化功能基因nirK丰度在不同氮添加处理中的变化范围为2.88×107~7.53×107 copies·g-1 soil之间(图 1D)。N15处理nirK基因丰度低于对照N0,但无显著差异(P > 0.05);N30、N50和N100处理nirK基因丰度均显著高于对照N0(P < 0.05);高氮添加处理(N150、N200和N300)nirK基因丰度显著低于对照N0(P < 0.05),显著提高了氨氧化/反硝化丰度比值(图 1F)。
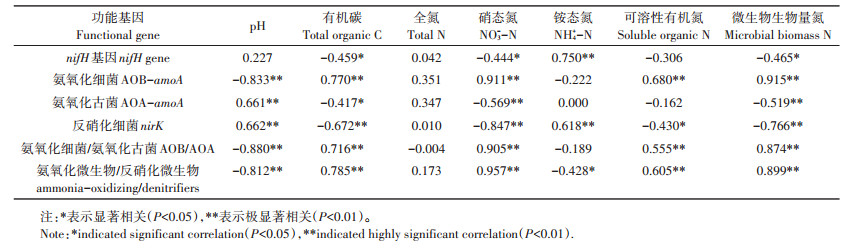
2.3 土壤氮素转化功能基因丰度与土壤因子的相关性土壤氮转化功能基因拷贝数与土壤化学性质相关性分析见表 3。土壤nifH基因拷贝数与总有机碳、硝态氮和微生物生物量氮含量呈显著负相关(P < 0.05),与土壤铵态氮含量呈极显著正相关(P < 0.01)。土壤AOB-amoA基因拷贝数与有机碳、硝态氮、可溶性有机氮和微生物生物量氮含量呈极显著正相关(P < 0.01),与土壤pH呈极显著负相关(P < 0.01)。土壤AOA-amoA基因拷贝数与土壤pH呈极显著正相关(P < 0.01),与土壤有机碳、硝态氮和微生物生物量氮含量呈显著负相关(P < 0.05)。土壤nirK 基因拷贝数与土壤pH和铵态氮含量呈极显著正相关关系(P < 0.01),与土壤有机碳、硝态氮、可溶性有机氮和微生物生物量氮含量呈显著负相关关系(P < 0.05)。AOB/AOA与土壤有机碳、硝态氮、可溶性有机氮和微生物生物量氮呈极显著正相关(P < 0.01),与pH呈极显著负相关(P < 0.01)。氨氧化/反硝化与土壤有机碳、硝态氮、可溶性有机氮和微生物生物量氮呈极显著正相关(P < 0.01),与pH和铵态氮含量呈显著负相关(P < 0.05)。
|
|
表 3 功能基因拷贝数与化学性质之间的相关性分析 Table 3 Correlation analysis between functional gene copies and soil chemical properties |
土壤固氮微生物是一类重要的氮转化功能微生物,其能够将空气中的氮还原成氨供给植物吸收利用。固氮微生物数量与输入土壤的外源氮素密切相关,施氮量过高会降低固氮菌的丰度并抑制固氮酶的活性,并对固氮菌群落组成产生显著影响[26]。本研究中,随着氮添加水平的升高,nifH丰度表现为先升高后降低趋势,N300处理降低了nifH基因相对丰度,其他6个氮添加处理均高于或显著高于对照N0。即低于200 kg N·hm-2·a-1时更有利于固氮菌的生长,而高于300 kg N·hm-2·a-1时反而抑制其生长。这与刘彩霞等[27]得出的氮沉降(铵态氮)高于60 kg N·hm-2·a-1抑制杉木林固氮功能微生物生长结论一致。Zhang等[7]研究也表明,高浓度无机氮添加抑制固氮菌生长。前人研究认为,氮肥输入降低固氮菌的丰度是由于土壤酸化或高氮含量造成的[28]。
土壤固氮菌丰度与土壤有机碳含量、pH、有效磷含量等其他化学因子密切相关。本研究相关分析表明,nifH基因丰度与土壤有机碳含量呈显著负相关性。由于生物固氮过程需要消耗大量能量,而该过程在很大程度上依赖于土壤中的有机碳含量。高氮添加虽然提高了土壤有机碳含量,但高氮添加导致高的速效氮含量反而会抑制土壤的生物固氮过程。本研究表明,nifH基因丰度与铵态氮含量呈极显著正相关,与土壤硝态氮含量呈显著负相关。表明氮添加量增加引起土壤可利用性氮含量变化是引起固氮功能基因丰度发生变化的重要驱动因素。Wang等[29]研究表明,长期施肥引起小麦生长季土壤铵态氮含量改变,显著影响固氮菌丰度,不利于土壤微生物的固氮功能。本研究中,低氮添加促进土壤的生物固氮过程,但过高氮添加反而会抑制土壤的生物固氮过程。表明未来连续高氮沉降可能不利于贝加尔针茅草原土壤微生物的固氮功能。
3.2 土壤氨氧化功能基因对氮沉降响应的综合分析硝化作用是氮素转化的关键过程,由含氨单加氧酶基因(amoA)、氨氧化细菌(AOB)和氨氧化古菌(AOA)共同驱动氨氧化过程是硝化作用的限速步骤,其数量和结构组成变化可反映土壤氮素变化[30]。目前氮肥或氮添加对土壤硝化作用的影响研究大多是针对这一步骤开展的。一般情况下,氨是微生物进行硝化作用的直接底物,酸性土壤中铵根离子分解处理的氨较少,从而抑制AOB生长[31]。本研究,低氮添加(N15、N30和N50)对氨氧化细菌AOB相对丰度无显著影响,而高氮添加显著提高了氨氧化细菌丰度。说明连续6 a高氮添加,促进了AOB的生长繁殖。这与Li等[32]对内蒙古草原10 a氮添加试验AOB基因丰度变化结果一致。Ai等[33]在对小麦玉米轮作潮土研究表明,氮肥添加可显著提高AOB的数量。其可能原因是土壤中存在对NH4+有较高亲和力的未知的AOB[34]或者是某些AOB的生长不以NH4+作为唯一底物[35]。与AOB丰度变化情况不同,除N30处理与对照相比无显著差异外,其余6种氮添加处理均降低了土壤氨氧化古菌基因丰度,说明长期氮沉降增加会导致土壤氨氧化古菌丰度降低。长期施氮肥导致南方红壤中氨氧化古菌丰度降低[36],这表明尽管草原暗栗钙土和南方红壤在pH、有机质和碳氮水平上存在较大差异,但长期氮添加和长期施用氮肥对这两种土壤中氨氧化古菌的影响存在一致性。在本研究中,各氮添加处理土壤AOB-amoA基因拷贝数比AOA-amoA基因拷贝数高一个数量级,可见本试验环境下更有利于AOB生长。这与Yao等[37]在茶园酸性土壤AOA主导氨氧化作用的研究结果不一致。前人研究表明,AOB和AOA生长对土壤含氮量响应不同,AOB对高氮环境较为适应,而AOA倾向于低氮环境[38]。本试验中高氮添加处理土壤硝态氮含量显著高于低氮添加和对照处理,从而促进了AOB生长。但对于这一原因推测的合理性还需要进一步深入研究。
大量研究表明,多种土壤环境因子,包括pH、温度、土壤类型和利用方式等都会影响氨氧化微生物的群落组成和数量,从而使氮添加对土壤氨氧化微生物影响存在很大不确定性。本研究氨氧化细菌和氨氧化古菌与土壤理化因子的相关分析表明,影响氨氧化功能基因相对丰度的土壤理化因子为pH、有机碳、硝态氮和微生物生物量氮含量。这与Hayden等[39]对澳大利亚农业与草地氮转化功能基因驱动因子研究结果一致。相关分析表明,AOB基因拷贝数与土壤有机碳含量呈极显著正相关,说明连续6 a氮添加提高了贝加尔针茅草原土壤有机碳含量是促进氨氧化细菌生长繁殖的重要因素。一些研究表明,氨氧化细菌适合在氮素含量丰富的土壤中生存,且其数量与土壤pH呈显著负相关[14, 40]。本研究中,氨氧化细菌基因拷贝数与土壤pH呈极显著负相关。这与其研究结果一致。原因可能是,随着氮添加水平的增加,土壤pH降低,使得土壤中耐酸性AOB数量增多[41]。氨氧化古菌与土壤化学因子的相关性分析表明,氨氧化古菌基因拷贝数与土壤pH呈极显著正相关,说明贝加尔针茅草原土壤中AOA基因丰度随着pH降低而显著降低。高氮添加增加了土壤中硝态氮含量,相应的AOB基因拷贝数增加,而AOA基因拷贝数则减少,AOB/AOA比值提高,且随着氮添加水平的增加,提高的幅度明显增加。表明连续6 a高氮添加,改变了氨氧化微生物相对丰度,氨氧化微生物对氨氧化的相对贡献也可能发生改变。但氨氧化微生物对氨氧化作用的相对贡献率是否发生改变,还需采用同位素示踪技术进一步研究。相关分析表明,AOA基因拷贝数与土壤硝态氮含量呈极显著负相关关系,这与Di等[38]的AOA适合于低氮环境的研究结论一致。本研究中长期连续氮添加导致的土壤化学性质变化,尤其是土壤pH降低导致土壤中AOA丰度明显降低,可能降低土壤中AOA的氨氧化功能,进一步影响贝加尔针茅草原土壤的硝化作用。
3.3 土壤反硝化功能基因对氮沉降响应的综合分析反硝化作用是将硝酸根还原为N2O或N2的过程,是由许多厌氧或兼性厌氧微生物参与的一系列酶催化过程。这些酶主要由功能基因硝酸还原酶narG/ napA、亚硝酸还原酶nirK/nirS、一氧化氮还原酶norB 和氧化亚氮还原酶nosZ编码。不同反硝化功能基因型对环境因子的影响存在较大差异。其中,亚硝酸还原酶nirK/nirS催化亚硝酸盐还原成NO是微生物驱动反硝化作用关键步骤。已有研究表明,nirK型反硝化细菌对施肥种类和施肥量都十分敏感[42]。连续6 a氮添加显著影响了nirK相对丰度,N30、N50和N100处理显著提高了nirK相对丰度,N150、N200和N300处理显著降低了nirK相对丰度。表明,高氮添加(N150、N200和N300)对nirK丰度有明显的抑制作用。
环境因子如土壤pH、温度、有机碳、硝态氮含量是影响反硝化作用的重要因素。本研究相关分析表明,nirk相对丰度与土壤pH、有机碳、硝态氮、铵态氮、可溶性有机氮和微生物生物量氮含量均呈显著相关性(表 3)。Xie等[43]研究认为土壤有机碳和硝酸盐含量是驱动青藏高原草甸反硝化功能基因nirK丰度的重要土壤因子。Jahangir等[44]研究表明,反硝化活性的高低与提供给细菌生长的有机质的质量和数量密切相关。土壤反硝化作用最适pH范围为7.0~8.0,在此范围内反硝化作用最好[45]。在本研究中,高氮添加(N150、N200和N300)处理pH显著低于对照N0,降低了土壤pH,抑制了反硝化功能基因nirK丰度,并由此可能对反硝化作用产生影响。一般情况下,土壤AOB基因丰度增加会提高反硝化作物的底物硝酸盐含量,进而促进反硝化微生物驱动的反硝化作用,造成氮素流失。但本研究中,高氮(N150、N200和N300)显著降低了反硝化细菌nirK基因丰度,而硝态氮含量却显著提高。说明高氮添加驱动的土壤氨氧化作用高于驱动的土壤的反硝化作用。土壤nirK基因丰度与土壤硝态氮含量呈极显著负相关验证了这一观点。
上述研究表明,贝加尔针茅草原土壤反硝化活性受土壤化学性质特别是pH、有机碳、硝态氮、铵态氮、可溶性有机氮和微生物生物量氮含量的显著影响。此外,由于不同反硝化过程所涉及的酶不同,同时携带不同反硝化基因的微生物种群对环境因子响应的差异,因此,揭示不同氮添加水平对反硝化功能微生物基因丰度的影响,需要对不同类型的反硝化微生物开展深入研究。
4 结论(1)随着氮添加水平的增加,固氮细菌nifH基因丰度呈现先升高后降低趋势。低于200 kg N·hm-2· a-1时促进固氮菌生长。影响固氮菌nifH 主要环境因子是土壤有机碳、铵态氮、硝态氮和微生物生物量氮含量。
(2)高氮添加(N100、N150、N200和N300)提高了AOB基因丰度,降低了AOA基因丰度,并显著提高了AOB/AOA,表明氮添加量增加可能使AOB在贝加尔针茅草原土壤硝化过程中占主导作用。影响AOB、AOA基因丰度的主要环境因子是土壤pH、有机碳、硝态氮和微生物生物量氮含量。
(3)高氮添加(N150、N200和N300)降低了反硝化细菌nirK基因丰度。氮添加量的增加促进了AOB主导的氨氧化过程,而反硝化微生物nirK丰度的降低提高了氨氧化产物的积累,继而提高了土壤中的硝态氮含量。影响反硝化细菌nirK的主要环境因子是土壤pH、有机碳、硝态氮、铵态氮、可溶性有机氮和微生物生物量氮含量。
| [1] |
Liu L, Zhang T, Gilliam F S, et al. Interactive effects of nitrogen and phosphorus on soil microbial communities in a tropical forest[J]. PLoS One, 2013, 8(4): e61188. DOI:10.1371/journal.pone.0061188 |
| [2] |
Eisenlord S D, Freedman Z, Zak D R, et al. Microbial mechanisms mediating increased soil C storage under elevated atmospheric N deposition[J]. Applied and Environmental Microbiology, 2013, 79: 1191-1199. DOI:10.1128/AEM.03156-12 |
| [3] |
Bai Y F, Wu J G, Clark C M, et al. Tradeoffs and thresholds in the effects of nitrogen addition on biodiversity and ecosystem functioning:Evidence from Inner Mongolia grasslands[J]. Global Change Biology, 2010, 16(1): 358-372. DOI:10.1111/j.1365-2486.2009.01950.x |
| [4] |
Hasselquist N J, Hogberg P. Dosage and duration effects of nitrogen additions on ectomycorrhizal sporocarp production and functioning:An example from two N-limited boreal forests[J]. Ecology and Evolution, 2014, 4(15): 3015-3026. DOI:10.1002/ece3.1145 |
| [5] |
BassiriRad H. Consequences of atmospheric nitrogen deposition in terrestrial ecosystems, old questions, new perspectives[J]. Oecologia, 2015, 177(1): 1-3. DOI:10.1007/s00442-014-3116-2 |
| [6] |
杨涵越, 张婷, 黄永梅, 等. 模拟氮沉降对内蒙古克氏针茅草原N2O排放的影响[J]. 环境科学, 2016, 37(5): 1900-1907. YANG Han-yue, ZHANG Ting, HUANG Yong-mei, et al. Effect of stimulated N deposition on N2O emission from a Stipa krylovii Steppe in Inner Mongolia, China[J]. Environmental Science, 2016, 37(5): 1900-1907. |
| [7] |
Zhang X M, Han X G. Nitrogen deposition alters soil chemical properties and bacterial communities in the Inner Mongolia grassland[J]. Journal of Environmental Science, 2012, 24(8): 1483-1491. DOI:10.1016/S1001-0742(11)60900-5 |
| [8] |
Jorquera M A, Martinez O A, Marileo L G, et al. Effects of nitrogen and phosphorus fertilization on the composition of rhizobacterial communites of two Chilean Andisol pastures[J]. World Journal of Microbiology and Biotechnology, 2014, 30(1): 99-107. |
| [9] |
Orr C H, Leifert C, Cummings S P, et al. Impacts of organic and conventional crop management on diversity and activity of free-living nitrogen fixing bacteria and total bacteria are subsidiary to temporal effects[J]. PLoS One, 2012, 7: 483-496. |
| [10] |
Zhang X M, Liu W, Schloter M, et al. Response of the abundance of key soil microbial nitrogen-cycling genes to multi-factorial global changes[J]. PLoS One, 2013, 8(10): e76500. DOI:10.1371/journal.pone.0076500 |
| [11] |
Ning Q S, Gu Q, Shen J P, et al. Effects of nitrogen deposition rates and frequencies on the abundance of soil nitrogen-related functional genes in temperate grassland of northern China[J]. Journal of Soils and Sediments, 2015, 15(3): 694-704. DOI:10.1007/s11368-015-1061-2 |
| [12] |
Shen X Y, Zhang L M, Shen J P, et al. Nitrogen loading levels affect abundance and composition of soil ammonia oxidizing prokaryotes in semiarid temperate grassland[J]. Journal of Soils and Sediments, 2011, 11(7): 1243-1252. DOI:10.1007/s11368-011-0375-y |
| [13] |
Zhou J, Guan D W, Zhou B K, et al. Influence of 34-years of fertilization on bacterial communities in an intensively cultivated black soil in northeast China[J]. Soil Biology and Biochemistry, 2015, 90: 42-51. DOI:10.1016/j.soilbio.2015.07.005 |
| [14] |
Wang C H, Zhu F, Zhao X, et al. The effects of N and P additions on microbial N transformations and biomass on saline-alkaline grassland of loess plateau of northern China[J]. Geoderma, 2014, 213(1): 419-425. |
| [15] |
Wang J, Bao J T, Su J Q, et al. Impact of inorganic nitrogen additions on microbes in biological soil crusts[J]. Soil Biology & Biochemistry, 2015, 88: 303-313. |
| [16] |
刘红梅, 张海芳, 皇甫超河, 等. 长期氮添加对贝加尔针茅草原土壤微生物群落多样性的影响[J]. 农业环境科学学报, 2017, 36(4): 709-717. LIU Hong-mei, ZHANG Hai-fang, HUANGFU Chao-he, et al. Effects of different long-term nitrogen addition on soil microbial diversity of Stipa baicalensis steppe in Inner Mongolia, China[J]. Journal of Agro-Environment Science, 2017, 36(4): 709-717. |
| [17] |
莫江明, 薛璟花, 方运霆. 鼎湖山主要森林植物凋落物分解及其对N沉降的响应[J]. 生态学报, 2004, 24(7): 1413-1420. MO Jiang-ming, XUE Jing-hua, FANG Yun-ting. Litter decomposition and its responses to simulated N deposition for the major plants of Dinghushan forests in subtropical China[J]. Acta Ecologica Sinica, 2004, 24(7): 1413-1420. DOI:10.3321/j.issn:1000-0933.2004.07.015 |
| [18] |
Clark C M, Cleland E E, Collins S L, et al. Environmental and plant community determinants of species loss following nitrogen enrichment[J]. Ecology Letters, 2007, 10(7): 596-607. DOI:10.1111/j.1461-0248.2007.01053.x |
| [19] |
鲍士旦. 土壤农化分析[M]. 第三版. 北京: 中国农业出版社, 2000. BAO Shi-dan. Soil and agricultural chemistry analysis[M]. Third Edition. Beijing: China Agricultural Press, 2000. |
| [20] |
吴金水, 林启美, 黄巧云, 等. 土壤微生物生物量测定方法及其应用[M]. 北京: 气象出版社, 2006. WU Jin-shui, LIN Qi-mei, HUANG Qiao-yun, et al. Determination of soil microbial biomass and its application[M]. Beijing: China Meteorological Press, 2006. |
| [21] |
倪银霞, 黄懿梅, 牛丹, 等. 宁南山区林地土壤原位矿化过程中碳氮转化耦合特征[J]. 环境科学, 2015, 36(9): 3401-3410. NI Yin-xia, HUANG Yi-mei, NIU Dan, et al. Characteristics and coupling relationship of soil carbon and nitrogen transformation during in-situ mineralization cultivation in forestlands in the mountain area of southern Ningxia[J]. Environmental Science, 2015, 36(9): 3401-3410. |
| [22] |
Rösch C, Mergel A, Bothe H. Biodiversity of denitrifying and dinitrogen-fixing bacteria in an acid forest soil[J]. Applied & Environmental Microbiology, 2002, 68(8): 3818-3829. |
| [23] |
Rotthauwe J H, Witzel K P, Liesack W. The ammonia monooxygenase structural gene amoA as a functional marker:Molecular fine-scale analysis of natural ammonia-oxidizing populations[J]. Applied and Environmental Microbiology, 1997, 63(12): 4704-4712. |
| [24] |
梁龙, 梁小兵. 氨氧化细菌和氨氧化古菌在百花湖沉积物中的垂直分布[J]. 矿物岩石地球化学通报, 2014, 33(2): 221-225. LIANG Long, LIANG Xiao-bing. Vertical distribution of ammonia oxidizing bacteria(AOB)and ammonia oxidizing archaea in the sediments of lake Baihua[J]. Geochemical Report of Minerals and Rocks, 2014, 33(2): 221-225. DOI:10.3969/j.issn.1007-2802.2014.02.010 |
| [25] |
Bremer C, Braker G, Matthies D, et al. Impact of plant functional group, plant species, and sampling time on the composition of nirK-type denitrifier communities in soil[J]. Applied & Environmental Microbiology, 2007, 73(21): 6876-6884. |
| [26] |
Reardon C L, Gollany H T, Wuest S B. Diazotroph community structure and abundance in wheat-fallow and wheat-pea crop rotations[J]. Soil Biology & Biochemistry, 2014, 69: 406-412. |
| [27] |
刘彩霞, 焦如珍, 董玉红, 等. 模拟氮沉降对杉木林土壤氮循环相关微生物的影响[J]. 林业科学, 2015, 51(4): 96-102. LIU Cai-xia, JIAO Ru-zhen, DONG Yu-hong, et al. Response of the N-Cycling associated soil microorganism to simulated N deposition in a plantation of Cunninghamia lanceolata[J]. Scientia Silvae Sinicae, 2015, 51(4): 96-102. |
| [28] |
Wang C, Zheng M M, Song W F, et al. Impact of 25 years of inorganic fertilization on diazotrophic abundance and community structure in an acidic soil in southern China[J]. Soil Biology and Biochemistry, 2017, 113: 240-249. DOI:10.1016/j.soilbio.2017.06.019 |
| [29] |
Wang J C, Zhang D, Zhang L, et al. Temporal variation of diazotrophic community abundance and structure in surface and subsoil under four fertilization regimes during a wheat growing season[J]. Agriculture Ecosystems and Environment, 2016, 216: 116-124. DOI:10.1016/j.agee.2015.09.039 |
| [30] |
Li S, Shakoor A, Wubet T, et al. Fine-scale variations of fungal community in a heterogeneous grassland in Inner Mongolia:Effects of the plant community and edaphic parameters[J]. Soil Biology and Biochemistry, 2018, 122: 104-110. DOI:10.1016/j.soilbio.2018.04.007 |
| [31] |
Xia W, Zhang C, Zeng X, et al. Autotrophic growth of nitrifying community in an agricultural soil[J]. The ISME Journal, 2011, 5(7): 1226. DOI:10.1038/ismej.2011.5 |
| [32] |
Li X X, Ying J Y, Chen Y, et al. Effects of nitrogen addition on the abundance and composition of soil ammonia oxidizers in Inner Mongolia grassland[J]. Acta Ecologica Sinica, 2011, 31(31): 174-178. |
| [33] |
Ai C, Liang G Q, Sun J W, et al. Different roles of rhizosphere effect and long-term fertilization in the activity and community structure of ammonia oxidizers in a calcareous fluvo-aquic soil[J]. Soil Biology and Biochemistry, 2013, 57: 30-42. DOI:10.1016/j.soilbio.2012.08.003 |
| [34] |
Yao H, Campbell C D, Chapman S J, et al. Multi-factorial drivers of ammonia oxidizer communities:Evidence from a national soil survey[J]. Environmental Microbiology, 2013, 15(9): 2545-2556. DOI:10.1111/1462-2920.12141 |
| [35] |
Tago K, Okubo T, Shimomura Y, et al. Environmental factors shaping the community structure of ammonia-oxidizing bacteria and archaea in sugarcane field soil[J]. Microbes and Environments, 2015, 30(1): 21-28. DOI:10.1264/jsme2.ME14137 |
| [36] |
张苗苗, 王伯仁, 李冬初, 等. 长期施加氮肥及氧化钙调节对酸性土壤硝化作用及氨氧化微生物的影响[J]. 生态学报, 2015, 35(19): 6362-6370. ZHANG Miao-miao, WANG Bo-ren, LI Dong-chu, et al. Effects of long-term N fertilizer application and liming on nitrification and ammonia oxidizers in acidic soils[J]. Acta Ecologica Sinica, 2015, 35(19): 6362-6370. |
| [37] |
Yao H Y, Gao Y M, Nicol G W, et al. Links between ammonia oxidizer community structure, abundance, and nitrification potential in acidic soils[J]. Applied and Environmental Microbiology, 2011, 77(13): 4618-4625. DOI:10.1128/AEM.00136-11 |
| [38] |
Di H J, Cameron K C, Shen J P, et al. Ammonia-oxidizing bacteria and archaea grow under contrasting soil nitrogen conditions[J]. FEMS Microbiology Ecology, 2010, 72: 386-394. DOI:10.1111/j.1574-6941.2010.00861.x |
| [39] |
Hayden H L, Drake J, Imhof M, et al. The abundance of nitrogen cycle genes amoA and nifH depends on land-uses and soil types in South-Eastern Australia[J]. Soil Biology & Biochemistry, 2010, 42(10): 1774-1783. |
| [40] |
杨亚东, 张明才, 胡君蔚, 等. 施氮肥对华北平原土壤氨氧化细菌和古菌数量及群落结构的影响[J]. 生态学报, 2017, 37(11): 3636-3646. YANG Ya-dong, ZHANG Ming-cai, HU Jun-wei, et al. Effects of nitrogen fertilizer application on abundance and community structure of ammonia oxidizing bacteria and archaea in a North China agricultural soil[J]. Acta Ecologica Sinica, 2017, 37(11): 3636-3646. |
| [41] |
任灵玲, 李秀玲, 刘灵芝. 不同施肥方式下土壤氨氧化细菌的群落特征[J]. 中国生态农业学报, 2019, 27(1): 11-19. REN Ling-ling, LI Xiu-ling, LIU Ling-zhi. Community characteristics of soil ammonia oxidizing bacteria after different fertilizer applications[J]. Chinese Journal of Eco-Agriculture, 2019, 27(1): 11-19. |
| [42] |
Yin C, Fan F L, Song A L, Li Z, et al. Different denitrification potential of aquic brown soil in Northeast China under inorganic and organic fertilization accompanied by distinct changes of nirS- and nirK- denitrifying bacterial community[J]. European Journal of Soil Biology, 2014, 65: 47-56. DOI:10.1016/j.ejsobi.2014.09.003 |
| [43] |
Xie Z, Roux X L, Wang C P, et al. Identifying response groups of soil nitrifiers and denitrifiers to grazing and associated soil environmental drivers in Tibetan alpine meadows[J]. Soil Biology & Biochemistry, 2014, 77(7): 89-99. |
| [44] |
Jahangir M M R, Khalil M I, Johnston P, et al. Denitrification potential in subsoils:A mechanism to reduce nitrate leaching to groundwater[J]. Agriculture, Ecosystems & Environment, 2012, 147: 13-23. |
| [45] |
刘秋丽, 马娟娟, 孙西欢, 等. 土壤的硝化-反硝化作用因素研究进展[J]. 农业工程, 2011, 1(4): 79-83, 13. LIU Qiu-li, MA Juan-juan, SUN Xi-huan, et al. Research advancement on soil nitrification-denitrification and its influencing factors[J]. Agricultural Engineering, 2011, 1(4): 79-83, 13. |
 2019, Vol. 38
2019, Vol. 38