2. 农业农村部环境保护科研监测所, 天津 300191;
3. 天津商业大学生物技术与食品科学学院, 天津 300134
2. Agro-Environmental Protection Institute, Ministry of Agriculture and Rural Affairs, Tianjin 300191, China;
3. College of Biotechnology and Food Science, Tianjin University of Commerce, Tianjin 300134, China
由于抗生素具有提高畜禽生长速度、预防和治疗畜禽疾病的作用,因此其在畜禽养殖场中的使用较为广泛,据专家统计,我国用于畜禽养殖业方面的抗生素占全国抗生素总使用量的52%[1]。但大量与长期使用抗生素会增加畜禽肠道中耐药菌的产生与累积,耐药菌与粪便共同进入环境,使畜禽粪便成为环境中抗生素抗性基因(Antibiotic resistance genes,ARGs)的重要来源[2]。此外,由于ARGs不仅在垂直转移方式上可亲代传递,其还具有水平转移的能力,可在养殖场周边的环境介质微生物中传播和扩散[3],甚至会扩散到致使人类患病的细菌中,从而威胁到人类健康[4]。已有很多学者对不同畜禽养殖场及周边环境中ARGs的污染状况进行了研究,研究主要集中在畜禽养殖场粪便、污水以及粪肥还田后土壤中ARGs的污染丰度和种类方面。例如,闫书海[5]对浙江和杭州不同规模养殖场(猪、鸡和鸭)的粪便和污水中2类典型ARGs进行检测,发现四环素类的tetA、tetG、tetM、tetO、tetQ、tetW与磺胺类的sul1、sul2在所有样品中均有检出,且磺胺类ARGs的相对丰度普遍高于四环素类;彭晶等[6]在施粪肥后的土壤中检出了大环内酯类中的erm35、ermB、ermT、ermX、ermF和erm36,其中丰度最高和最低的分别为ermB和erm36;MCKINNEY等[7]在猪场和鸡场氧化塘沉积物中检测出磺胺类ARGs(sul1和sul2)和四环素类ARGs(tetO和tetW),且含量为猪场高于鸡场;YUAN等[8]在武汉畜禽养殖场的废水中检测到了四环素类tetA、磺胺类sul1以及喹诺酮类oqxB等常见的ARGs。
综上所述,目前国内外研究主要聚焦于ARGs的污染水平和种类等污染特征分析。然而,随着集约化养殖场的发展和在环保要求下,畜禽养殖场采用的不同粪污处理工艺,粪污中污染物的转归和降解日益受到重视,但是这些粪污处理工艺对ARGs的去除效果仍不太清楚,尤其是完全针对畜禽养殖场粪便和污水不同处理工艺中ARGs的赋存规律与去除效果的研究十分有限。因此,本文以天津市猪场、牛场、鸡场中典型粪污处理工艺为研究对象,研究目的:(1)分析不同畜禽养殖场新鲜粪污中ARGs的总体污染特征;(2)探讨不同养殖场污水处理工艺对ARGs的总体去除效果,以及不同处理单元中ARGs的赋存规律;(3)比较不同粪便处理工艺对ARGs的去除效果,以及关键环节中ARGs的赋存规律。从而为控制我国畜禽养殖场中ARGs污染与减少周边环境耐药性传播提供科学依据,为养殖场粪污中ARGs的消减提供基础数据。
1 材料与方法 1.1 养殖场的基本信息本次试验选取天津市具有较为常见、通用和完整的粪污处理工艺的3类畜禽养殖场为研究对象,其中牛场粪便的处理方式主要是堆肥和灭菌晾晒作垫料,两者基本原理均为好氧发酵,区别主要是灭菌作垫料处理是为了保护牛的健康增加灭菌流程;鸡场粪便的处理方式为堆肥;猪场使用异位发酵床对畜禽粪便进行处理,异位发酵床与堆肥基本原理均为好氧发酵,但异位发酵床中有芽孢杆菌、酵母菌和乳酸菌等多种菌株参与。牛场和猪场对污水的处理方式都主要以厌氧发酵为主。三家养殖场粪污处理工艺流程如图 1所示。

|
图 1 三家养殖场粪污处理工艺流程 Figure 1 Manure treatment process flow of three farms |
牛场,猪场和鸡场的地理位置分别为39°14′29″ N、117°34′12″E,39°21′31″N、117°46′01″E和39°44′ 33″ N、117°14′ 18″ E;存栏量分别为2 000、1 500头和600 000只;粪便日排放量分别为4 000、30 000 kg·d-1和60 000 kg·d-1,牛场和猪场污水日排放量分别为20 000 L·d-1和72 000 L·d-1。
1.2 样品的采集、运输和保存试验样品采集于2019年7月上旬,采集的样品为畜禽新鲜粪污,以及各处理单元中粪污,具体采样点如图 1所示。采集液体样品时,均设置多点采集,然后混合均匀为1份样品,连续采样3 d,作为每个采样点的3个平行样品,共计27个样品(猪场12个和牛场15个);固体样品为多点采集并混合为均一样品,不同采样点采集3个平行样品,共计45个样品(猪场9个、牛场15个和鸡场21个)。采集后的样品放到含有冰袋的白色泡沫箱中,低温储存,5 h内送到实验室-20 ℃的冰箱保存,等待处理。
1.3 样品的预处理固体样品放入冷冻干燥机中去除水分,并将得到的干样磨碎,混合,过2 mm的网筛,液体样品过5 µm滤膜以去除漂浮的杂质。在提取DNA前,将样品于-20 ℃冷冻储存。
1.4 基因组DNA的提取与PCR反应试验使用的Fast DNA SPIN Kit for soil试剂盒购买于美国MP Biomedicals生物医药公司,对样品DNA的提取严格遵守试剂盒的操作指南,提取出DNA的纯度采用超微量紫外可见分光光度计检测。以目标DNA模板进行PCR检测,PCR反应体系为25 µL,包括12.5 µL 2×EasyTaq PCR SuperMix(0.1 U·μL-1 Taq DNA Polymerase、2×PCR buffer、3 mmol·L-1 MgCl 2和0.4 mmol·L-1 dNTPs),前后端引物各0.5 µL(10 μmol·L-1),0.5 µL DNA模板和11 µL ddH2O。反应条件:95 ℃预变性5 min,95 ℃变性30 s,退火30 s后,72 ℃延伸30 s,共35个循环。基因引物的详细信息如表 1所示。反应结束后,采用DNA凝胶电泳检测,观察条带是否有目的基因存在。
|
|
表 1 PCR引物的详细信息 Table 1 The detail information of PCR primer |
试验检测目标包含四环素类(tetX、tetW、tetQ、tetO和tetL)、磺胺类(sul1和sul2)、大环内酯类(ermB和ermC)、喹诺酮类(oqxB、qnrB和qnrS)、链霉素类(strA)抗性基因以及基因元件intI1,PCR引物的详细信息如表 1所示。qPCR的反应体系为20 µL:TB Green Premix Ex Taq 10.0 µL、前后端引物各0.4 µL、ROX Reference DyeⅡ0.4 µL、ddH2O 6.8 µL和DNA模板(已稀释10倍)2 µL(对照以ddH2O为模板),3个平行,用7500实时荧光定量PCR仪(Applied Biosystems美国)进行实时qPCR检测。qPCR扩增过程:95 ℃预变性30 s,95 ℃变性5 s,60 ℃退火34 s,40个循环。在60~95 ℃之间进行熔点曲线分析,根据标准曲线[9]计算出目标基因的拷贝数,随后计算出目的基因的相对丰度(ARGs拷贝数/16S rRNA基因拷贝数)。抗性基因去除率计算公式:
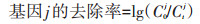
|
式中:j表示特定的基因;C0j为基因j处理前的相对丰度;Ctj为基因j处理后的相对丰度。
1.6 数据分析相对丰度、平均值、标准差和lg值在Excel 2007中计算;使用R软件的Pheatmap制作热图;堆积柱状图使用Origin2017绘制;采样流程图使用Word绘制。
2 结果与讨论 2.1 不同畜禽养殖场原始粪污中ARGs的污染特征新鲜粪污中ARGs的丰度和种类如图 2所示,13种ARGs与intI1在被检测样品中全部检出,这表明目标ARGs在畜禽养殖场粪污中普遍存在。值得注意的是,在养殖场粪污的5类ARGs中,喹诺酮类ARGs的相对丰度最低,为10-7~10-5,四环素类和磺胺类ARGs的相对丰度普遍较高,分别为10-4~10-1和10-4~10-2,其原因可能是养殖场粪污中四环素和磺胺类抗生素残留水平较高。例如,阮蓉等[18]在检测天津市20家牛场、猪场和鸡场粪污中的抗生素残留情况后,发现3种畜禽养殖场中四环素类抗生素污染水平较高,这可能是导致四环素类ARGs污染水平较高的原因。对于不同畜禽粪便,3个畜种养殖场粪便中ARGs的污染特征有较大差异,鸡场和猪场粪便中大环内酯类ARGs的相对丰度(10-4~10-2)明显高于牛场(10-6~ 10-5);鸡场和牛场中磺胺类ARGs的相对丰度(4.85× 10-4~1.36×10-3)低于猪场(1.36×10-3~3.63×10-3)。此外,鸡场和猪场粪便中目标四环素类ARGs的相对丰度普遍较高(10-4~10-1),这与邹威等[19]对华北地区鸡场和猪场粪便中ARGs的相对丰度比较后的结果相似,这可能与四环素类抗生素使用量大以及该类ARGs较高的水平扩散能力有关。猪场和牛场污水中ARGs的污染特征如图 2中PW和CW所示,除喹诺酮类外,其他类ARGs污染水平普遍较高,这说明养殖污水同样是ARGs的储存库。值得注意的是,猪场污水中四环素类、磺胺类和大环内酯类ARGs的相对丰度(10-3~10-2)明显高于牛场的(10-6~10-4),这与ZHANG等[20]对猪场和牛场污水中ARGs丰度的对比结果一致。
|
JM:鸡场粪便;PM:猪场粪便;CM:牛场粪便;PW:猪场污水;CW:牛场污水 JM: chicken farm feces; PM: pig farm feces; CM: cattle farm feces; PW: pig farm sewage; CW: cattle farm sewage 图 2 鸡场、猪场和牛场原始粪污中ARGs的相对丰度 Figure 2 Relative abundance of ARGs in raw manure from chicken, pig and cattle farm |
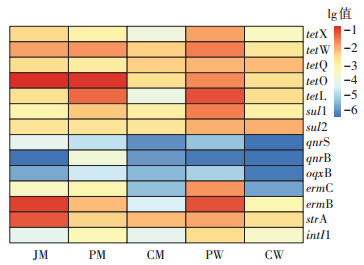
猪场污水各处理单元中13种ARGs和基因元件intI1的相对丰度如图 3(A)所示,除在氧化塘中未检出qnrB外,其余ARGs均在所有处理单元中检出。5类目标ARGs中,喹诺酮类ARGs的相对丰度最低,为4.31×10-7~9.61×10-5,其余ARGs的相对丰度普遍较高,其中四环素类ARGs中tetO和tetL(10-3~10-2)普遍高于tetX和tetQ(10-4~10-3),大环内酯类ermB的相对丰度(10-3~10-2)普遍高于ermC(10-4~10-2)。
|
图 3 不同污水处理单元中ARGs的相对丰度 Figure 3 The relative abundance of ARGs in sewage from different treatment units |
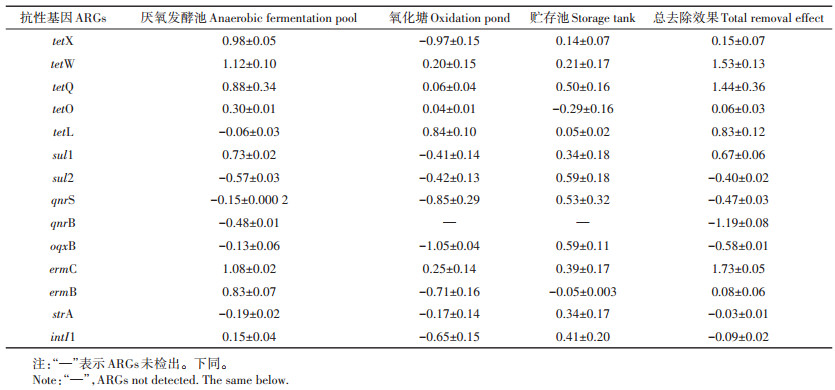
表 2为猪场污水中13种ARGs及基因元件intI1在各处理单元中的去除效果和总去除效果。首先分析猪场污水处理系统对ARGs的总体去除情况,其中tetX、tetW、tetQ、tetO、tetL、ermC、ermB和sul1总体呈现正去除效果,相对丰度(lg值)下降了0.06(tetO)~1.53(tetW),而sul2、qnrS、qnrB、oqxB和strA的相对丰度(lg值)上升了0.03(strA)~1.19(qnrB)。CHEN等[21]发现厌氧发酵处理对江苏省猪场污水中四环素类tetW、tetQ、tetO与大环内酯类ermC、ermB有良好的去除效果,同时未能有效去除喹诺酮类qnrS、oqxB与磺胺类sul1、sul2,与本试验的结果一致。intI1相对丰度(lg值)增加了0.09。此外,不同处理单元对ARGs的去除效果不同,ARGs去除效果最好的主要集中在厌氧发酵池和贮存池,其中贮存池为该处理工艺的最后一环,污水储存的时间较长,可继续进行厌氧或兼性厌氧等发酵反应,此外,污水中微生物群落的变化,同样可能对ARGs产生一定的降解作用。tetX、tetW、tetQ、tetO、sul1、ermC和ermB在厌氧发酵单元中降低较多,其中tetW的去除效果最好,相对丰度(lg值)下降了1.12;tetQ、sul2、qnrS、oqxB和ermC的相对丰度在贮存池单元中下降较多,其中sul2和oqxB的去除效果最好,相对丰度(lg值)均下降了0.59。
|
|
表 2 猪场污水中ARGs在不同处理单元中的去除效果(lg值) Table 2 Removal effect of ARGs in pig farm sewage in different treatment units(lgs) |
如图 3(B)所示,在对牛场污水中13种ARGs及基因元件intI1检测后发现,除了oqxB和ermB外,其余目标ARGs在所有处理单元中均有检出,其中磺胺类ARGs相对丰度最高,为6.83×10-4~1.73×10-2;喹诺酮类ARGs相对丰度最低,为3.92×10-7~2.45×10-6,此外,四环素类ARGs中tetX、tetW、tetQ和tetO(10-4~ 10-3)的相对丰度高于tetL(10-5~10-3),大环内酯类ermB(10-4)的相对丰度高于ermC(10-6)。
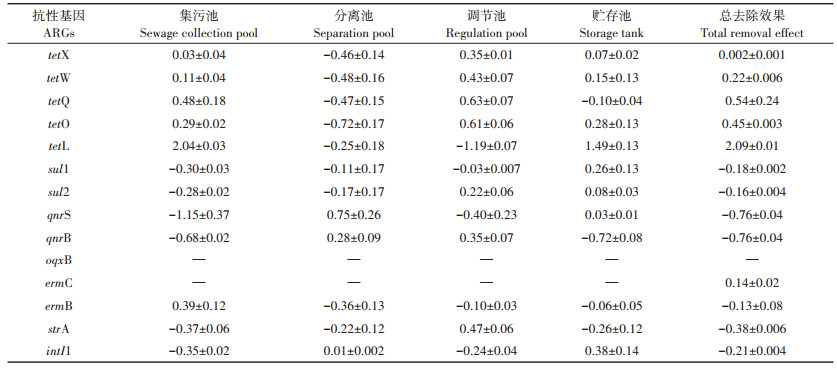
表 3为牛场废水中13种ARGs及基因元件intI1在各处理单元中的去除效果和总去除效果,其中tetX、tetW、tetQ、tetO、tetL和ermC总体呈现正去除效果,相对丰度(lg值)降低了0.002(tetX)~2.09(tetL);其余ARGs均表现出富集现象,其中相对丰度升高最多的为qnrS和qnrB,lg值均升高了0.76,这可能与这几种ARGs的宿主微生物在不同环境条件下具有较强的生存能力有关。此外,ARGs在调节池单元中的去除效果优于其他处理单元,tetX、tetW、tetQ、te⁃ tO、sul2、qnrB和strA在该处理单元中相对丰度均呈现下降趋势,其lg值下降了0.22(sul2)~0.63(tetQ)。分离池中ARGs去除效果最差,13种ARGs中有9种(除qnrB和qnrS外)出现富集现象,其中tetO的相对丰度增加最多(0.72),另外在该单元中,基因元件intI1虽有所去除,但相对丰度(lg值)仅下降了0.01。
|
|
表 3 牛场污水中ARGs在不同处理单元中的去除效果(lg值) Table 3 Removal effect of ARGs in cattle farm sewage in different treatment units(lgs) |
综上所述,畜禽养殖污水处理各单元中ARGs普遍存在,猪场污水中ARGs的检出率为97%,牛场污水中的检出率为95%。猪场和牛场污水处理工艺对ARGs的去除效果有较大差异,猪场污水在厌氧发酵后,8种ARGs的相对丰度下降,lg值平均下降了0.81;牛场污水中6种ARGs的相对丰度下降,lg值平均下降了0.57,值得注意的是,猪场和牛场的污水处理工艺对5种四环素类ARGs均呈现正去除效果,下降最多的为tetL(2.09);此外,猪场污水工艺对tetX、tetW、tetQ、sul1、ermC和ermB的去除效果优于牛场处理工艺,而对tetO和tetL去除能力,则是牛场优于猪场。
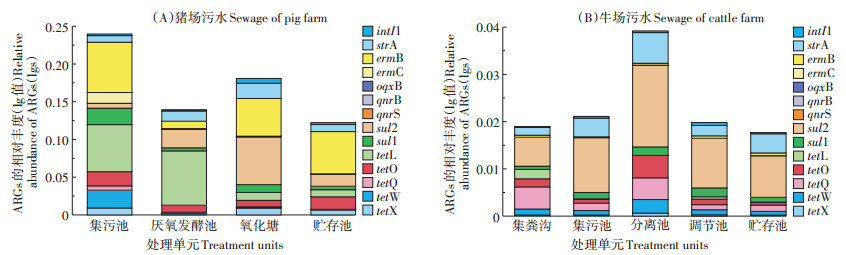
2.3 猪场、牛场和鸡场粪便处理工艺对ARGs的去除效果 2.3.1 猪场粪便处理工艺对ARGs的去除效果图 4(A)为猪场粪便处理工艺各单元中ARGs的污染情况及赋存规律。13种ARGs及基因元件intI1中,除了部分样品中喹诺酮类qnrS、qnrB和oqxB未检出外,其他目标ARGs均有检出,其中tetO相对丰度最高,为2.42×10-3~1.17×10-1,qnrB的相对丰度最低,为6.02×10-6~1.68×10-3,此外,sul1、sul2、strA和intI1在不同处理单元中的相对丰度的数量级没有变化。
|
图 4 不同粪便处理单元中ARGs的相对丰度 Figure 4 The relative abundance of ARGs in different feces treatment units |
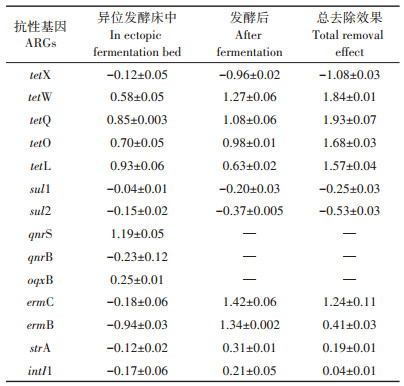
经过粪便处理工艺后,13种目标ARGs以及基因元件intI1的去除效果见表 4。结果表明,处理后的粪便与原始粪便相比,13种目标ARGs以及intI1的相对丰度普遍降低,只有tetX、sul1和sul2出现富集现象,其中tetX的富集现象最为明显,相对丰度的lg值升高了1.08,该工艺对其余4种四环素类ARGs有良好的去除效果,相对丰度(lg值)下降了1.57(tetL)~1.93(tetQ),此外该工艺对粪便中大环内酯类ermC和ermB的去除效果有较大差异,对ermC的去除效果优于ermB,基因元件intI1虽为正去除,但相对丰度(lg值)仅下降了0.04。GUO等[22]发现在猪粪堆肥后,其中四环素类ARGs和大环内酯类ARGs的丰度会降低,其中tetW和ermC的丰度减少90% 以上;TAO等[23]对猪场常温堆肥的研究发现,在完成堆肥后磺胺类sul1和sul2的丰度(lg值)上升1.11~2.49,ZHANG等[24]在研究猪粪堆肥样品时也得出了类似结果。
|
|
表 4 猪场粪便中ARGs在各处理单元中的去除效果(lg值) Table 4 Table 4 Removal effect of ARGs in pig feces in each treatment units (lgs) |
经过处理工艺后,牛场粪便中ARGs相对丰度的变化如图 4(B)所示。在不同处理单元中,磺胺类sul1和sul2的相对丰度最高,为1.51×10-3~4.42×10-2,喹诺酮类qnrS、qnrB和oqxB的相对丰度最低,为1.68 × 10-6~3.21×10-5。同类型ARGs的相对丰度存在较大差异,四环素类tetX、tetW和tetL(10-4~10-3)的相对丰度高于tetO和tetQ(10-6~10-3),大环内酯类ermB(10-4~10-2)高于ermC(10-5~10-6),此外,链霉素类strA和基因元件intI1在整个粪便处理过程中相对丰度的数量级没有变化,分别为10-3和10-4。
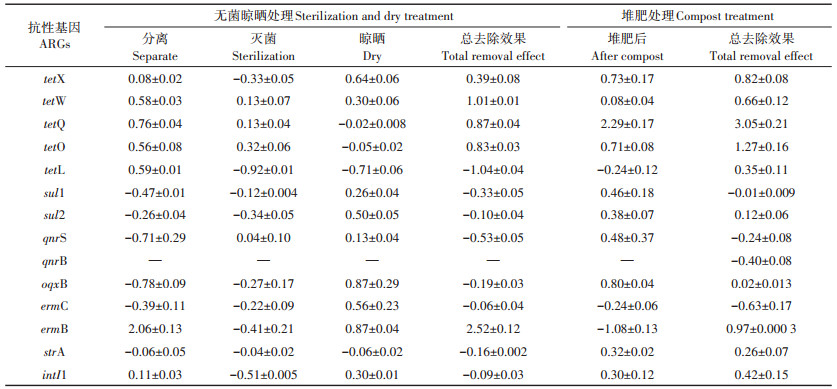
牛场粪便中13种目标ARGs以及基因元件intI1的去除效果如表 5所示。通过比较两种处理手段下ARGs的去除效果发现,牛场粪便在经过堆肥处理后,13种ARGs中有9种的相对丰度(lg值)下降了0.02(oqxB)~3.05(tetQ);4种的相对丰度(lg值)上升了0.01(sul1)~0.63(ermC);intI1的相对丰度(lg值)下降了0.42。而粪便在经过无菌晾晒处理后,有5种ARGs的含量降低,相对丰度(lg值)下降了0.39(tetX)~2.52(ermB);7种相对丰度(lg值)上升了0.06(ermC)~1.04(tetL);intI1的相对丰度(lg值)升高了0.09,值得注意的是,牛粪在经过灭菌处理后,部分ARGs出现富集现象,其原因可能是高温灭菌处理(50~60 ℃)虽然可以消除部分致病菌,但是也可能会促进部分细菌增殖,从而造成耐药基因丰度升高。总体而言,相对于晾晒处理,堆肥处理可能对牛粪中ARGs有更好的去除效果。此外,在堆肥处理后,5种目标四环素类ARGs丰度降低趋势明显,lg值下降了0.35~3.05,这与DIEHL等[25]对牛场堆肥发酵的结果一致,表明堆肥发酵处理可能对大部分四环素类ARGs有较好的去除效果;磺胺类ARGs和喹诺酮类ARGs的去除效果不理想;大环内酯类ermB表现出正去除效果,相对丰度(lg值)下降了0.97,这与彭晶等[26]对牛粪堆肥后大环内酯类ARGs丰度变化的研究结果不一致,这可能与牛粪堆肥原料、堆肥环境以及微生物群落组成的差异有关。
|
|
表 5 牛场粪便中ARGs在各处理单元中的去除效果(lg值) Table 5 Removal effect of ARGs in cattle feces in each treatment unit(lgs) |
随时间变化,好氧堆肥后鸡粪中ARGs相对丰度的变化规律及污染特征如图 4(C)所示。在13种ARGs中,喹诺酮类ARGs的含量最低,相对丰度为3.72×10-7~5.04×10-5,其余目标ARGs的相对丰度均在10-5以上,其中同类ARGs的含量也有差异,磺胺类的相对丰度sul2(10-4~10-2)高于sul1(10-4~10-3),大环内酯类的相对丰度ermB(10-4~10-2)高于ermC(10-4~ 10-3),此外基因元件intI1的相对丰度为4.14×10-5~ 7.21×10-4。
不同堆肥时间下,粪便中ARGs的相对丰度变化如表 6所示。在13种目标ARGs中,tetW、tetQ、tetO、qnrS、oqxB、ermB和strA的相对丰度(lg值)下降了0.27(oqxB)~2.85(tetO),而tetX、tetL、sul1、sul2和ermC出现富集现象,相对丰度(lg值)上升了0.03(tetX)~1.52(sul2),这说明堆肥可能成为了部分ARGs含量增加的良好反应器。就去除不同种类ARGs而言,好氧堆肥对四环素类ARGs的去除效果高于其余ARGs,其中tetW、tetQ和tetO的去除效果最好,相对丰度(lg值)下降了1.20~2.85;磺胺类sul1和sul2呈现负去除效果,相对丰度(lg值)上升了0.96和1.52;大环内酯类ARGs的去除效果有明显差异,其中ermC出现富集现象,相对丰度(lg值)升高了1.43,而ermB的相对丰度(lg值)下降了1.83。LI等[27]在对堆肥26 d后的鸡粪检测中发现,tetW和ermB等四环素类与大环内酯类ARGs的相对丰度降低,磺胺类sul1的相对丰度升高了7.5倍,与本试验ARGs的相对丰度的变化相似。此外,基因元件intI1的相对丰度(lg值)上升了1.24,SELVAM等[28]研究了高温条件下鸡粪堆肥后ARGs的变化,发现intI1明显增值,与本试验的结果一致。值得注意的是,不同堆肥时间,粪便中ARGs的相对丰度呈现波动式变化,第2 d,目标ARGs整体呈现下降趋势,第4 d ARGs整体出现富集现象,其中ermB的相对丰度升高最多,第8 d所有ARGs和intI1的相对丰度均下降,而第16 d ARGs以及intI1的相对丰度(lg值)上升了0.14~1.03。在堆肥过程中,ARGs的相对丰度出现上下波动现象,这可能是由于不同堆肥时期的微生物群落变化的原因[29]。
|
|
表 6 鸡场粪便中ARGs在堆肥处理中的去除效果(lg值) Table 6 Removal effect of ARGs in chicken feces during composting(lgs) |
综上所述,3种养殖场处理工艺对畜禽粪便中ARGs的去除效果有较大差异,但总体对ARGs表现出良好的去除效果,均可以有效去除四环素类ARGs和大环内酯类ermB,但无法有效去除磺胺类ARGs和大环内酯类ermC。值得注意的是,猪场粪便在经过异位发酵床处理后,10种ARGs的相对丰度(lg值)平均下降了1.11;牛场粪便在堆肥后,9种ARGs的相对丰度(lg值)平均下降了0.83,在灭菌作垫料后,5种ARGs的相对丰度(lg值)平均下降了1.12;鸡场粪便堆肥后,8种ARGs的相对丰度(lg值)平均下降了1.32。
3 结论(1)畜禽养殖场粪便和污水中抗生素抗性基因普遍存在(10-7~10-1),但不同畜禽养殖场粪便中ARGs的污染程度存在差异,猪场污水中ARGs的污染水平普遍高于牛场。
(2)经过不同污水处理单元后,猪场和牛场对四环素类ARGs的去除效果相对较好,且相对于牛场,猪场污水处理工艺对ARGs有更强的去除效果;厌氧发酵单元、贮存池单元和调节池单元可能对ARGs去除起到较大作用。
(3)3种养殖场的粪便处理工艺对四环素类ARGs的去除效果较好,对磺胺类ARGs的去除效果较差。
| [1] |
ZHANG Q Q, YING G G, PAN C G, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modeling, and linkage to bacterial resistance[J]. Environmental Science & Technology, 2015, 49(11): 6772-6782. |
| [2] |
邓雯文, 杨盛智, 何雪萍, 等. 牛粪发酵过程中抗生素耐药基因及相关菌群组成变化规律[J]. 生态毒理学报, 2019, 14(2): 153-163. DENG W W, YANG S Z, HE X P, et al. Change of antibiotic resistance genes and bacterial communities during dairy manure composting process[J]. Asian Journal of Ecotoxicology, 2019, 14(2): 153-163. |
| [3] |
ZHU Y G, JOHNSON T A, SU J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3435-3440. DOI:10.1073/pnas.1222743110 |
| [4] |
FORSBERG K J, REYES A, WANG B, et al. The shared antibiotic resistome of soil bacteria and human pathogens[J]. Science, 2012, 337(6098): 1107-1111. DOI:10.1126/science.1220761 |
| [5] |
闫书海. 畜禽养殖废水/粪便中典型抗药基因的调查研究[D]. 杭州: 浙江大学, 2013. YAN S H. Research on representative antibiotic resistance genes in livestock lagoons and manures[D]. Hangzhou: Zhejiang University, 2013. |
| [6] |
彭晶, 高英志, 谷月, 等. 鸡粪有机肥对土壤中抗生素抗性基因和整合酶基因的影响[J]. 环境工程学报, 2019, 13(4): 984-991. PENG J, GAO Y Z, GU Y, et al. Effects of chicken manure organic fertilizer on antibiotic resistance genes and integrase genes in soil[J]. Chinese Journal of Environmental Engineering, 2019, 13(4): 984-991. |
| [7] |
MCKINNEY C W, LOFTIN K A, MEYER M T, et al. tet and sul antibiotic resistance genes in livestock lagoons of various operation type, configuration, and antibiotic occurrence[J]. Environmental Science & Technology, 2010, 44(16): 6102-6109. |
| [8] |
YUAN Q B, ZHAI Y F, MAO B Y, et al. Antibiotic resistance genes and, IntI1 prevalence in a swine wastewater treatment plant and correlation with metal resistance, bacterial community and wastewater parameters[J]. Ecotoxicology and Environmental Safety, 2018, 161: 251-259. DOI:10.1016/j.ecoenv.2018.05.049 |
| [9] |
ZHANG X X, ZHANG T. Occurrence, abundance, and diversity of tetracycline resistance genes in 15 sewage treatment plants across China and other global locations[J]. Environmental Science & Technology, 2011, 45(7): 2598-2604. |
| [10] |
AMINOV R, GARRIGUES-JEANJEAN N, MACKIE R. Molecular ecology of tetracycline resistance: Development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins[J]. Applied and Environmental Microbiology, 2001, 67(1): 22-32. DOI:10.1128/AEM.67.1.22-32.2001 |
| [11] |
MU Q, LI J, SUN Y, et al. Occurrence of sulfonamide-, tetracycline-, plasmid-mediated quinolone-and macrolide-resistance genes in livestock feedlots in northern, China[J]. Environmental Science and Pollution Researsh, 2015, 22(9): 6932-6940. DOI:10.1007/s11356-014-3905-5 |
| [12] |
AMINOV R, CHEE-SANFORD J, GARRIGUES N, et al. Development, validation, and application of PCR primers for detection of tetracycline efflux genes of gram-negative bacteria[J]. Applied and Environmental Microbiology, 2002, 68(4): 1786-1793. DOI:10.1128/AEM.68.4.1786-1793.2002 |
| [13] |
PRUDEN A, PEI R, STORTEBOOM H, et al. Antibiotic resistance genes as emerging contaminants: Studies in northern Colorado[J]. Environmental Science & Technology, 2006, 40(23): 7445-7450. |
| [14] |
CATTOIR V, POIREL L, ROTIMI V, et al. Multiplex PCR for detection of plasmid-mediated quinolone resistance qnr genes in ESBLproducing enterobacterial isolates[J]. Journal of Antimicrobial Chemotherapy, 2007, 60(2): 394-397. DOI:10.1093/jac/dkm204 |
| [15] |
KIM H B, WANG M, PARK C H, et al. oqxAB encoding a multidrug efflux pump in human clinical isolates of Enterobacteriaceae[J]. Antimicrobial Agents and Chemotherapy, 2009, 53(8): 3582-3584. DOI:10.1128/AAC.01574-08 |
| [16] |
WALSH F, INGENFELD A, ZAMPICOLLI M, et al. Real-time PCR methods for quantitative monitoring of streptomycin and tetracycline resistance genes in agricultural ecosystems[J]. Journal of Microbiological Methods, 2011, 86(2): 150-155. DOI:10.1016/j.mimet.2011.04.011 |
| [17] |
FRANK T, GAUTIER V, TALARMIN A, et al. Characterization of sulphonamide resistance genes and class 1 integron gene cassettes in Enterobacteriaceae, Central African Republic(CAR)[J]. The Journal of Antimicrobial Chemotherapy, 2007, 59(4): 724-725. |
| [18] |
阮蓉, 张克强, 杜连柱, 等. 天津市家庭养殖环境中抗生素污染特征与风险评估[J]. 农业环境科学学报, 2021, 40(1): 202-210. RUAN R, ZHANG K Q, DU L Z, et al. Pollution characteristics and risk assessment of antibiotics in a family farm breeding environment in Tianjin[J]. Journal of Agro-Environment Science, 2021, 40(1): 202-210. |
| [19] |
邹威, 金彩霞, 魏闪, 等. 华北地区不同规模畜禽养殖场粪便中抗生素抗性基因污染特征[J]. 农业环境科学学报, 2020, 39(11): 2640-2652. ZOU W, JIN C X, WEI S, et al. Occurrence of antibiotic resistance genes in livestock farms of different scales in north China[J]. Journal of Agro-Environment Science, 2020, 39(11): 2640-2652. DOI:10.11654/jaes.2020-0403 |
| [20] |
ZHANG Y, ZHANG C, PARKER D B, et al. Occurrence of antimicrobials and antimicrobial resistance genes in beef cattle storage pondsandswine treatment lagoons[J]. Science of the Total Environment, 2013, 463: 631-638. |
| [21] |
CHEN B, HAO L J, GUO X Y, et al. Prevalence of antibiotic resistance genes of wastewater and surface water in livestock farms of Jiangsu Province, China[J]. Environmental Science and Pollution Research, 2015, 22(18): 13950-13959. DOI:10.1007/s11356-015-4636-y |
| [22] |
GUO A, GU J, WANG X J, et al. Effects of superabsorbent polymers on the abundances of antibiotic resistance genes, mobile genetic elements, and the bacterial community during swine manure composting[J]. Bioresource Technology, 2017, 244: 658-663. DOI:10.1016/j.biortech.2017.08.016 |
| [23] |
TAO C W, HSU B M, JI W T, et al. Evaluation of five antibiotic resistance genes in wastewater treatment systems of swine farms by realtime PCR[J]. Science of the Total Environment, 2014, 496: 116-121. DOI:10.1016/j.scitotenv.2014.07.024 |
| [24] |
ZHANG R, GU J, WANG X, et al. Relations hips between sulfachloropyridazine sodium, zinc, and sulfonamide resistance genes during the anaerobic digestion of swine manure[J]. Bioresource Technology, 2017, 225: 343-348. DOI:10.1016/j.biortech.2016.10.057 |
| [25] |
DIEHL D L, LAPARA T M. Effect of temperature on the fate of genes encoding tetracycline resistance and the integrase of class 1 integrons within anaerobic and aerobic digesters treating municipal wastewater solids[J]. Environmental Science & Technology, 2010, 44(23): 9128-9133. |
| [26] |
彭晶, 王科, 谷月, 等. 牛粪堆肥系统环境因子对抗性基因的影响[J]. 环境科学, 2019, 40(3): 1439-1445. PENG J, WANG K, GU Y, et al. Effects of environmental factors on tetracycline and macrolides resistance genes in cattle manure composting systems[J]. Environmental Science, 2019, 40(3): 1439-1445. |
| [27] |
LI H, DUAN M L, GU J, et al. Effects of bamboo charcoal on antibiotic resistance genes during chicken manure composting[J]. Ecotoxicology and Environmental Safety, 2017, 140: 1-6. DOI:10.1016/j.ecoenv.2017.01.007 |
| [28] |
SELVAM A, XU D L, ZHAO Z Y, et al. Fate of tetracycline, sulfonamide and fluoroquinolone resistance genes and the changes in bacterial diversity during composting of swine manure[J]. Bioresource Technology, 2012, 126: 383-390. DOI:10.1016/j.biortech.2012.03.045 |
| [29] |
SONG C, LI M, JIA X, et al. Comparison of bacterial community structure and dynamics during the thermophilic composting of different types of solid wastes: Anaerobic digestion residue, pig manure and chicken manure[J]. Microbial Biotechnology, 2014, 7(5): 424-433. DOI:10.1111/1751-7915.12131 |
 2021, Vol. 40
2021, Vol. 40